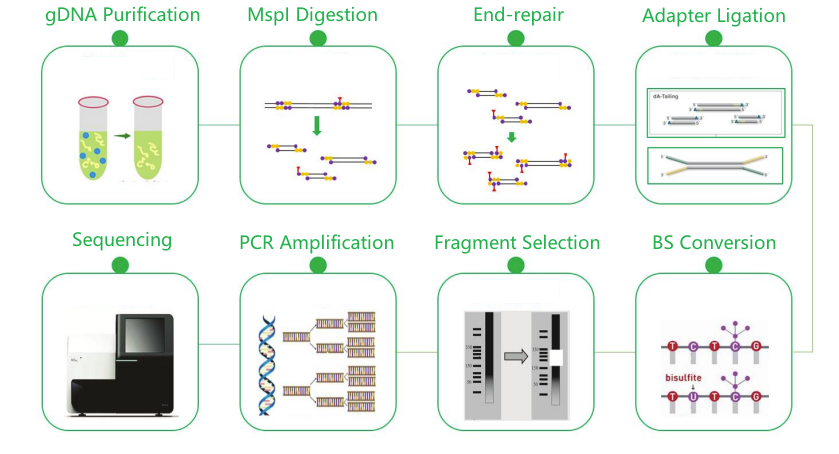
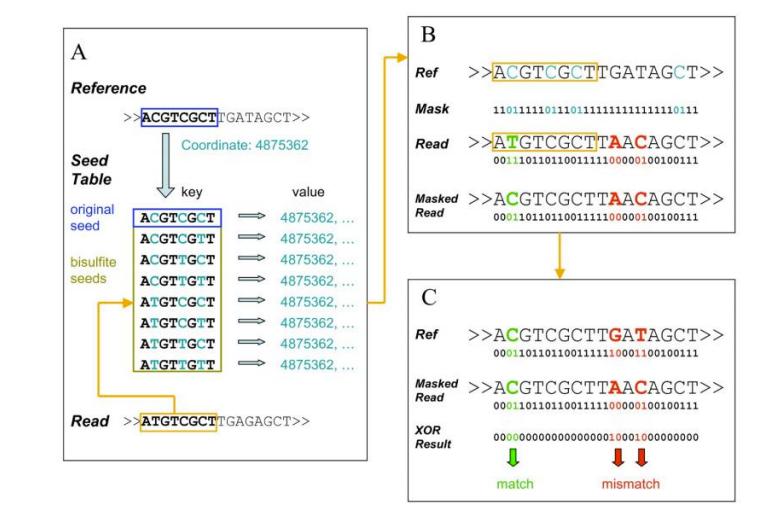
Csökkent reprezentációs biszulfit szekvenálás (RRBS)
Szolgáltatási szolgáltatások
● Szükség van referencia -genomra.
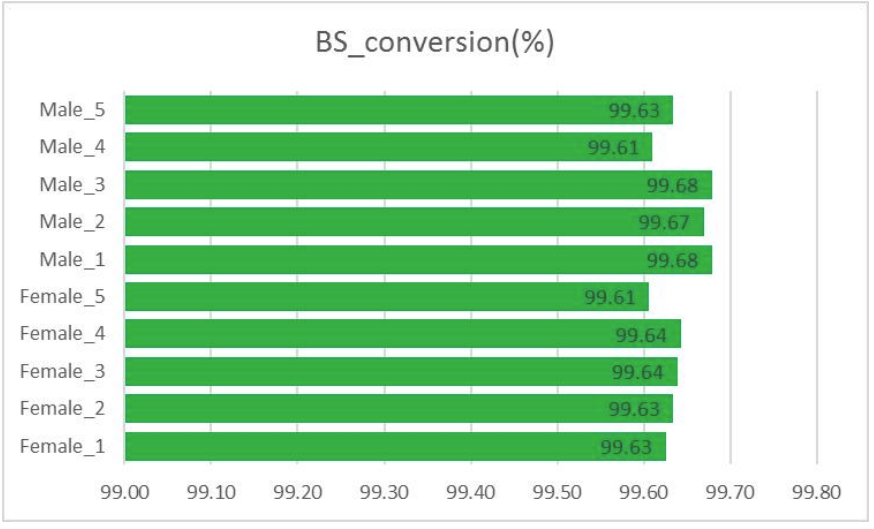
● A lambda DNS -t használják a biszulfit konverziós hatékonyságának monitorozására.
● Az MSPI emésztés hatékonyságát szintén megfigyeljük.
● Kettős enzim emésztés a növényi mintákhoz.
● Szekvenálás az Illumina Novaseq -en.
Szolgáltatási előnyök
●Költséghatékony és hatékony alternatíva a WGB-khez: Az elemzés lehetővé tétele, amelyet alacsonyabb költséggel és alacsonyabb minta követelményekkel kell elvégezni.
●Teljes platform:Biztosítson egyablakos szolgáltatást a mintafeldolgozásból, a könyvtári felépítésből és a szekvenálásból a bioinformatikai elemzéshez.
●Kiterjedt szakértelem: Az RRBS szekvenálási projektekkel, amelyek sikeresen befejeződtek a fajok sokféleségében, a Bmkgene több mint egy évtizedes tapasztalat, magasan képzett elemzőcsoport, átfogó tartalom és kiváló értékesítés utáni támogatás.
Szolgáltatási előírások
| Könyvtár | Szekvenálási stratégia | Ajánlott adatkimenet | Minőség -ellenőrzés |
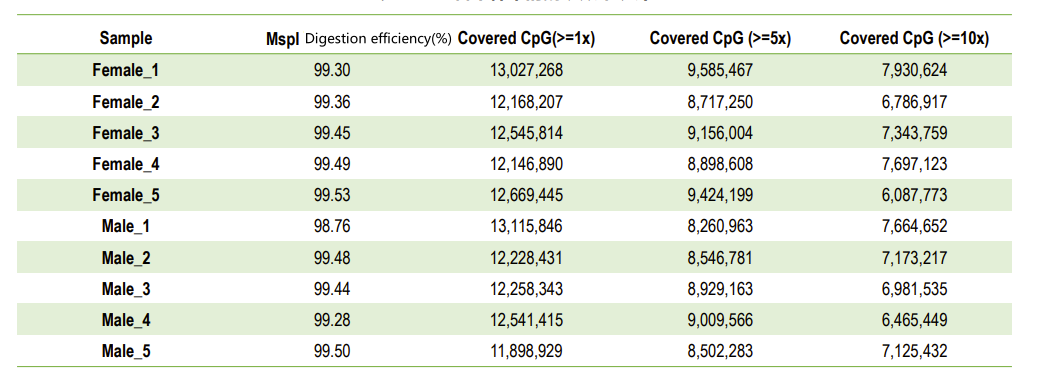
| Az MSPI emésztett és biszulfit kezelt könyvtár | Illumina PE150 | 8 GB | Q30 ≥ 85% Bisulfit konverzió> 99% MSPI vágási hatékonyság> 95% |
Minta követelmények
| Koncentráció (ng/µl) | Teljes összeg (µg) |
| |
| Genomiális DNS | ≥ 30 | ≥ 1 | Korlátozott lebomlás vagy szennyeződés |
Szolgáltatási munka folyamat

Minta szállítás

Könyvtári felépítés

Szekvenálás

Adatelemzés
Értékesítés utáni szolgáltatások
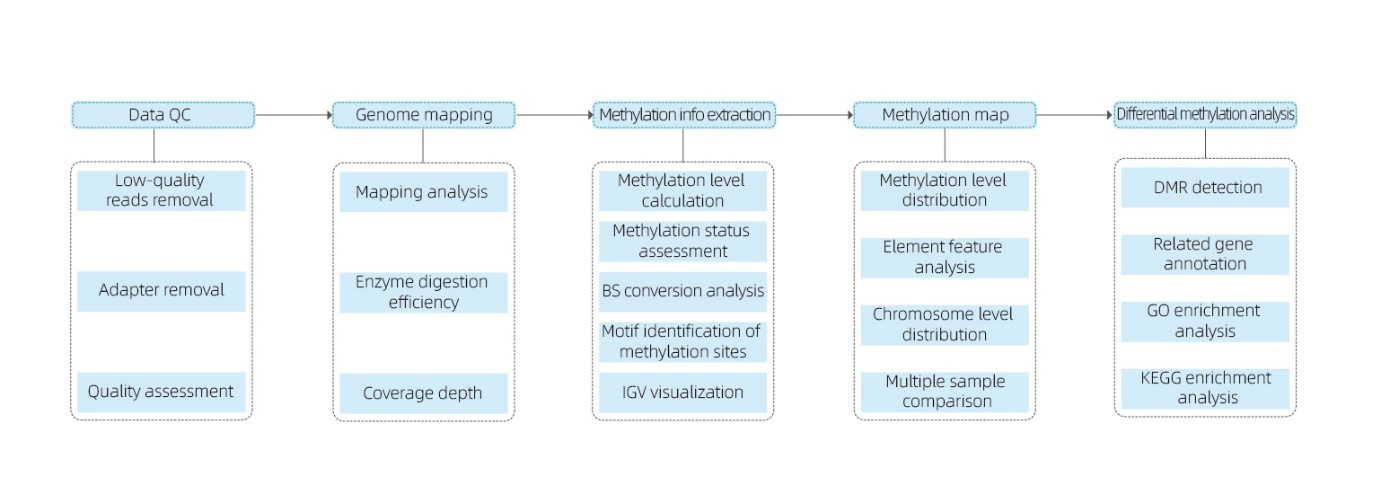
Tartalmazza a következő elemzést:
● nyers szekvenálás minőség -ellenőrzés;
● A referencia -genom feltérképezése;
● 5MC metilezett bázisok kimutatása és a motívumok azonosítása;
● A metilezés eloszlásának és a minta összehasonlításának elemzése;
● a differenciálisan metilezett régiók (DMR) elemzése;
● A DMR -khez kapcsolódó gének funkcionális kommentálása.
Minőségellenőrzés: Az emésztés hatékonysága (a genom feltérképezésében)
Minőségellenőrzés: Bisulfite átalakítás (metilezési információk extrahálásában)
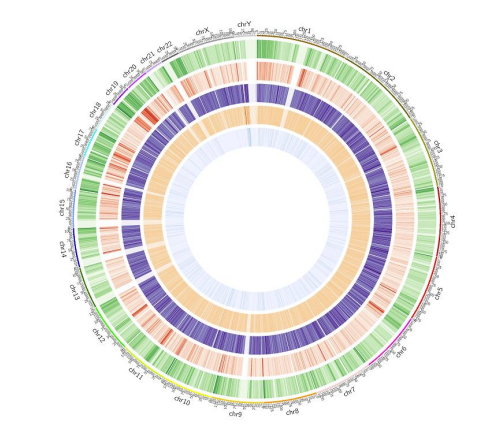
Metilezési térkép: 5mc metilezés genom széles eloszlás
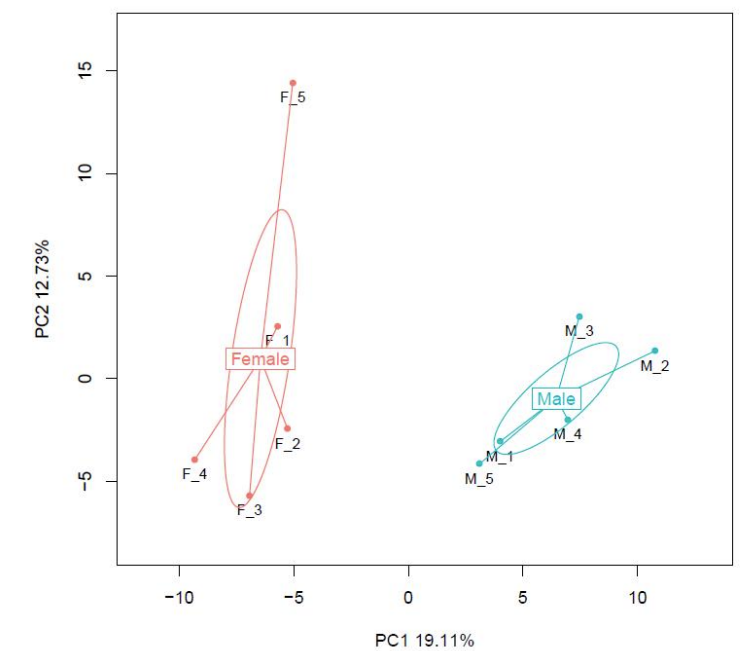
Minta összehasonlítás: Fő komponens -elemzés
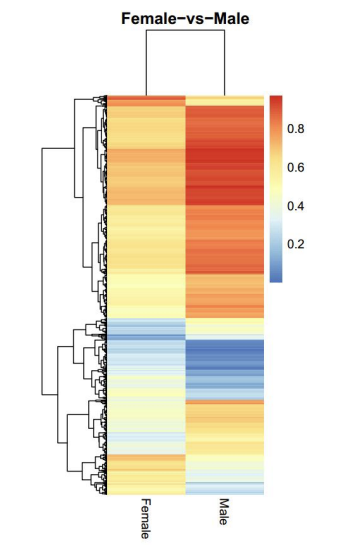
Differenciálisan metilezett régiók (DMRS) elemzés: Heatmap
Fedezze fel a Bmkgene teljes genom biszulfit szekvenálási szolgáltatásainak elősegített kutatási előrelépéseit a publikációk kurátus gyűjteményén keresztül.
Li, Z. et al. (2022) „A nagy hűséges átprogramozás Leydig-szerű sejtekké CRISPR aktiválás és paracrin tényezőkkel”,PNAS Nexus, 1 (4). doi: 10.1093/pnasnexus/pgac179.
Tian, H. et al. (2023) „A test összetételének genomszintű DNS-metilezési elemzése a kínai monozigótikus ikreknél”,Európai Journal of Clinical Investigation, 53 (11), p. E14055. doi: 10.1111/eci.14055.
Wu, Y. et al. (2022) „DNS-metilezés és derék-csípő arány: egy epigenóma széles asszociációs tanulmány a kínai monozigotikus ikreknél”,Endokrinológiai nyomozás folyóirat, 45 (12), 2365–2376. doi: 10.1007/s40618-022-01878-4.