Emberi egész exome szekvenálás
Szolgáltatási jellemzők
● Két exome panel áll rendelkezésre a szondákkal végzett céldúsítás alapján: Sure Select Human All Exon v6 (Agilent) és xGen Exome Hybridization Panel v2 (IDT).
● Szekvenálás az Illumina NovaSeq-en.
● Betegségelemzésre vagy tumorelemzésre irányuló bioinformatikai csővezeték.
Szolgáltatás előnyei
●A fehérjekódoló régiót célozza meg: A fehérjét kódoló régiók rögzítésével és szekvenálásával a hWES-t a fehérje szerkezetével kapcsolatos változatok feltárására használják.
●Költséghatékony:A hWES az emberi betegséggel összefüggő mutációk körülbelül 85%-át adja az emberi genom 1%-ából.
●Nagy pontosságú: A nagy szekvenálási mélységgel a hWES megkönnyíti mind a gyakori, mind a ritka, 1%-nál alacsonyabb frekvenciájú változatok észlelését.
●Szigorú minőségellenőrzés: Öt alapvető kontrollpontot valósítunk meg minden szakaszban, a minta- és könyvtár-előkészítéstől a szekvenálásig és a bioinformatikáig. Ez az aprólékos ellenőrzés biztosítja a folyamatosan jó minőségű eredményeket.
●Átfogó bioinformatikai elemzés: csővezetékünk túlmutat a referenciagenom variációinak azonosításán, mivel olyan fejlett elemzéseket tartalmaz, amelyek kifejezetten a betegségek genetikai vonatkozásaival vagy a daganatanalízissel kapcsolatos kutatási kérdésekre irányulnak.
●Értékesítés utáni támogatás:Elkötelezettségünk a projekt befejezésén túl is 3 hónapos értékesítés utáni szolgáltatási időszakkal terjed ki. Ez idő alatt projektkövetést, hibaelhárítási segítséget és kérdés-feleleteket kínálunk az eredményekkel kapcsolatos kérdések megválaszolásához.
Minta specifikációk
| Exon Capture Stratégia | Szekvenálási stratégia | Ajánlott adatkimenet |
| Biztos Select Human All Exon v6 (Agilent) vagy xGen Exome Hybridization Panel v2 (IDT)
| Illumina NovaSeq PE150 | 5-10 Gb Mendeli rendellenességek/ritka betegségek esetén: > 50x Tumorminták esetén: ≥ 100x |
Mintakövetelmények
| Minta típusa
| Összeg(Qubit®)
| Koncentráció | Kötet
| Tisztaság (NanoDrop™) |
|
Genomi DNS
| ≥ 50 ng | ≥ 6 ng/μL | ≥ 15 μL | OD260/280=1,8-2,0 nincs lebomlás, nincs szennyeződés
|
Bioinformatika

A hWES-betegség mintáinak bioinformatikai elemzése a következőket tartalmazza:
● Sorozati adatok minőségellenőrzése
● Referencia genom-igazítás
● SNP-k és InDel-ek azonosítása
● SNP-k és InDel-ek funkcionális megjegyzései

A tumorminták bioinformatikai elemzése a következőket tartalmazza:
● Sorozati adatok minőségellenőrzése
● Referencia genom-igazítás
● SNP-k, InDel-ek és szomatikus variációk azonosítása
● Csíravonal-változatok azonosítása
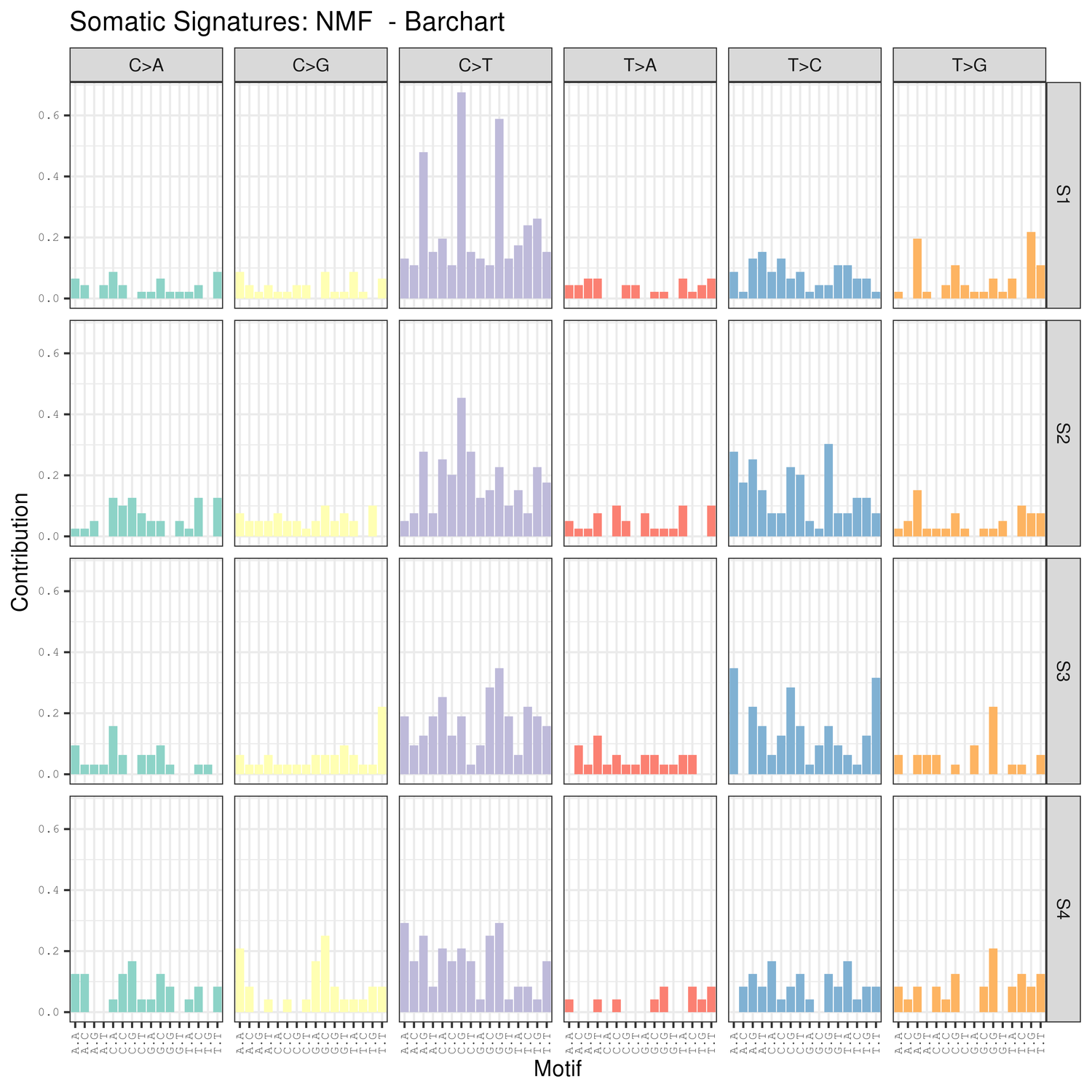
● Mutációs aláírások elemzése
● Meghajtó gének azonosítása funkciónövekedési mutációk alapján
● Mutációs annotáció a gyógyszerérzékenység szintjén
● Heterogenitás analízis – tisztaság és ploiditás számítása
Szerviz munkafolyamat

Mintaszállítás

DNS kivonás

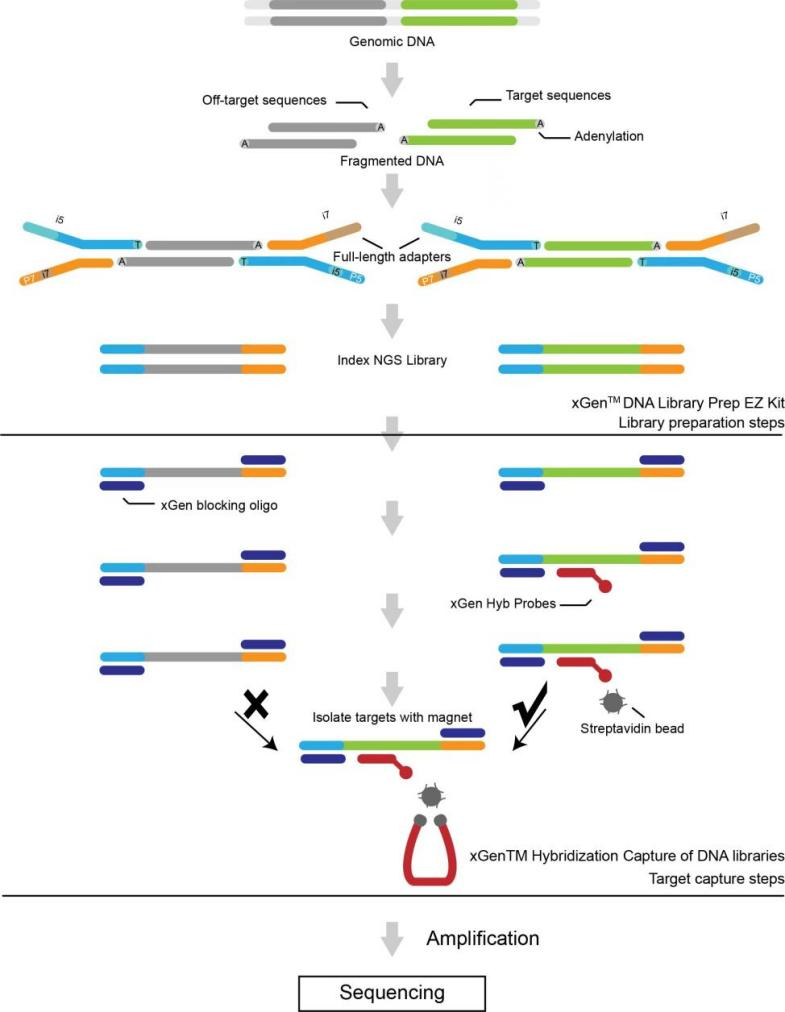
Könyvtárépítés

Sorrendezés

Adatelemzés

Adatszolgáltatás

Értékesítés utáni szolgáltatások
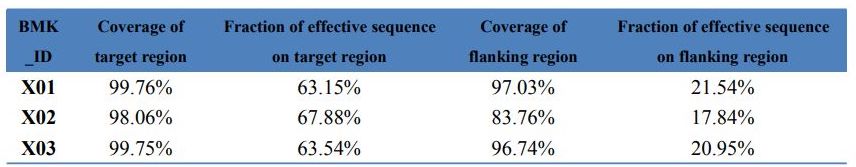
Data QC – Exome-rögzítés statisztikái
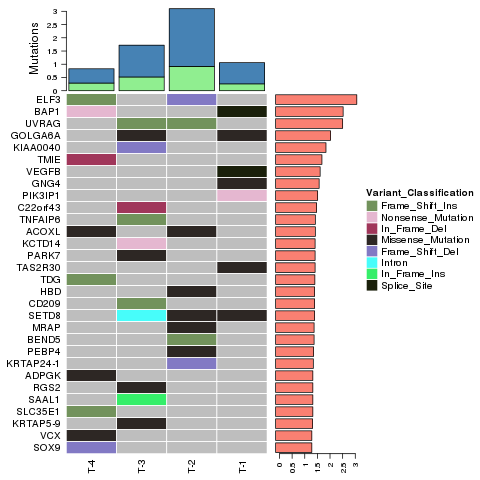
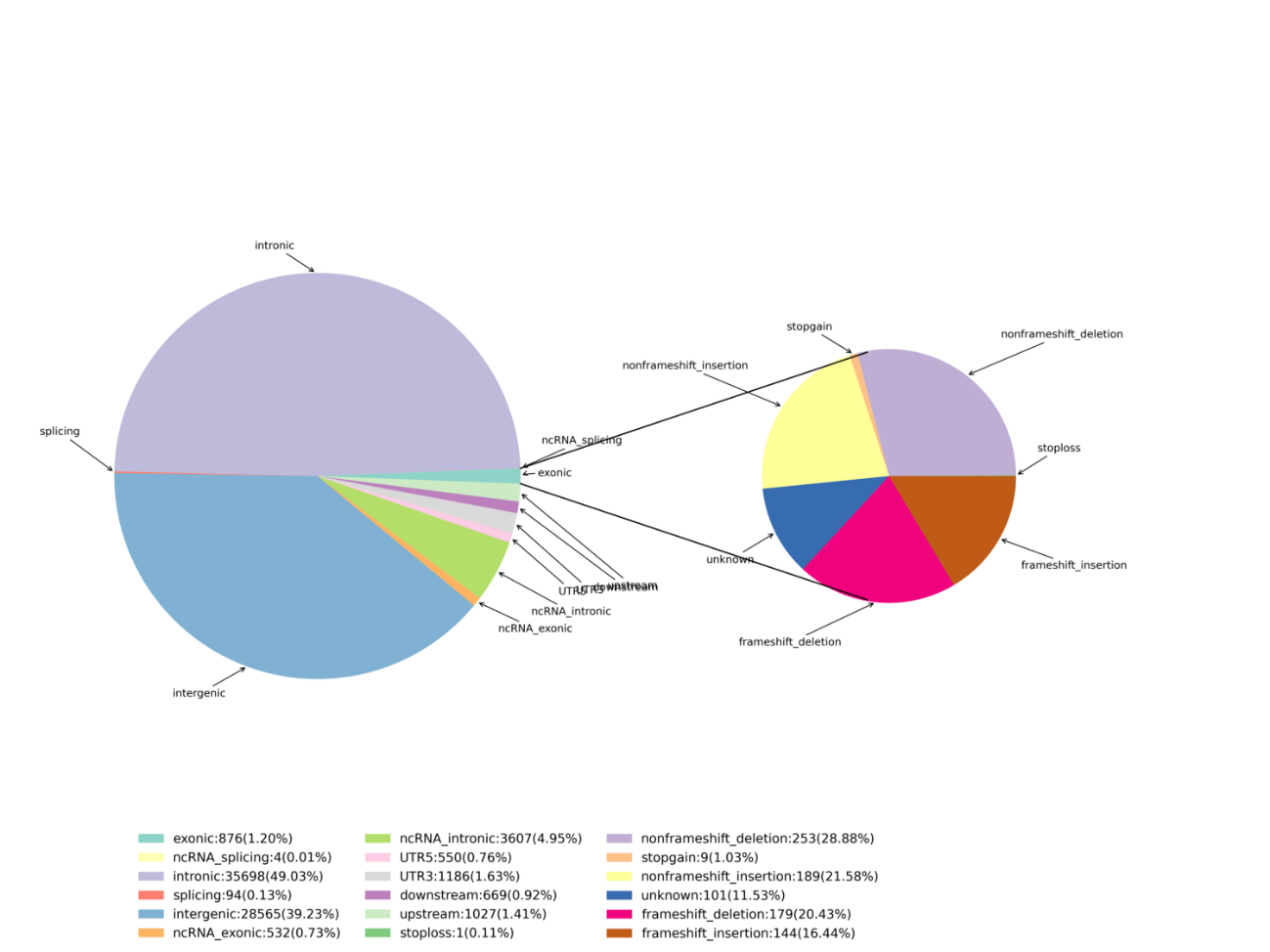
Változat azonosítás – InDels
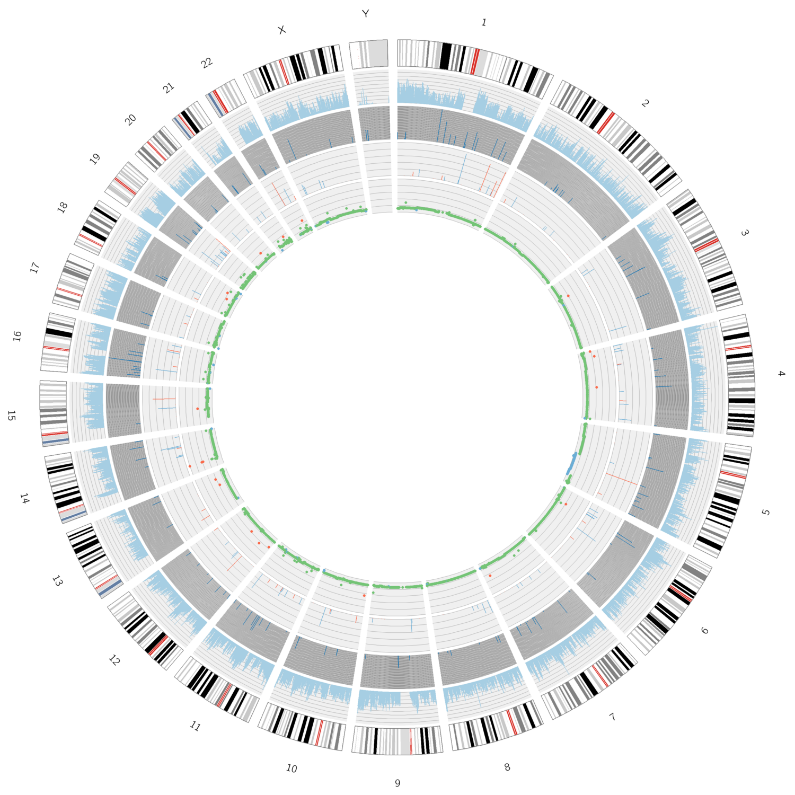
Speciális elemzés: a káros SNP-k/InDelek azonosítása és eloszlása – Circos plot
Tumoranalízis: szomatikus mutációk azonosítása és eloszlása – Circos plot
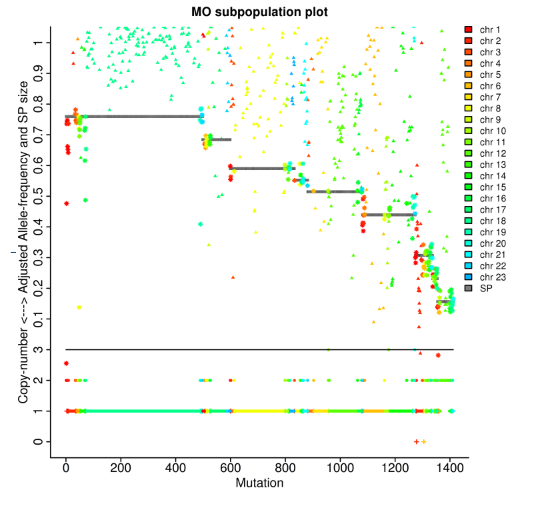
Tumoranalízis: klonális vonalak