Hi-C alapú genom összeállítás
Szolgáltatási jellemzők
● Szekvenálás Illumina NovaSeq-en PE150-el.
● A szolgáltatáshoz az extrahált nukleinsavak helyett a szövetminták formaldehiddel való keresztkötését és a DNS-fehérje kölcsönhatások megőrzését igénylik.
● A Hi-C kísérlet magában foglalja a ragadós végek biotinnal történő korlátozását és véghelyreállítását, majd a kapott tompa végek körkörösítését, miközben megőrzi a kölcsönhatásokat. A DNS-t ezután sztreptavidin gyöngyökkel lehúzzuk, és a következő könyvtár-előállításhoz megtisztítjuk.
Szolgáltatás előnyei

A Hi-C áttekintése
(Lieberman-Aiden E et al.,Tudomány, 2009)
●A genetikai populációs adatok iránti igény megszüntetése:A Hi-C helyettesíti a kontig horgonyzáshoz szükséges alapvető információkat.
●Magas markersűrűség:ami magas, 90% feletti kontig horgonyzási arányt eredményez.
●Széleskörű szakértelem és publikációs nyilvántartás:A BMKGene hatalmas tapasztalattal rendelkezik több mint 2000 Hi-C Genome Assembly esetében 1000 különböző fajból és különböző szabadalmakból. Több mint 200 közzétett eset 2000 feletti kumulatív hatástényezővel rendelkezik.
●Magasan képzett bioinformatikai csapat:A saját fejlesztésű vizualizációs adatszoftver a Hi-C kísérletekhez és adatelemzéshez szükséges belső szabadalmakkal és szoftver szerzői jogokkal rendelkezik, amely lehetővé teszi a blokkok kézi mozgatását, megfordítását, visszavonását és újrakészítését.
●Értékesítés utáni támogatás:Elkötelezettségünk a projekt befejezésén túl is 3 hónapos értékesítés utáni szolgáltatási időszakkal terjed ki. Ez idő alatt projektkövetést, hibaelhárítási segítséget és kérdés-feleleteket kínálunk az eredményekkel kapcsolatos kérdések megválaszolásához.
●Átfogó megjegyzés: több adatbázist használunk, hogy funkcionálisan annotáljuk a gének azonosított változatait, és elvégezzük a megfelelő dúsítási elemzést, betekintést nyújtva több kutatási projektbe.
Szolgáltatási specifikációk
| Könyvtári előkészítés | Szekvenálási stratégia | Javasolt adatkiadás | Minőségellenőrzés |
| Hi-C könyvtár | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85% |
Mintakövetelmények
| Szövet | Szükséges összeg |
| Állati zsigerek | ≥ 2 g |
| Állati izom | |
| Emlősvér | ≥ 2 ml |
| Baromfi/halvér | |
| Növény - friss levél | ≥ 3 g |
| Tenyésztett sejtek | ≥ 1x107 |
| Rovar | ≥ 2 g |
Szerviz munkafolyamat

Kísérleti tervezés

Mintaszállítás

Könyvtárépítés

Sorrendezés

Adatelemzés

Értékesítés utáni szolgáltatások
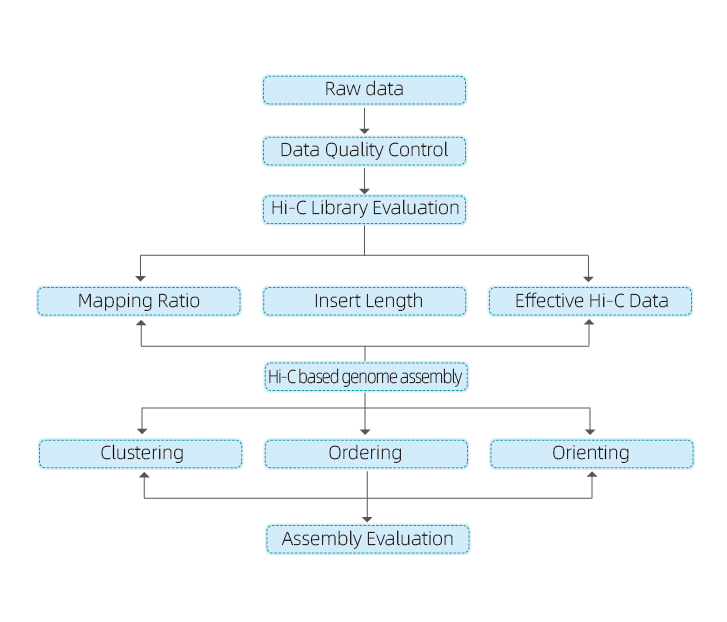
1) Nyers adatok minőségellenőrzése
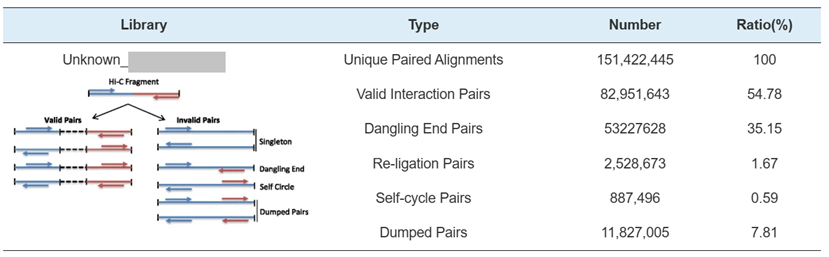
2) Hi-C könyvtár QC: az érvényes Hi-C kölcsönhatások becslése
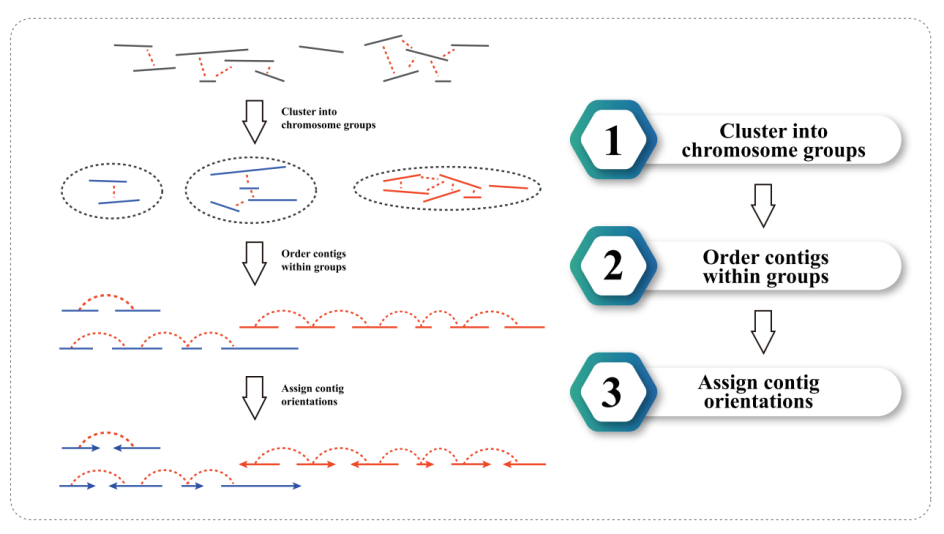
3) Hi-C összeállítás: a kontigek csoportosítása, majd az egyes csoportokon belüli kontig rendezés és a kontig orientáció hozzárendelése
4) Hi-C értékelés
Hi-C Library QC – a Hi-C érvényes interakciós párok becslése
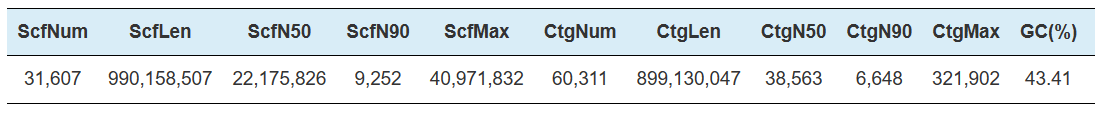
Hi-C Assembly – statisztika
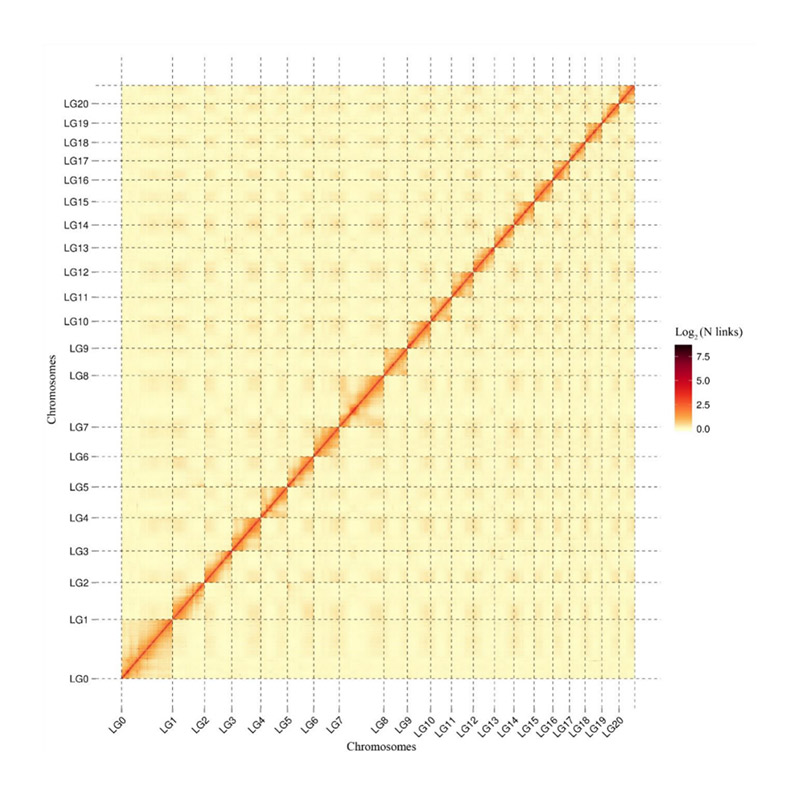
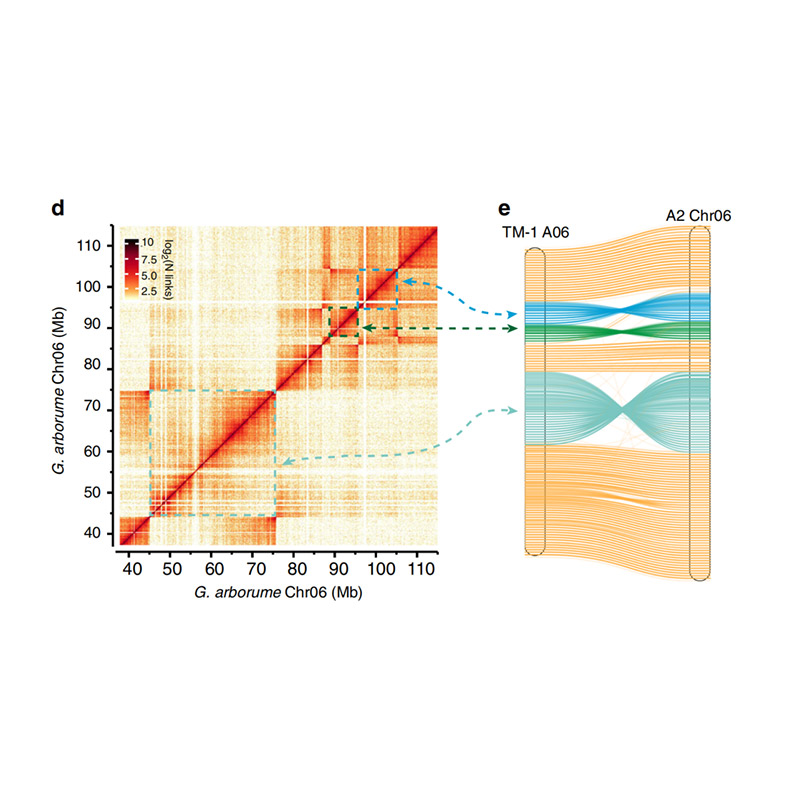
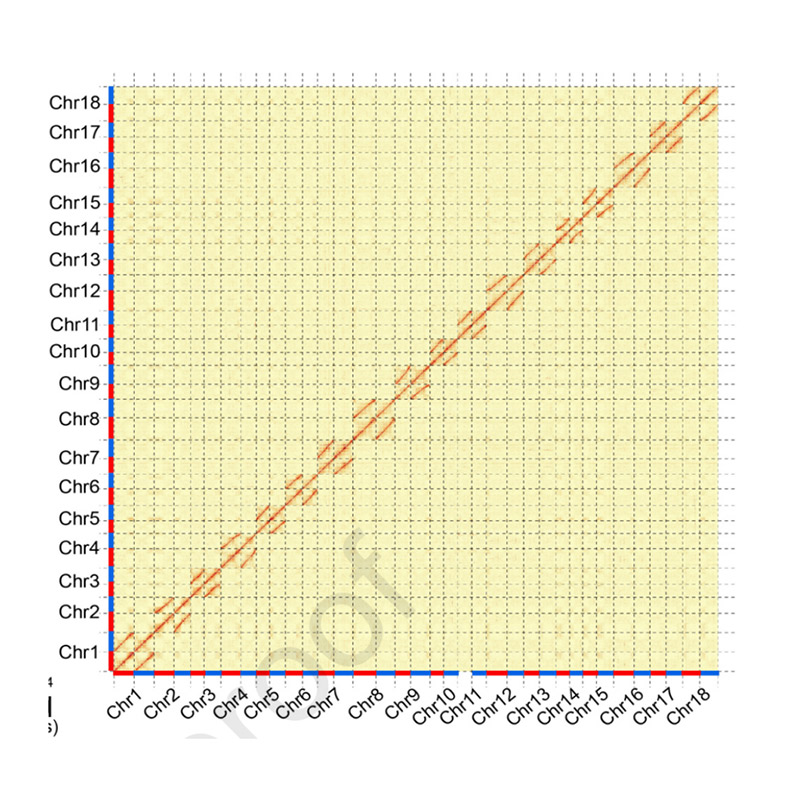
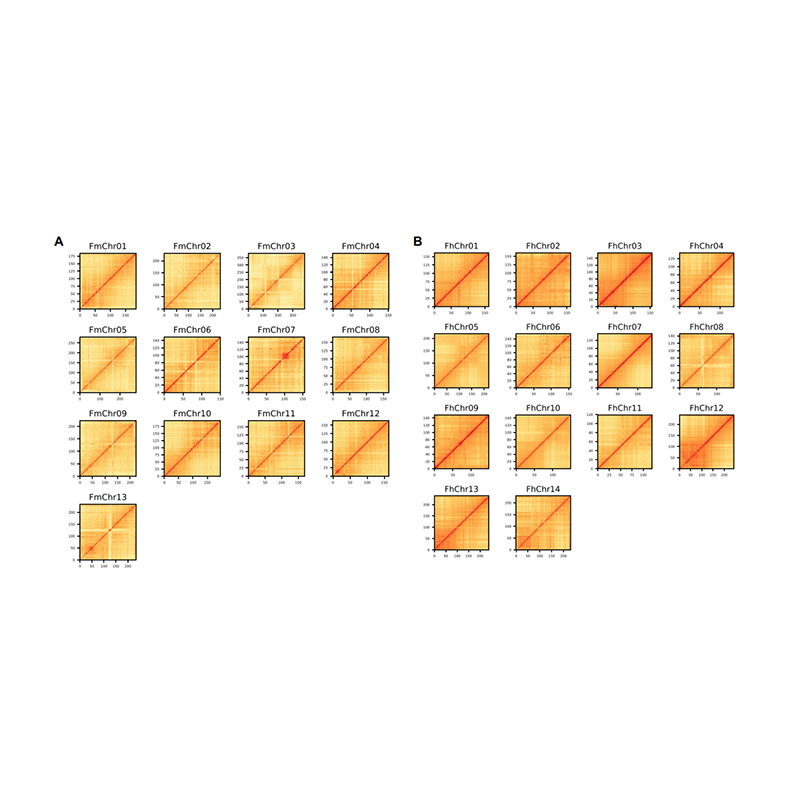
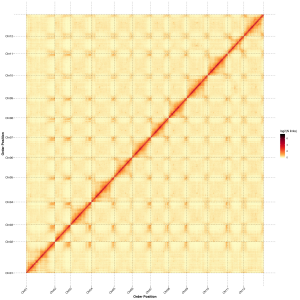
Összeszerelés utáni értékelés – a binek közötti jelintenzitás hőtérképe
Fedezze fel a BMKGene Hi-C összeszerelési szolgáltatásai által elérhető fejlesztéseket egy válogatott kiadványgyűjtemény segítségével.
Tian, T. et al. (2023) „Egy kiemelkedő, szárazságtűrő kukoricacsíra genom-összeállítása és genetikai dissekciója”, Nature Genetics 2023 55:3, 55(3), 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) „Az ázsiai mézelő méh Apis cerana genomjának kromoszóma-skálás összeállítása”, Frontiers in Genetics, 11. o. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) „A tropánalkaloid bioszintézis evolúciójának feltárása a Solanaceae család két genomjának elemzésével”, Nature Communications 2023 14:1, 14(1), 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) „A banyánfa és a beporzó darázs genomjai betekintést nyújtanak a füge-darázs koevolúciójába”, Cell, 183(4), 875-889.e17. doi: 10.1016/J.CELL.2020.09.043