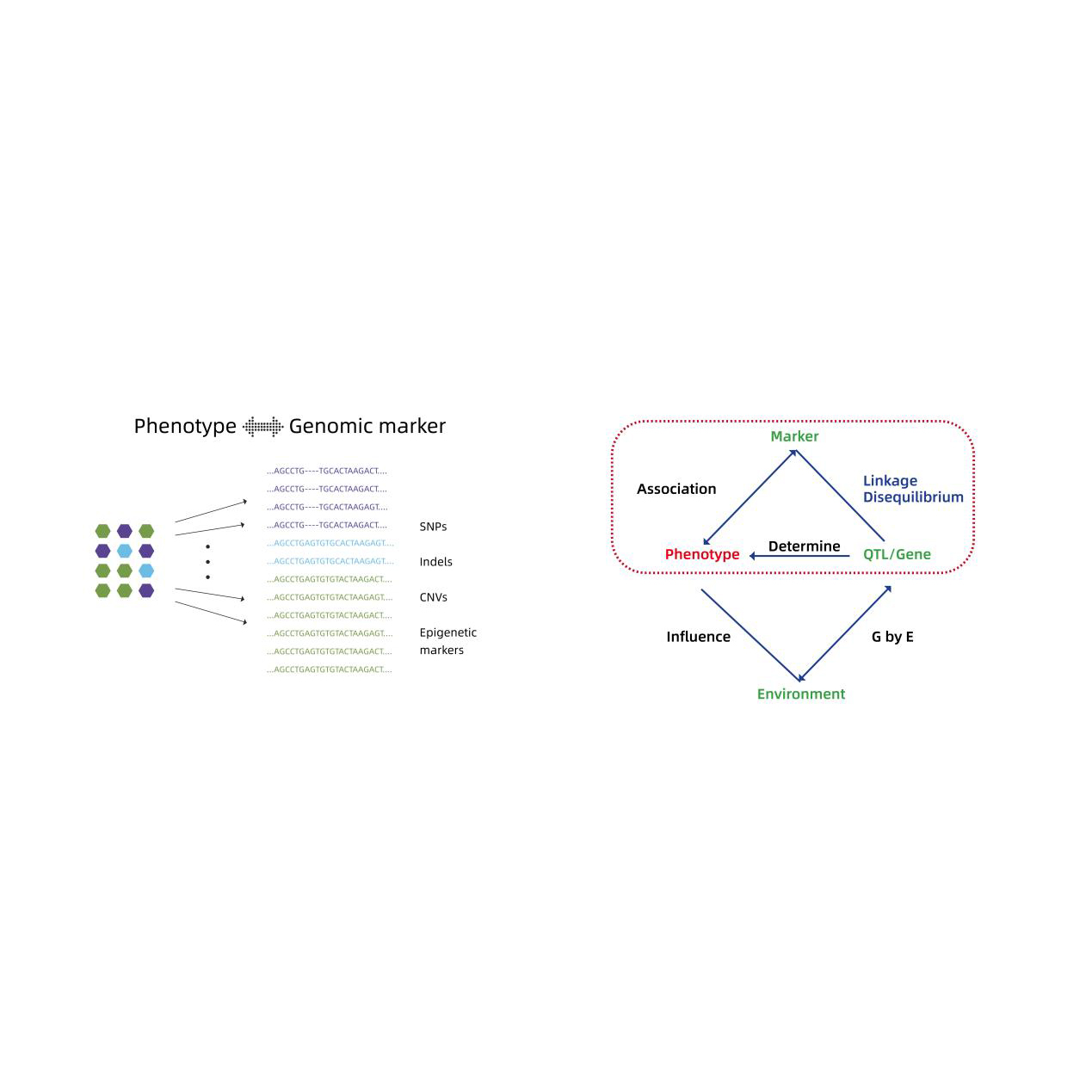
Genom-szerte asszociációs elemzés
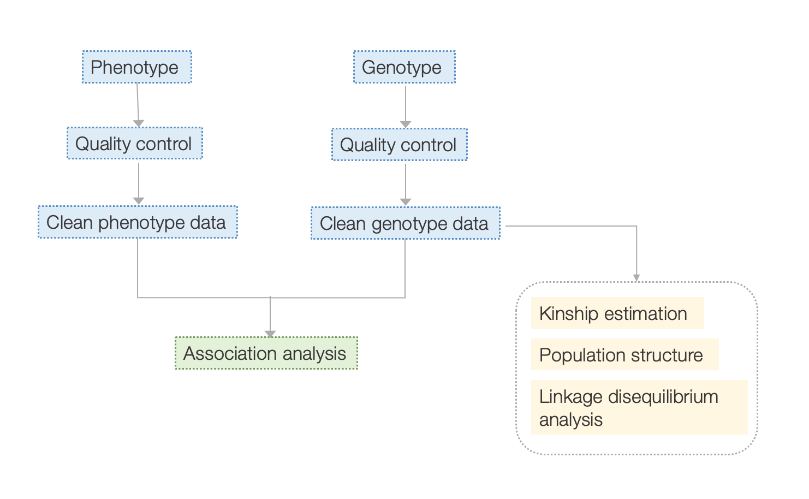
Munkafolyamat

Szolgáltatás előnyei
●Széleskörű szakértelem és publikációs nyilvántartás: a GWAS-ban felhalmozott tapasztalattal a BMKGene több száz fajprojektet hajtott végre a populációs GWAS-kutatásban, segített a kutatóknak több mint 100 cikk publikálásában, és a kumulált impakt faktor elérte az 500-at.
● Átfogó bioinformatikai elemzés: a munkafolyamat magában foglalja az SNP-jellemzők asszociációs elemzését, amely egy sor jelölt gént és a megfelelő funkcionális annotációt szolgáltat.
●Magasan képzett bioinformatikai csapat és rövid elemzési ciklus: a fejlett genomikai elemzés terén nagy tapasztalattal rendelkező BMKGene csapata átfogó elemzéseket készít, gyors átfutási idővel.
●Értékesítés utáni támogatás:Elkötelezettségünk a projekt befejezésén túl is 3 hónapos értékesítés utáni szolgáltatási időszakkal terjed ki. Ez idő alatt projektkövetést, hibaelhárítási segítséget és kérdés-feleleteket kínálunk az eredményekkel kapcsolatos kérdések megválaszolásához.
Szolgáltatási előírások és követelmények
| A szekvenálás típusa | Javasolt populációs skála | Szekvenálási stratégia | Nukleotid követelmények |
| Teljes genom szekvenálás | 200 minta | 10x | Koncentráció: ≥ 1 ng/µL Teljes mennyiség ≥ 30 ng Korlátozott vagy nincs bomlás vagy szennyeződés |
| Specific-Locus Amplified Fragment (SLAF) | Címkemélység: 10x Címkék száma: < 400 Mb: WGS ajánlott < 1 Gb: 100 000 címke 1 Gb > 2 Gb: 300 000 címke Max 500 ezer címke | Koncentráció ≥ 5 ng/µL Teljes mennyiség ≥ 80 ng Nanodrop OD260/280=1,6-2,5 Agaróz gél: nincs bomlás vagy szennyeződés, vagy csak korlátozott mértékben
|
Anyag kiválasztása



Különböző fajták, alfajok, tájfajták/génbankok/vegyes családok/vad erőforrások
Különböző fajták, alfajok, tájfajták
Féltestvér család/teljes testvércsalád/vad erőforrások
Szerviz munkafolyamat

Kísérleti tervezés

Mintaszállítás

RNS extrakció

Könyvtárépítés

Sorrendezés

Adatelemzés

Értékesítés utáni szolgáltatások
A következő elemzést tartalmazza:
- Genom-szerte asszociációs elemzés: LM, LMM, EMMAX, FASTLMM modell
- A jelölt gének funkcionális annotációja
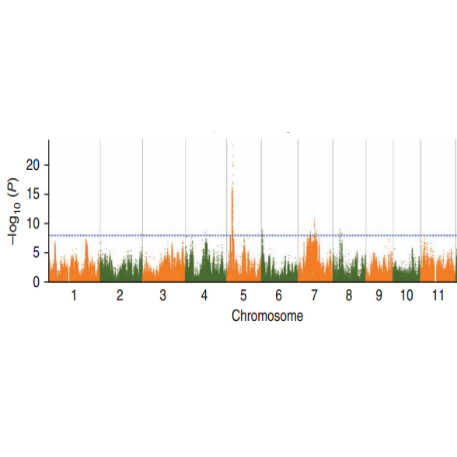
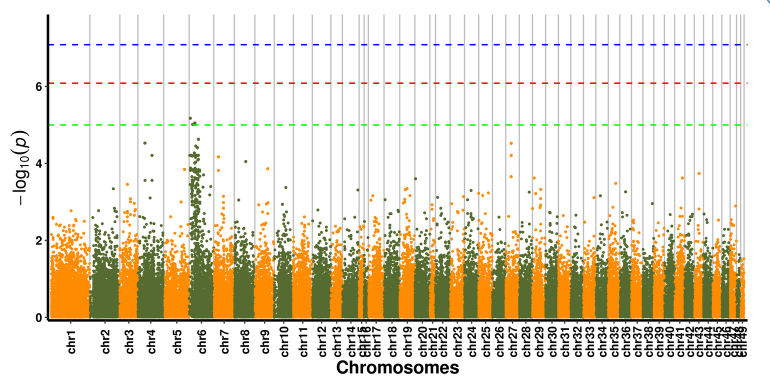
SNP-vonás asszociációs elemzés – Manhattan plot
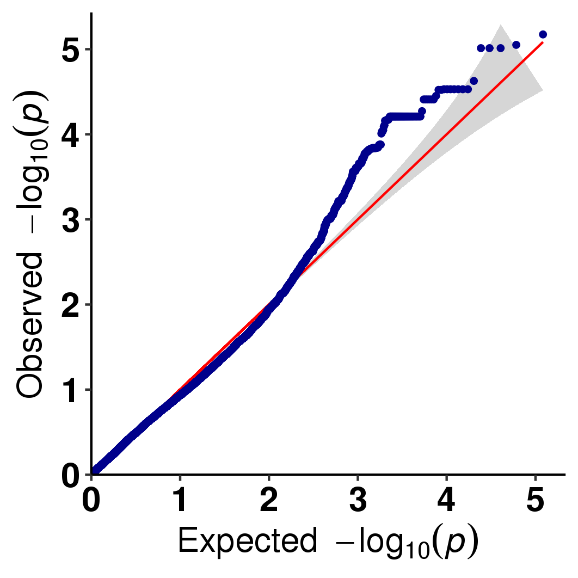
SNP-vonás asszociációs elemzés – QQ diagram
Fedezze fel a BMKGene de GWAS szolgáltatásai által elősegített fejlesztéseket egy válogatott kiadványgyűjtemény segítségével:
Lv, L. et al. (2023) „Betekintés a Sinonovacula constricta borotvakagyló ammóniatoleranciájának genetikai alapjába genomszintű asszociációs vizsgálattal”,Akvakultúra, 569, p. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et al. (2022) „398 rókafarkköles-származék multi-omikai elemzése a háziasítással, metabolitjellemzőkkel és gyulladáscsökkentő hatásokkal kapcsolatos genomiális régiókat tár fel”Molekuláris növény, 15(8), 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) „Genome-Wide Association Mapping of Hulles Barely Phenotypes in Drought Environment”,A növénytudomány határai, 13. o. 924892. doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) „A szulfotranszferázt kódoló GmST1 rezisztenciát biztosít a szójabab mozaikvírus G2 és G3 törzseivel szemben”,Növény, sejt és környezet, 44(8), 2777–2792. doi: 10.1111/PCE.14066.