Meghatalmazott szegregáns elemzés
Szolgáltatási előnyök

Takagi et al., The Plant Journal, 2013
● Pontos lokalizáció: A botok keverése 30+30-200+200 egyénnel a háttérzaj minimalizálása érdekében; Nem szinonim mutatánsok alapú jelölt régió előrejelzése.
● Átfogó elemzés: Mélyreható jelölt génfunkció-kommentár, beleértve az NR, a Swissprot, a GO, a KEGG, a COG, a KOG stb.
● Gyorsabb fordulási idő: A gyors gén lokalizációja 45 munkanapon belül.
● KOCKÁZATOS TAPASZTALOM: A BMK több ezer tulajdonsággal járult hozzá, különféle fajok, például növények, vízi termékek, erdők, virágok, gyümölcsök stb.
Szolgáltatási előírások
Lakosság:
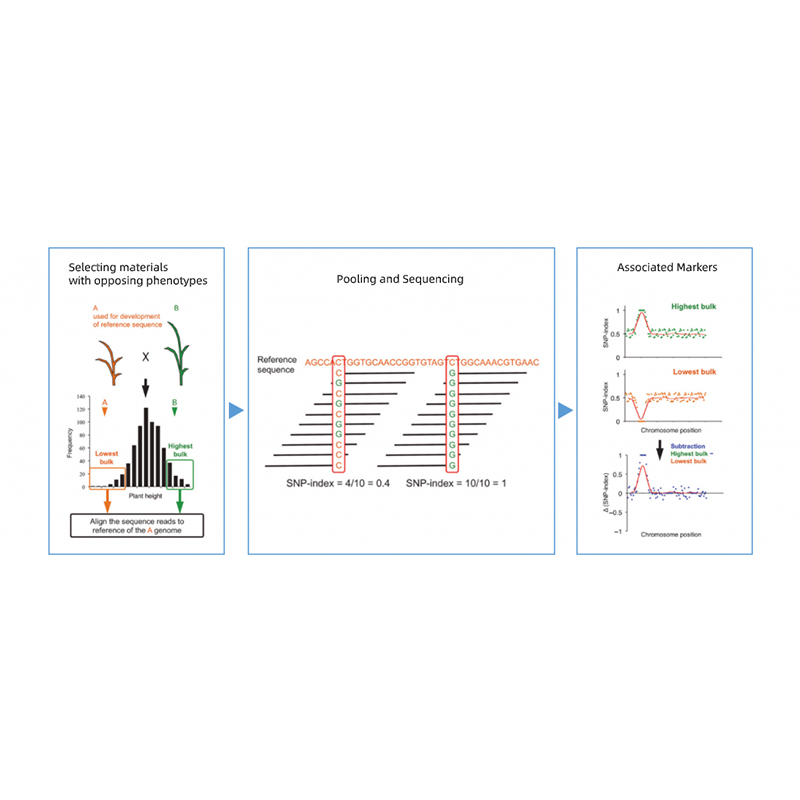
A szülőknek az ellenkező fenotípusokkal rendelkező utódok elkülönítése.
pl. F2 utódok, háttérkép (BC), rekombináns beltenyésztett vonal (RIL)
Keverőkészlet
Minőségi tulajdonságokhoz: 30-50 egyed (minimum 20)/ömlesztett
A kvantitatív Tratis esetében: a legnépszerűbb 5–10% -os egyéneknek, akiknek a teljes populációban vannak szélsőséges fenotípusai (legalább 30+30).
Ajánlott szekvenálási mélység
Legalább 20x/szülő és 1x/utódok egyéni (pl. 30+30 egyéni utódkeverési készlet esetén a szekvenálási mélység ömlesztettenként 30x lesz)
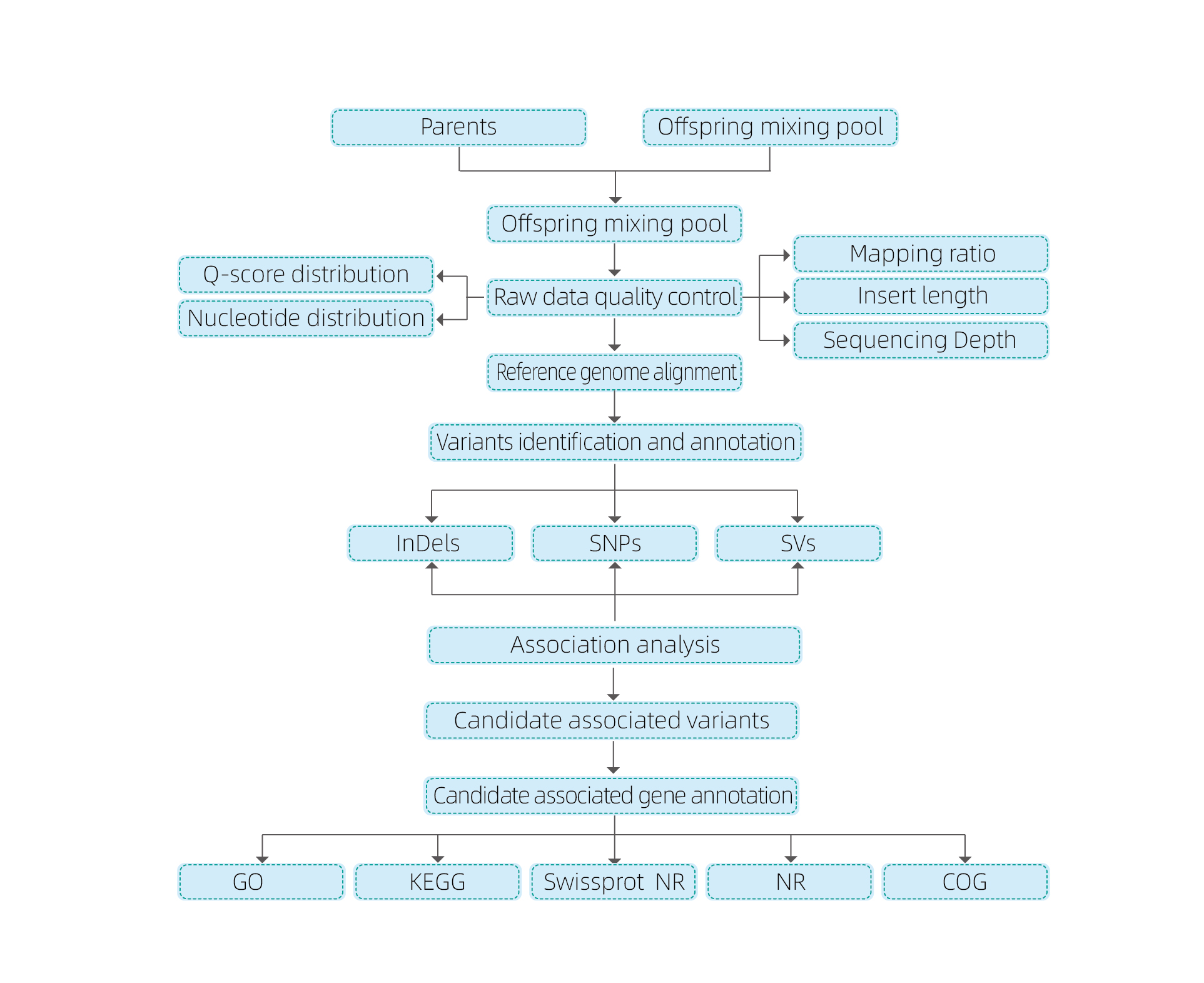
Bioinformatikai elemzések
● Az egész genom újrafestés
● Adatfeldolgozás
● SNP/indel hívás
● A jelölt régió szűrése
● A jelölt génfunkció -kommentár

Minta követelmények és szállítási követelmények
Minta követelmények:
Nukleotidok:
| gDNS -minta | Szöveti minta |
| Koncentráció: ≥30 ng/μL | Növények: 1-2 g |
| Mennyiség: ≥2 μg (röplab ≥15 μl) | Állatok: 0,5-1 g |
| Tisztítás: OD260/280 = 1,6-2,5 | Teljes vér: 1,5 ml |
Szolgáltatási munka folyamat

Kísérleti tervezés

Minta szállítás

RNS -extrakció

Könyvtári felépítés

Szekvenálás

Adatelemzés

Értékesítés utáni szolgáltatások
( A következő ábra
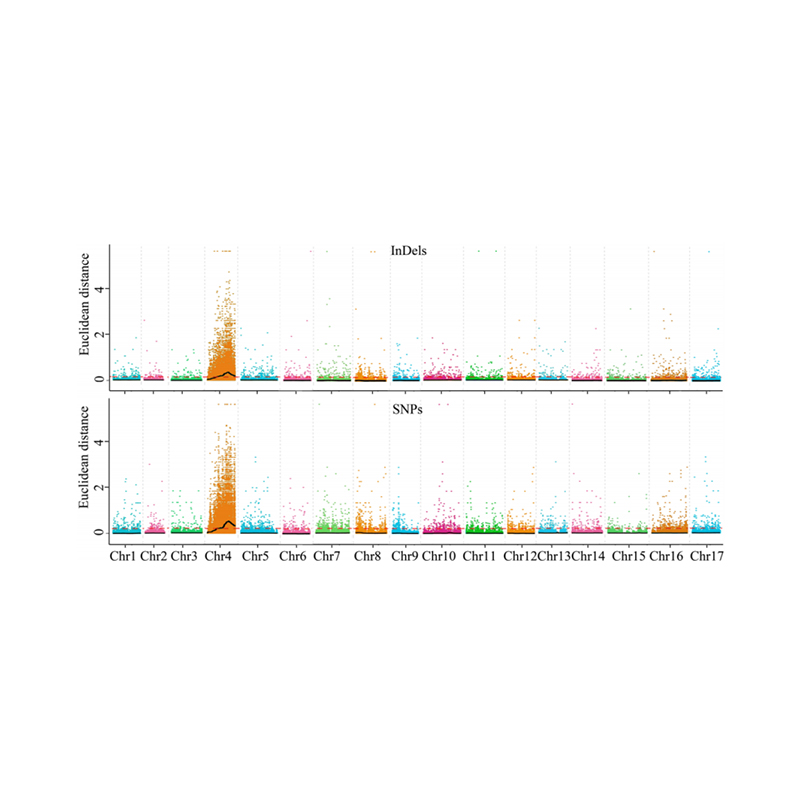
X tengely: kromoszóma száma; Minden pont az SNP ED értékét képviseli. A fekete vonal megfelel a felszerelt ED értéknek. A magasabb ED érték azt jelzi, hogy a hely és a fenotípus között jelentősebb összefüggést mutat. A Red Dash Line a jelentős asszociáció küszöbét képviseli.

2. Association analízis nem SNP-index alapú
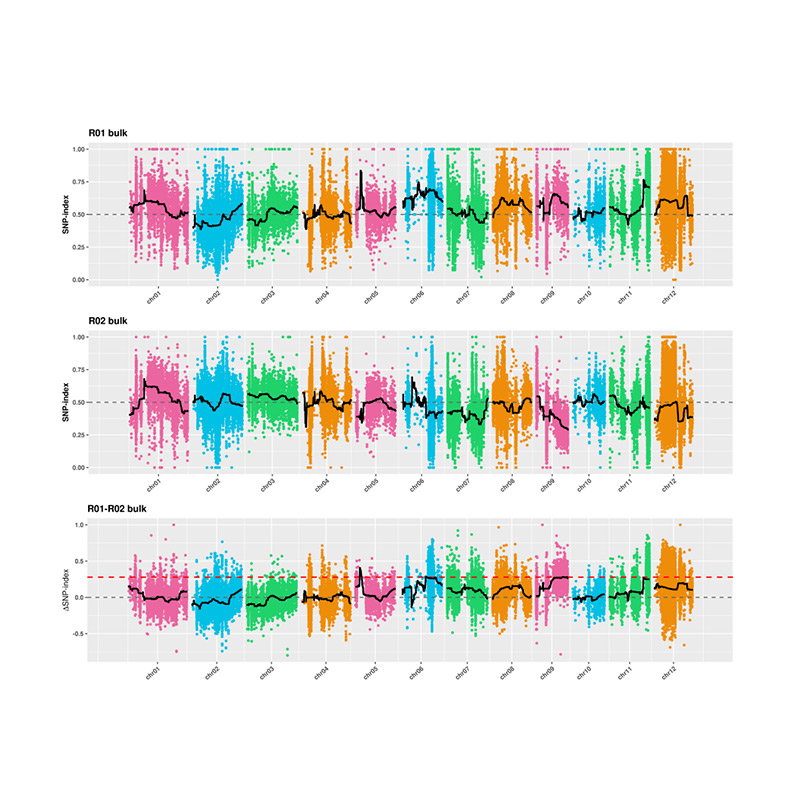
X tengely: kromoszóma száma; Minden pont az SNP-index értéket képviseli. A fekete vonal a felszerelt SNP-index értéket jelenti. Minél nagyobb az érték, annál jelentősebb az egyesület.

BMK -eset
Az FNL7.1, a Locus Locus legfontosabb kvantitatív tulajdonsága egy késői embriogenezist kódol, amely az uborka gyümölcsnyak hosszához társítva.
Közzétett: Növényi biotechnológiai folyóirat, 2020
Szekvenálási stratégia:
Szülők (JIN5-508, YN): A teljes genom újraelvezetése 34 × és 20 ×.
DNS-medencék (50 hosszú nyakú és 50 rövid nyakú): 61 × és 52 ×
Kulcsfontosságú eredmények
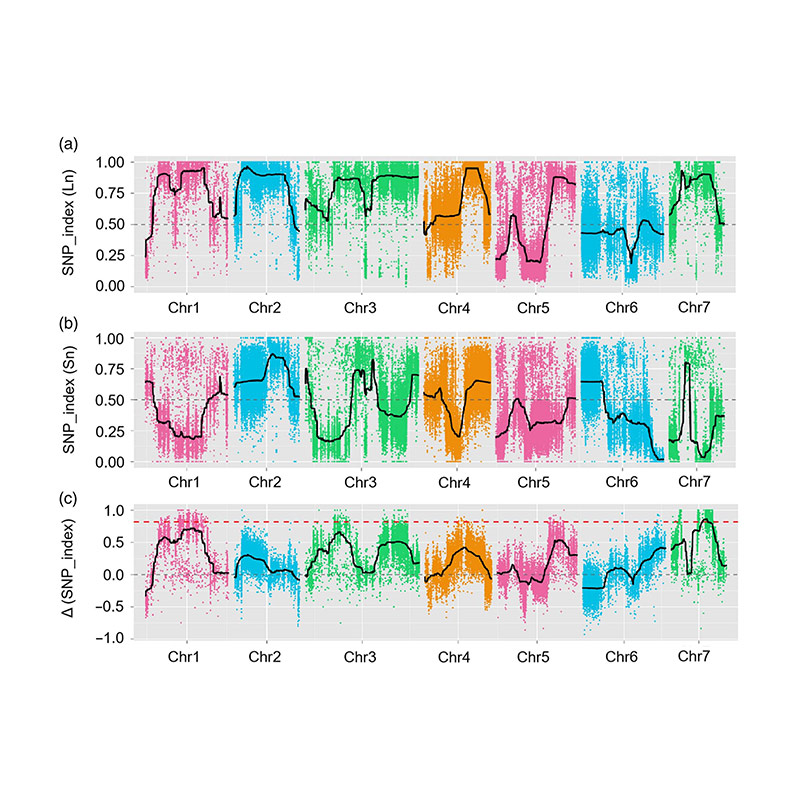
Ebben a tanulmányban a szegregáló populációt (F2 és F2: 3) a JIN5-508 hosszú nyakú uborka és a rövid nyakú YN átlépésével állítottuk elő. Két DNS-medencét 50 szélsőséges hosszú nyakú egyén és 50 extrém rövid nyakú személy épített. A főhatást a QTL-t a ChR07-en BSA elemzéssel és a hagyományos QTL leképezéssel azonosították. A jelölt régiót tovább szűkítették a finom leképezés, a génexpressziós mennyiségi meghatározás és a transzgenikus kísérletek, amelyek feltárták a kulcsfontosságú gént a nyakhosszúság, a CSFNL7.1 szabályozásában. Ezenkívül azt találták, hogy a polimorfizmus a CSFNL7.1 promoter régióban társul a megfelelő expresszióhoz. A további filogenetikai elemzés azt sugallta, hogy az FNL7.1 lókusz valószínűleg Indiából származik.
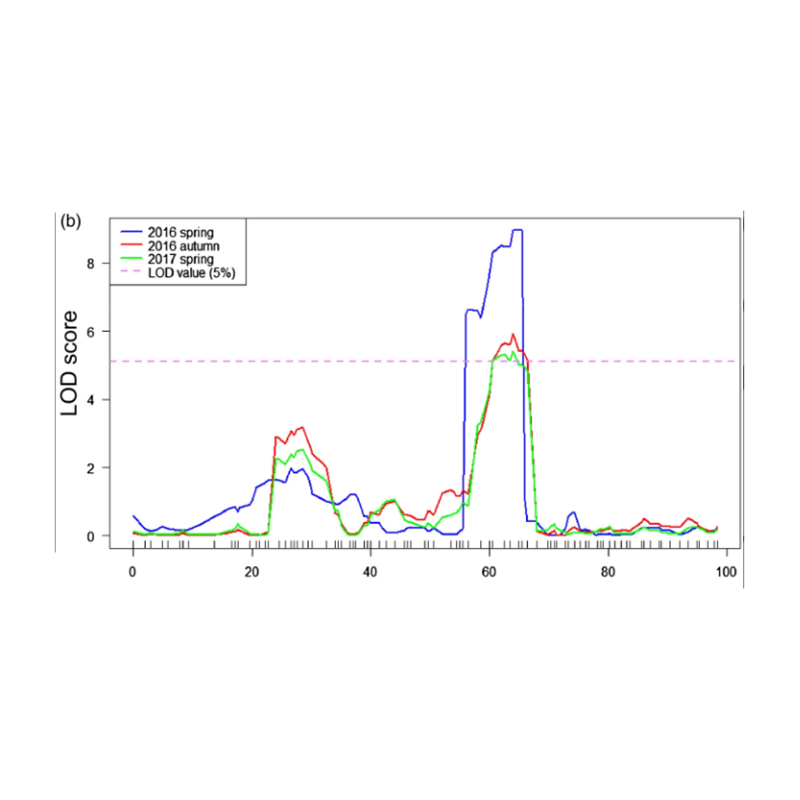
 QTL-térképezés a BSA elemzésben az uborka nyakhosszához kapcsolódó jelölt régió azonosítására |  Az uborka nyakhosszú QTL LOD profiljai a Chr07-en azonosítottak |
Xu, X., et al. "Az FNL7.1, a Locus Locus legfontosabb kvantitatív tulajdonsága egy késői embriogenezist kódol, amely az uborka gyümölcsnyak hosszához társul." Plant Biotechnology Journal 18.7 (2020).