Tout sekans transkriptòm - Illumina
Karakteristik
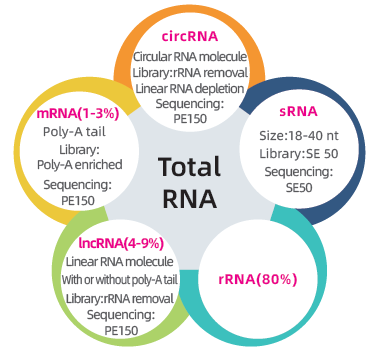
● Doub Bibliyotèk nan sekans Transcriptome a konplè: rediksyon rRNA ki te swiv pa PE150 Preparasyon Bibliyotèk ak Seleksyon Size ki te swiv pa SE50 Preparasyon Bibliyotèk
● Ranpli analiz bioinformatics nan mRNA, lncRNA, circRNA, ak miRNA nan separe rapò bioinformatics
● Joint analiz de tout ekspresyon RNA nan yon rapò konbine, ki gen ladan CERNA rezo analiz.
Avantaj Sèvis
●Analiz an pwofondè nan rezo regilasyon: se analiz rezo CERNA pèmèt pa sekans nan jwenti nan mRNA, lncRNA, circRNA, ak miRNA ak pa yon workflow konplè bioinformatic.
●Complète anotasyon: Nou itilize baz done miltip pou anote fonksyonèl jèn yo diferansye eksprime (DEG) ak fè analiz la anrichisman korespondan, bay Sur nan pwosesis yo selilè ak molekilè kache repons lan transkriptòm.
●Anpil ekspètiz: Avèk yon dosye track nan fèmen avèk siksè sou 2100 pwojè transkriptòm antye nan domèn rechèch divès kalite, ekip nou an pote yon richès nan eksperyans nan chak pwojè.
●Kontwòl kalite solid: Nou aplike pwen kontwòl debaz atravè tout etap, ki soti nan echantiyon ak preparasyon bibliyotèk nan sekans ak bioinformatics. Sa a siveyans metikuleu asire livrezon an nan toujou-wo kalite rezilta yo.
●Post-lavant sipò: Angajman nou an pi lwen pase fini pwojè ak yon peryòd sèvis 3-mwa apre-sale. Pandan tan sa a, nou ofri pwojè swiv-up, depanaj asistans, ak Q & Yon sesyon yo adrese nenpòt kesyon ki gen rapò ak rezilta yo
Egzanp kondisyon ak livrezon
| Bibliotèk | Estrateji sekans | Done rekòmande | Kontwòl kalite |
| rRNA apovri | Illumina PE150 | 16 GB | Q30≥85% |
| Gwosè chwazi | Illumina SE50 | 10-20m li |
Egzijans Egzanp:
Nukleotid:
| Conc. (Ng/μl) | Kantite lajan (μg) | Pite | Entegrite |
| ≥ 80 | ≥ 1.6 | OD260/280 = 1.7-2.5 OD260/230 = 0.5-2.5 Limite oswa pa gen pwoteyin oswa ADN kontaminasyon yo montre sou jèl. | Rin≥6.0 5.0≥28s/18s≥1.0; Limite oswa pa gen okenn elevasyon debaz |
Rekòmande livrezon echantiyon
Veso: 2 ml tib santrifijez (papye fèblan pa rekòmande)
Etikèt Etikèt: Gwoup+replike EG A1, A2, A3; B1, B2, B3.
Chajman:
1. sèk-glas: echantiyon yo dwe chaje nan sache ak antere l 'nan sèk-glas.
2. Tib rnastable: echantiyon RNA ka seche nan tib estabilizasyon RNA (egzanp RNASTABLE®) ak anbake nan tanperati chanm.
Sèvis koule travay

Eksperyans Design

Livrezon echantiyon

Ekstraksyon RNA

Konstriksyon Bibliyotèk

Syen

Done analiz

Apre-sale sèvis yo
Bioinformatics
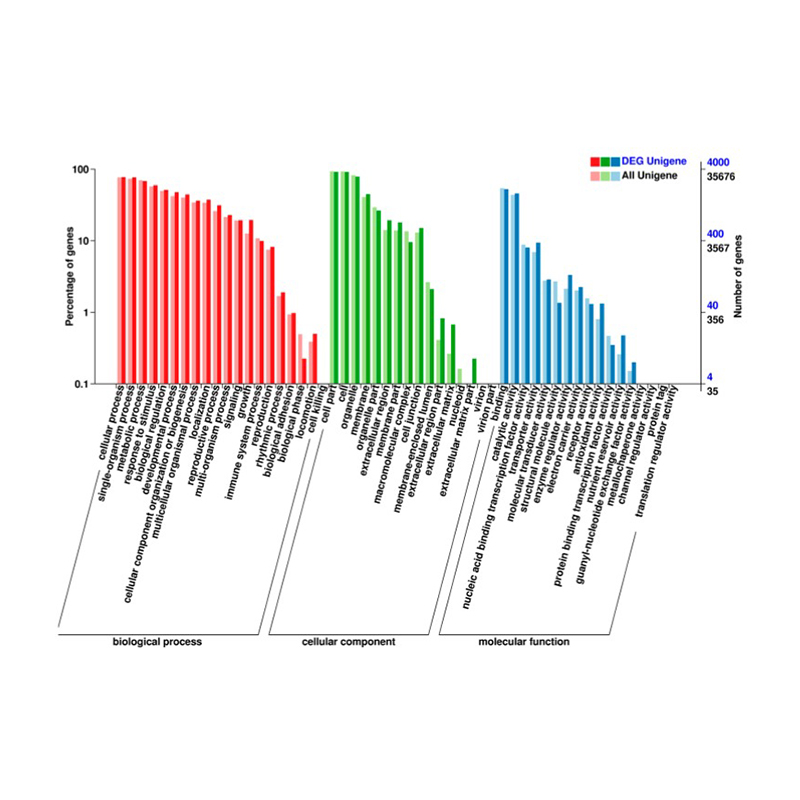
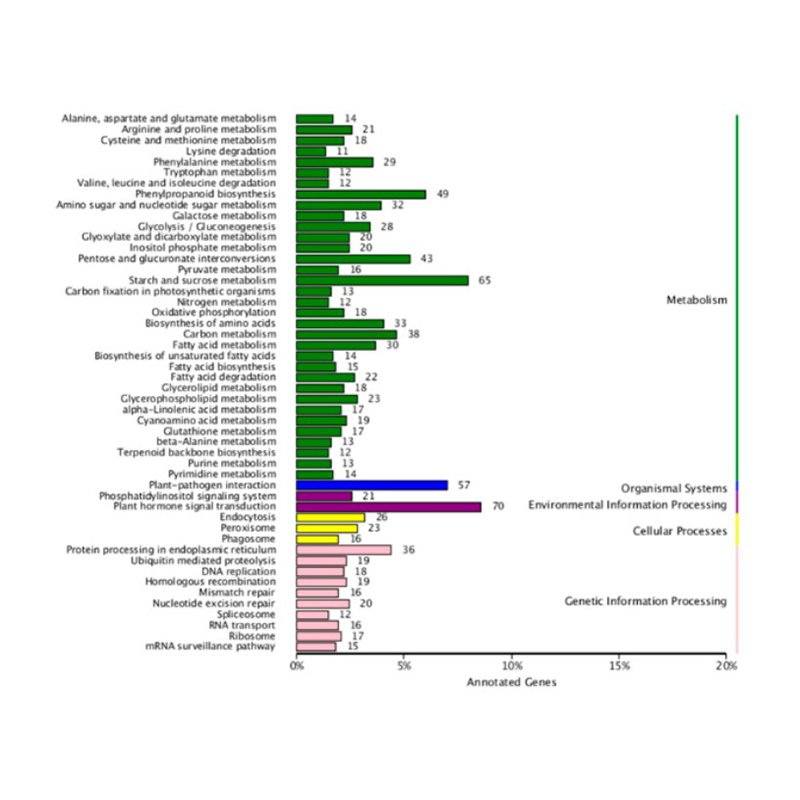
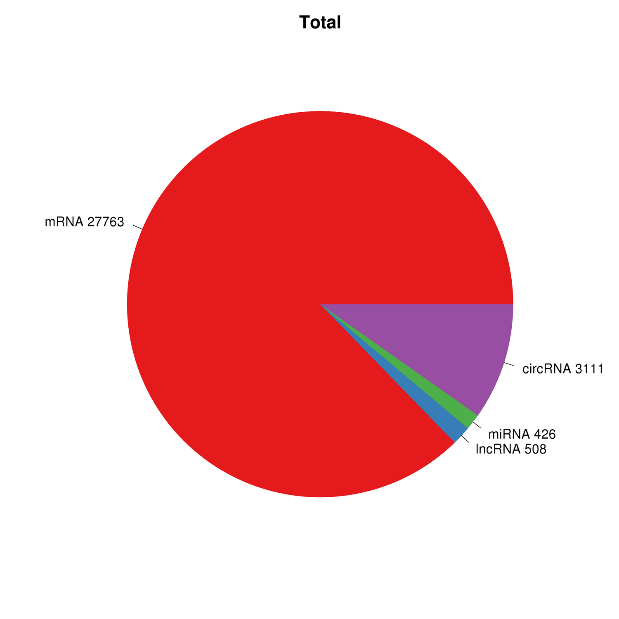
Ekspresyon RNA Apèsi sou lekòl la
Diferansyèlman eksprime jèn yo
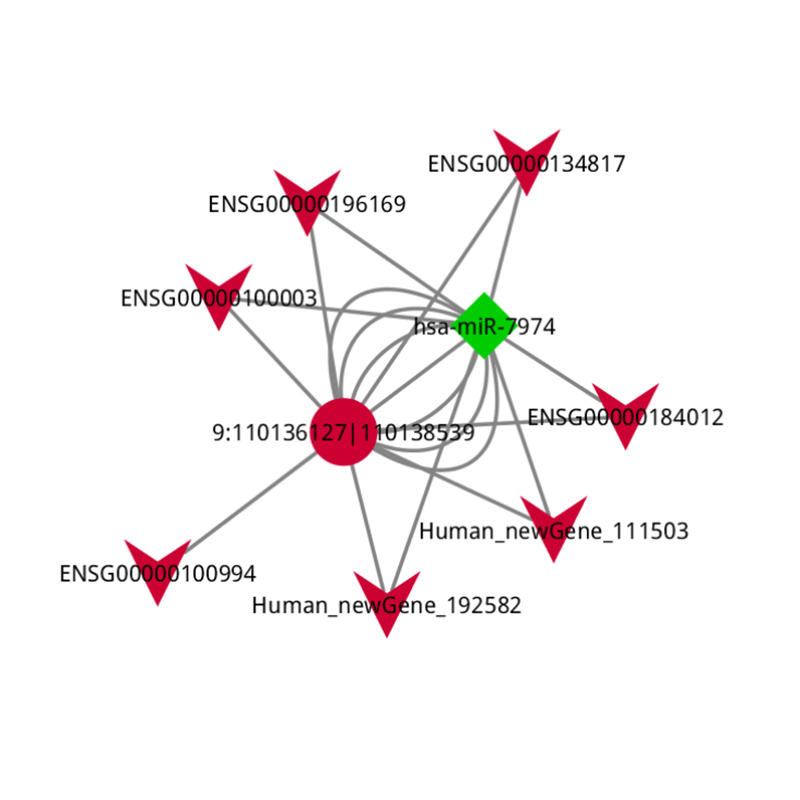
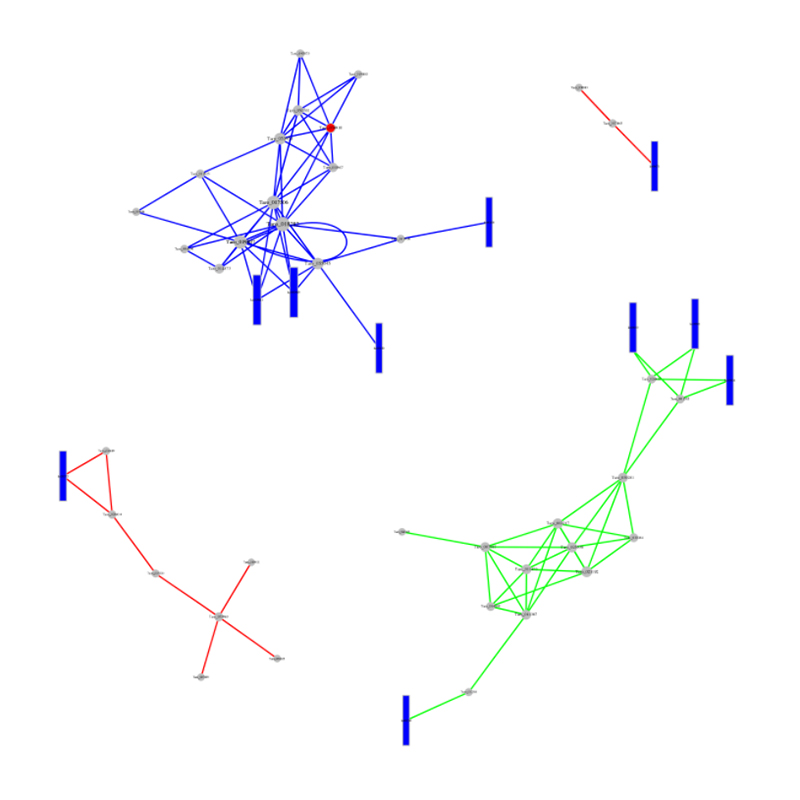
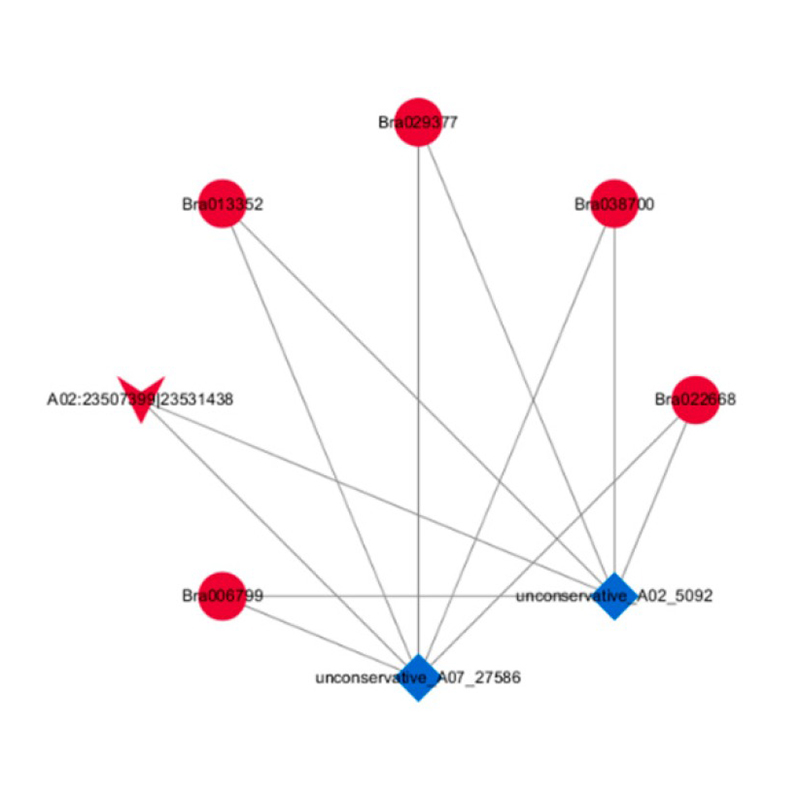
analiz CERNA
Eksplore pwogrè rechèch yo fasilite pa sèvis sekans transkriptòm BMKGENE 'nan yon koleksyon òganize nan piblikasyon.
Dai, Y. et al. (2022) 'Comprehensive ekspresyon Des mRNAs, lncRNAs ak miRNAs nan Kashin-Beck maladi idantifye pa RNA-sekans', molekilè omics, 18 (2), pp 154-166. doi: 10.1039/d1mo00370d.
Liu, N. Nan et al. (2022) 'Transcriptomes longè konplè analiz de frèt-rezistans nan APIs cerana nan Changbai Mountain pandan peryòd ivèrnaj.', Gene, 830, pp 146503-146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ et al. (2022) 'Multi-omics entegrasyon ki baze sou priyorite nan konpetisyon rezo andojèn RNA règleman nan ti kansè nan poumon selil: karakteristik molekilè ak kandida dwòg', fontyè nan onkoloji, 12, p. 904865. DOI: 10.3389/fonc.2022.904865/BIBTEX.
Xu, P. et al. (2022) 'analiz entegre nan lncRNA/circRNA-miRNA-mRNA ekspresyon Des revele nouvo Sur nan mekanis potansyèl an repons a rasin-ne nematod nan pistach', BMC Genomics, 23 (1), pp. 1–12. doi: 10.1186/s12864-022-08470-3/figi/7.
Yan, Z. et al. (2022) 'tout-transcriptome RNA sekans mete aksan sou mekanis yo molekilè ki asosye ak antretyen nan bon jan kalite postharvest nan bwokoli pa wouj ki ap dirije iradyasyon', postharvest byoloji ak teknoloji, 188, p. 111878. DOI: 10.1016/j.postharvbio.2022.111878.