Ki pa Peye-referans ki baze sou mRNA sekans-NGS
Karakteristik
● Kaptire poly mRNA anvan preparasyon bibliyotèk la
● Endepandan de nenpòt ki genomic referans: ki baze sou asanble de novo nan relve nòt, génération yon lis unigenes ki anote ak baz done miltip (NR, Swis-Prot, COG, Kog, eggnog, PFAM, GO, KEGG)
● Comprehensive analiz bioinformatic de ekspresyon jèn ak estrikti transkripsyon
Avantaj Sèvis
●Anpil ekspètiz: Avèk yon dosye track nan pwosesis sou 600,000 echantiyon nan BMKGENE, spanning divès kalite echantiyon tankou kilti selil, tisi, ak likid kò, ekip nou an pote yon richès nan eksperyans nan chak pwojè. Nou te fèmen avèk siksè plis pase 100,000 pwojè mRNA-seq nan domèn rechèch divès kalite.
●Kontwòl kalite solid: Nou aplike pwen kontwòl debaz atravè tout etap, ki soti nan echantiyon ak preparasyon bibliyotèk nan sekans ak bioinformatics. Sa a siveyans metikuleu asire livrezon an nan toujou-wo kalite rezilta yo.
● Anotasyon konplè: Nou itilize baz done miltip pou anote fonksyonèl jèn yo eksprime diferans (DEG) ak fè analiz la anrichisman korespondan, bay Sur sou pwosesis yo selilè ak molekilè kache repons lan transkriptòm.
●Post-lavant sipò: Angajman nou an pi lwen pase fini pwojè ak yon peryòd sèvis 3-mwa apre-sale. Pandan tan sa a, nou ofri pwojè swiv-up, depanaj asistans, ak Q & Yon sesyon adrese nenpòt kesyon ki gen rapò ak rezilta yo.
Egzanp kondisyon ak livrezon
| Bibliotèk | Estrateji sekans | Done rekòmande | Kontwòl kalite |
| Poly yon rich | Illumina PE150 Dnbseq-t7 | 6-10 GB | Q30≥85% |
Egzijans Egzanp:
Nukleotid:
| Conc. (Ng/μl) | Kantite lajan (μg) | Pite | Entegrite |
| ≥ 10 | ≥ 0.2 | OD260/280 = 1.7-2.5 OD260/230 = 0.5-2.5 Limite oswa pa gen pwoteyin oswa ADN kontaminasyon yo montre sou jèl. | Pou plant: Rin≥4.0; Pou bèt: Rin≥4.5; 5.0≥28s/18s≥1.0; Limite oswa pa gen okenn elevasyon debaz |
● Plant yo:
Rasin, tij oswa petal: 450 mg
Fèy oswa pitit pitit: 300 mg
Fwi: 1.2 g
● Animal:
Kè oswa trip: 300 mg
Viscera oswa sèvo: 240 mg
Misk: 450 mg
Zo, cheve oswa po: 1g
● Arthropods:
Ensèk: 6g
Crustacea: 300 mg
● san antye: 1 tib
● Selil yo: 106 selil
Rekòmande livrezon echantiyon
Veso: 2 ml tib santrifijez (papye fèblan pa rekòmande)
Etikèt Etikèt: Gwoup+replike EG A1, A2, A3; B1, B2, B3.
Chajman:
1. sèk-glas: echantiyon yo dwe chaje nan sache ak antere l 'nan sèk-glas.
2. Tib rnastable: echantiyon RNA ka seche nan tib estabilizasyon RNA (egzanp RNASTABLE®) ak anbake nan tanperati chanm.
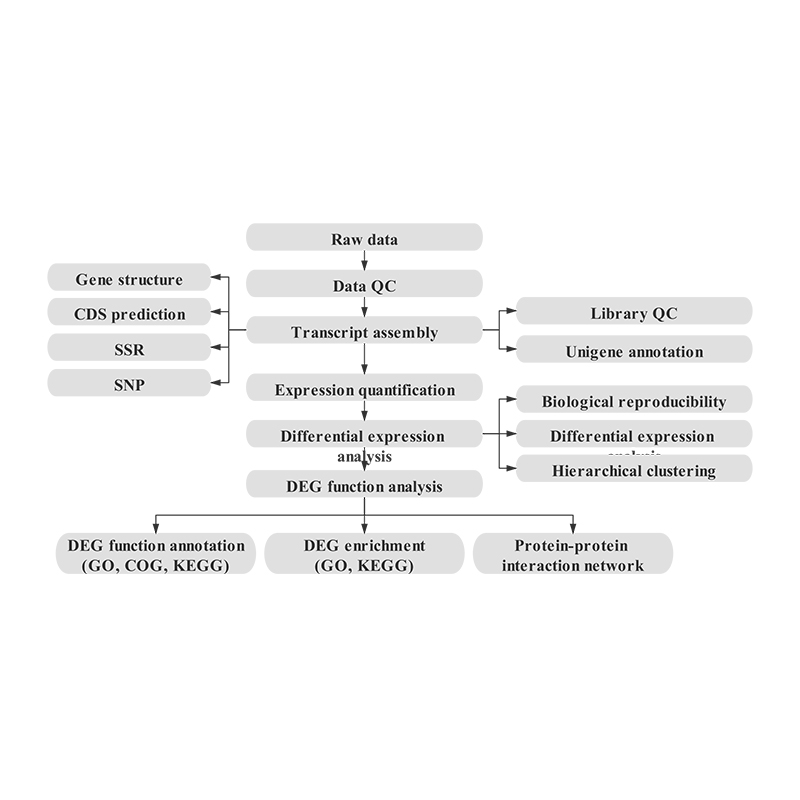
Sèvis koule travay

Eksperyans Design

Livrezon echantiyon

Ekstraksyon RNA

Konstriksyon Bibliyotèk

Syen

Done analiz

Apre-sale sèvis yo
Bioinformatics
Asanble transkriptòm ak seleksyon unigene
Anotasyon unigene
Egzanp korelasyon ak evalyasyon repwodiksyon byolojik
Diferansyèlman eksprime jèn (DEG)
Anotasyon fonksyonèl nan DEGs
Fonksyonèl anrichisman nan DEGs
Eksplore pwogrè yo fasilite pa BMKGENE a ekaryotik NGS sèvis sekans mRNA nan yon koleksyon òganize nan piblikasyon.
Shen, F. et al. (2020) 'De novo asanble transkriptòm ak ekspresyon jèn sèks-partial nan gonad yo nan pwason chat Amur (Silurus asotus)', jenomik, 112 (3), pp 2603-2614. doi: 10.1016/j.ygeno.2020.01.026.
Zhang, C. et al. (2016) 'analiz transkriptòm nan metabolis sikwoz pandan anpoul anfle ak devlopman nan zonyon (Allium Cepa L.)', Frontiers nan Syans Plant, 7 (septanm), p. 212763. DOI: 10.3389/fpls.2016.01425/BIBTEX.
Zhu, C. et al. (2017) 'de novo asanble, karakterizasyon ak anotasyon pou transkriptòm nan sarcocheilichthys sinensis', plo yon sèl, 12 (2). doi: 10.1371/journal.pone.0171966.
Zou, L. et al. (2021) 'De novo analiz transkriptòm bay Sur nan tolerans nan sèl nan Podocarpus macrophyllus anba Salinity Estrès', BMC Plant Biyoloji, 21 (1), pp 1–17. doi: 10.1186/s12870-021-03274-1/figi/9.