10x jenomik Visium espasyal transkriptòm
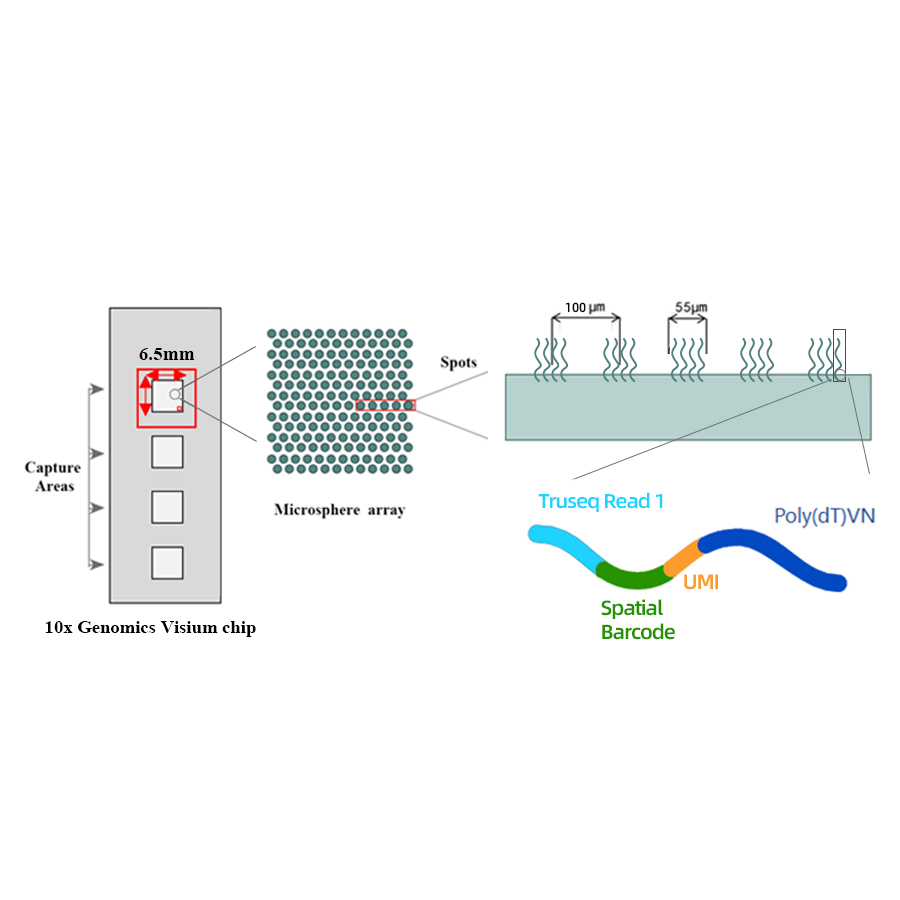
Konplo teknik

Karakteristik
● Rezolisyon: 100 µm
● Dyamèt tach: 55 µm
● Nimewo nan tach: 4992
● Zòn kaptire: 6.5 x 6.5 mm
● Chak plas barcoded chaje ak prime ki konpoze de 4 seksyon:
- Poly (DT) ke pou mRNA primin ak sentèz cDNA
- Idantifikatè inik molekilè (UMI) pou korije patipri anplifikasyon
- Barcode espasyal
- Sekans obligatwa nan yon pati nan li 1 sekans Jadendanfan
● H & E tach nan seksyon
Avantaj
●Yon sèl-sispann sèvis yo: Entegre tout eksperyans ak konpetans ki baze sou etap, ki gen ladan kri-sectioning, tach, optimize tisi, barcoding espasyal, preparasyon bibliyotèk, sekans ak bioinformatics.
● Ekip teknik trè kalifye: Avèk eksperyans nan plis pase 250 kalite tisi ak 100+ espès ki gen ladan moun, sourit, mamifè, pwason ak plant yo.
●Mizajou an tan reyèl sou pwojè a tout antye: ak kontwòl konplè sou pwogrè eksperimantal.
●Comprehensive estanda bioinformatics:Pake gen ladan 29 analyses ak 100+ -wo kalite figi.
●Analiz done Customized ak vizyalizasyon: Disponib pou demann rechèch diferan.
●Si ou vle analiz jwenti ak sèl-selil mRNA sekans
Espesifikasyon
| Egzanp kondisyon | Bibliotèk | Estrateji sekans | Done rekòmande | Kontwòl kalite |
| Oct-entegre echantiyon cryo (Optimal dyamèt: approx. 6x6x6 mm³) 2 blòk pou chak echantiyon | 10x Visium CDNA Bibliyotèk | Illumina PE150 | 50k PE li pou chak plas (60GB) | Rin> 7 |
Pou plis detay sou gid preparasyon echantiyon ak workflow sèvis, tanpri santi yo lib pou pale ak yonEkspè bmkgene
Sèvis workflow
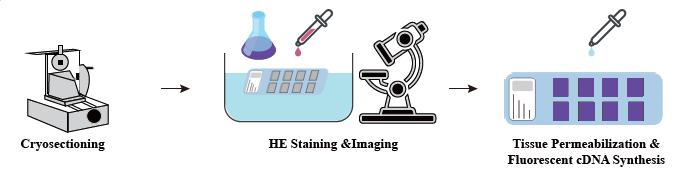
Nan faz nan preparasyon echantiyon, se yon premye jijman ekstraksyon RNA esansyèl fèt asire yon RNA-wo kalite ka jwenn. Nan etap nan optimize tisi seksyon yo tache ak vizualiz ak kondisyon yo permeabilization pou mRNA lage nan tisi yo optimisé. Se pwotokòl la optimize Lè sa a, aplike pandan konstriksyon bibliyotèk, ki te swiv pa sekans ak analiz done.
Workflow nan sèvis konplè enplike nan dènye an tan reyèl ak konfimasyon kliyan yo kenbe yon bouk fidbak reponn, asire lis ekzekisyon pwojè.

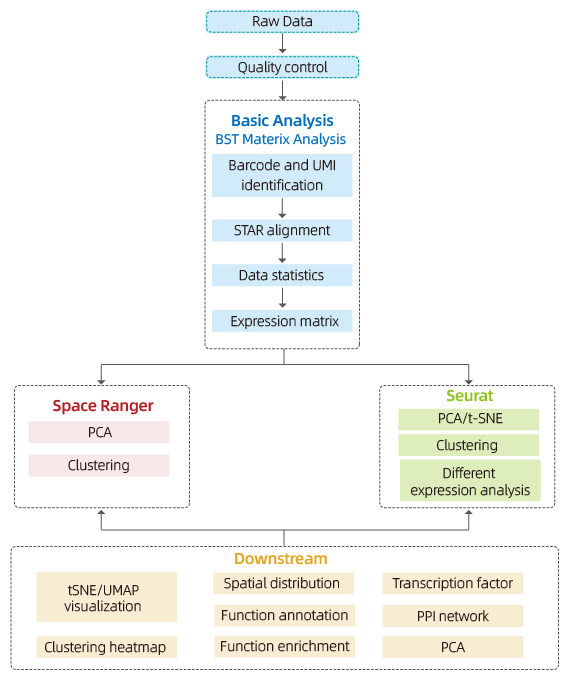
Gen ladan analiz sa a:
Kontwòl Kalite Done:
o Done pwodiksyon ak bon jan kalite distribisyon nòt
o Deteksyon jèn pou chak plas
o pwoteksyon tisi
Analiz enteryè-echantiyon:
o Richès jèn
o Gwoupman tach, ki gen ladan analiz dimansyon redwi
o Analiz ekspresyon diferans ant grap: idantifikasyon jèn makè yo
o Anotasyon fonksyonèl ak anrichisman nan jèn makè
Analiz entè-gwoup
o Re-konbinezon tach ki soti nan tou de echantiyon yo (eg malad ak kontwòl) ak re-gwoup
o Idantifikasyon nan jèn makè pou chak gwoup
o Anotasyon fonksyonèl ak anrichisman nan jèn makè
o ekspresyon diferans nan menm gwoup la ant gwoup yo
Analiz enteryè-echantiyon
Gwoupman tach

Idantifikasyon jèn makè ak distribisyon espasyal


Analiz entè-gwoup
Done konbinezon soti nan tou de gwoup yo ak re-gwoup
Makè jèn nan nouvo grap
Eksplore pwogrè yo fasilite pa BMKGENE a espasyal sèvis transcriptomics pa 10x Visium nan sa yo piblikasyon chin an tap:
Chen, D. et al. (2023) 'MTHL1, yon omolog potansyèl Drosophila nan GPCR adezyon mamifè, ki enplike nan reyaksyon antitumoral nan enjeksyon selil onkojèn nan mouch',Pwosedi Akademi Nasyonal Syans nan Etazini nan Amerik la, 120 (30), p. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'Steel pèmèt delimiter segondè-rezolisyon nan done transcriptomic spatiotemporal',Briefings nan bioinformatics, 24 (2), pp. 1–10. doi: 10.1093/bib/bbad068.
Liu, C. et al. (2022) 'Yon Atlas spatiotemporal nan òganojenèz nan devlopman nan flè Orchid',Rechèch asid nikleyik, 50 (17), pp 9724-9737. doi: 10.1093/nar/gkac773.
Wang, J. et al. (2023) 'Entegre transcriptomics espasyal ak sekans sèl-nwayo RNA revele potansyèl estrateji yo ki ka geri ou pou leiomyoma matris',Creole Journal of Byolojik Syans, 19 (8), pp 2515-2530. doi: 10.7150/ijbs.83510.