Secuenciación de ARN de núcleo único
Esquema Técnico
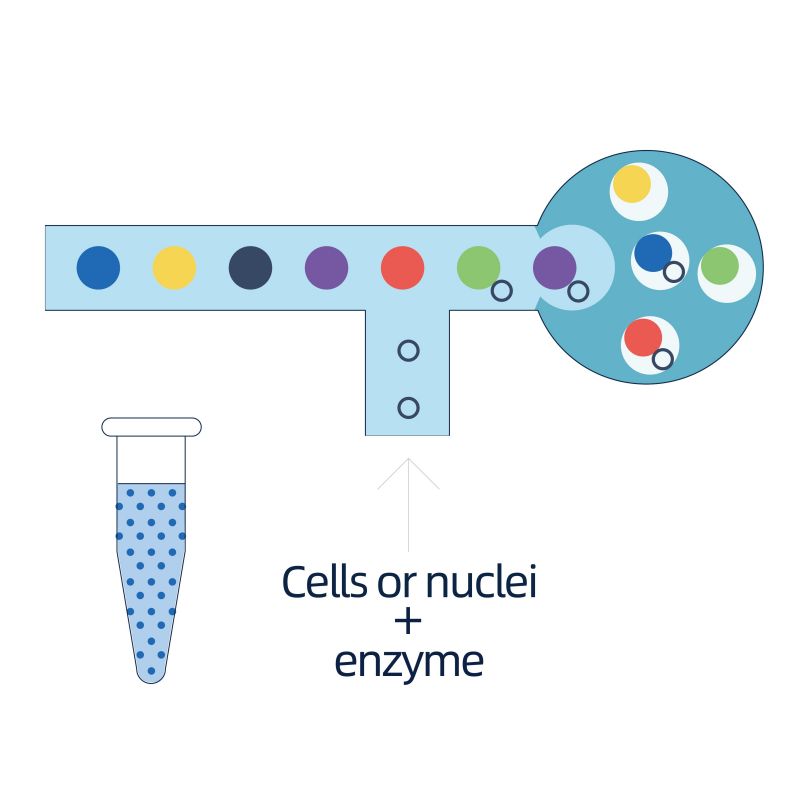
O illamento dos núcleos conséguese mediante 10× Genomics Chromium™, que consiste en un sistema microfluídico de oito canles con dobres cruzamentos. Neste sistema, unhas esferas de xel con códigos de barras e imprimación, encimas e un só núcleo están encapsulados nunha gota de aceite do tamaño dun nanolitro, xerando Gel Bead-in-Emulsion (GEM). Unha vez que se forman os GEM, realízase a lise celular e a liberación de códigos de barras en cada GEM. O ARNm transcríbense inversamente en moléculas de ADNc con códigos de barras 10× e UMI, que están ademais suxeitos á construción de bibliotecas de secuenciación estándar.

Características
● Preparación de suspensión de núcleo único a partir de tecidos conxelados
● Formación de perlas de xel en emulsión (GEM) seguida da síntese de cDNA
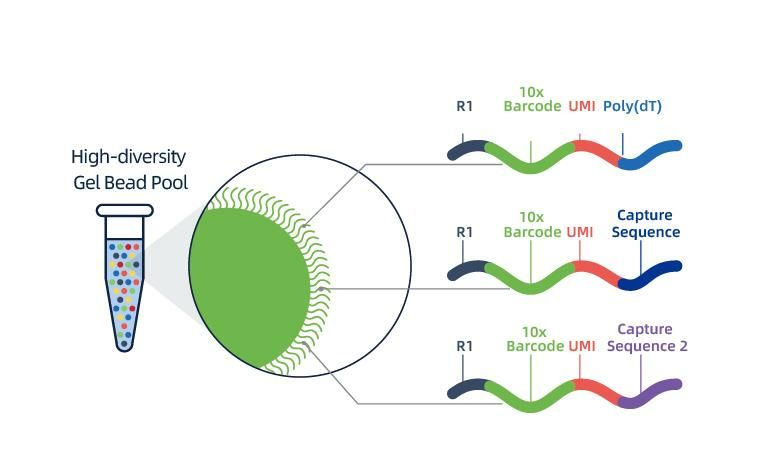
● Cada perla dun GEM está cargada con imprimacións compostas por 4 seccións:
cola poli(dT) para cebado de ARNm e síntese de ADNc,
Identificador molecular único (UMI) para corrixir o sesgo de amplificación
10x código de barras
Secuencia de unión do cebador de secuenciación de lectura parcial 1
Vantaxes
A secuenciación de ARN dun só núcleo elude as limitacións da secuenciación de ARN unicelular, permitindo:
● O uso de mostras conxeladas e non só limitadas a mostras frescas
● Baixo estrés das células conxeladas en comparación co tratamento enzimático de células frescas, reflectido nos datos do transcriptoma en forma de xenes menos inducidos polo estrés.
● Non é necesaria a eliminación previa de glóbulos vermellos
● Diámetro celular ilimitado
● Gran variedade de mostras aptas para a análise, incluídos os tipos de tecidos complexos e fráxiles que son propensos a aglutinación ou destrución celular durante a disociación do tecido.
Mostras que non se poden analizar mediante a secuenciación de ARN unicelular e son aptas para a secuenciación de ARN de núcleo único:
| Célula/Tecido | Razón |
| Tecido conxelado non fresco | Non se poden obter organizacións novas ou gardadas desde hai moito tempo |
| Célula muscular, megacariocito, graxa... | O diámetro da cela é demasiado grande para entrar no instrumento |
| Fígado… | Demasiado fráxil para romper, incapaz de distinguir células individuais |
| Célula neuronal, cerebro... | Máis sensible, fácil de estresar, cambiará os resultados da secuenciación |
| Páncreas, Tiroides... | Rico en enzimas endóxenas, que afectan á produción de suspensión celular única |
Núcleo único vs
| Un único núcleo | Monocélula |
| Diámetro celular ilimitado | Diámetro celular: 10-40 μm |
| O material pode ser tecido conxelado | O material debe ser tecido fresco |
| Baixo estrés das células conxeladas | O tratamento con enzimas pode provocar reaccións de estrés celular |
| Non é necesario eliminar glóbulos vermellos | Os glóbulos vermellos deben ser eliminados |
| Nuclear expresa bioinformación | Toda a célula expresa bioinformación |
Especificacións
| Requisitos de mostra | Biblioteca | Estratexia de secuenciación | Datos recomendados | Control de calidade |
| Tecido animal ≥ 200 mg Tecido vexetal ≥ 400 mg | 10x biblioteca de ADNc de Genomics sn | Illumina PE150 | 100K lecturas PE por cela (100-200 Gb) | 700-1200 núcleos/μl e integridade dos núcleos observada ao microscopio |
Para obter máis detalles sobre a orientación de preparación de mostras e o fluxo de traballo do servizo, non dubide en falar con aExperto en BMKGENE
Fluxo de traballo do servizo

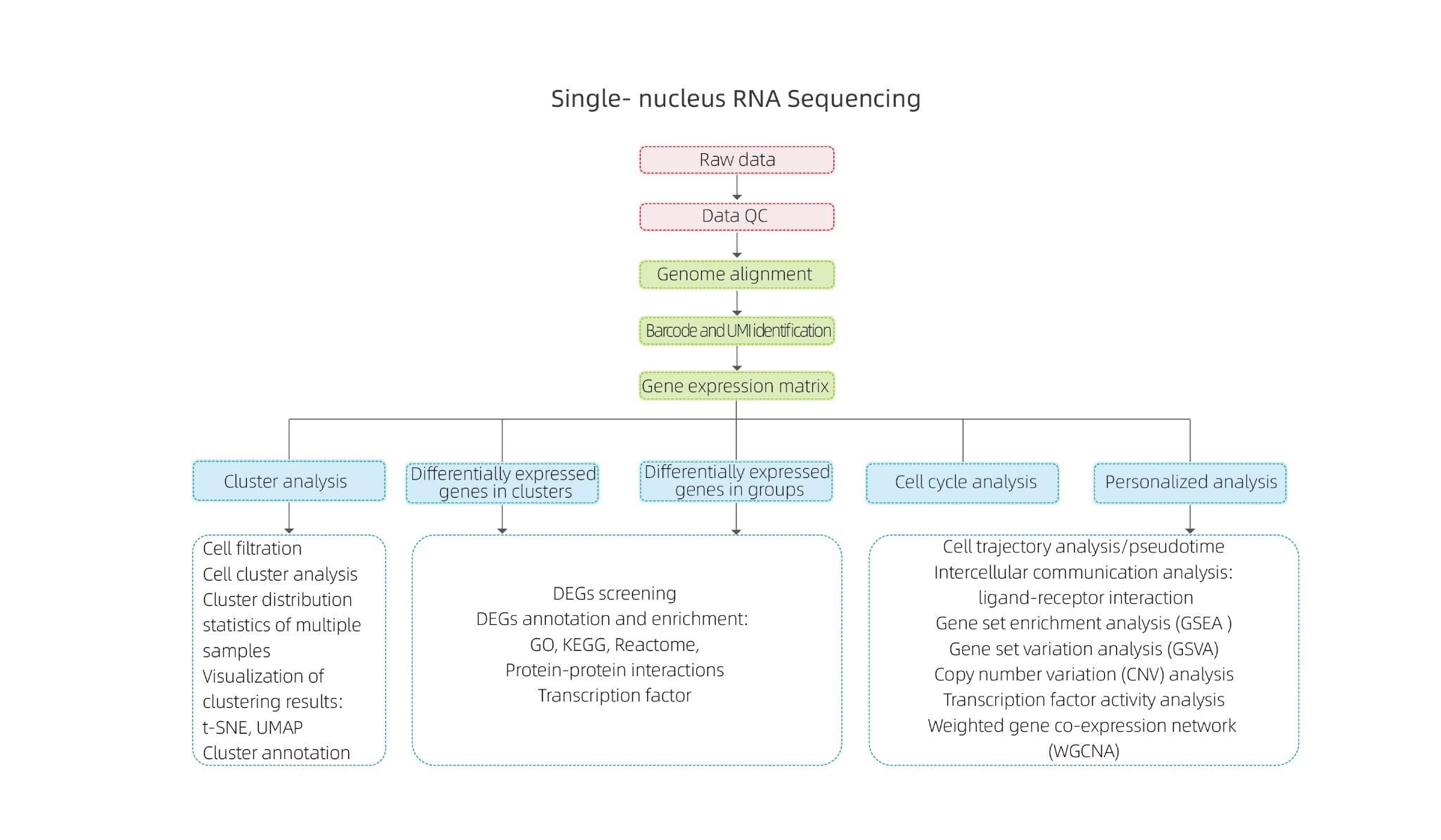
Inclúe a seguinte análise:
● Control de calidade: número de células, detección de xenes, identificación precisa de células, moléculas de ARN e cuantificación da expresión.
● Análise de mostra interna:
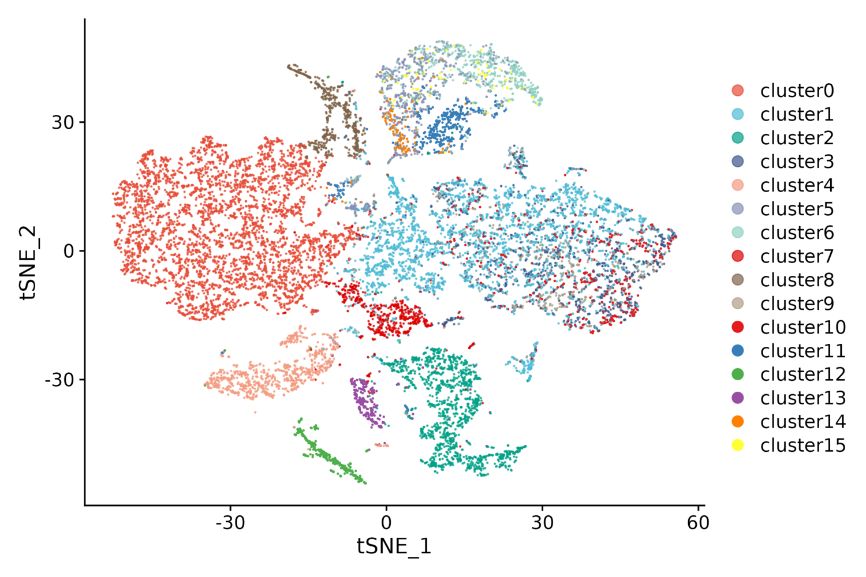
Agrupación celular e anotación de cluster
Análise da expresión diferencial: identificación de DEG en clusters
Anotación funcional e enriquecemento dos DEG de clúster
● Análise intergrupal:
Combinación de datos
Análise da expresión diferencial: identificación de DEG en grupos
Anotación funcional e enriquecemento dos DEG de grupo
● Análise avanzada:
Análise do ciclo celular
Análise pseudotemporal
Análise da comunicación celular (CellPhoneDB)
Análise de enriquecemento de conxuntos xenéticos (GSEA)
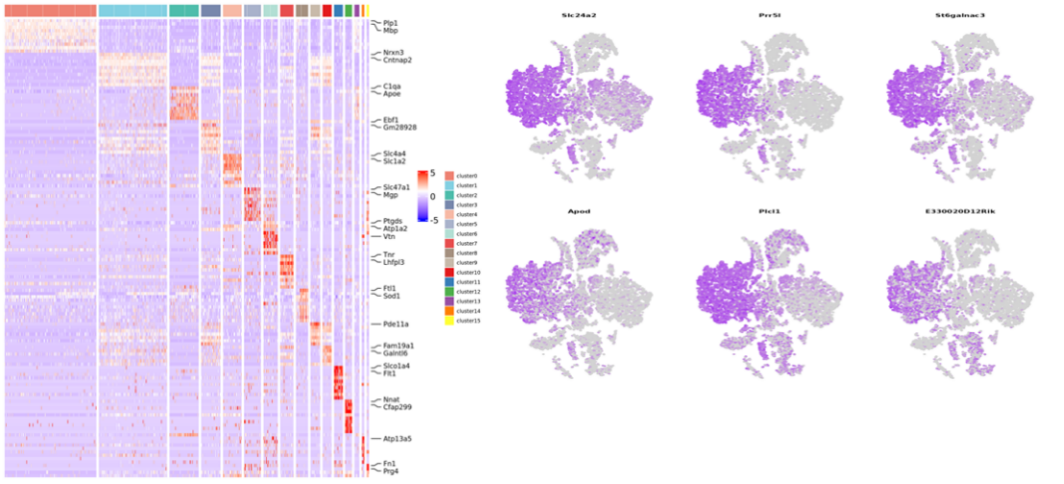
Análise da mostra interna
Agrupación celular:
Análise da expresión diferencial: DEG de clúster
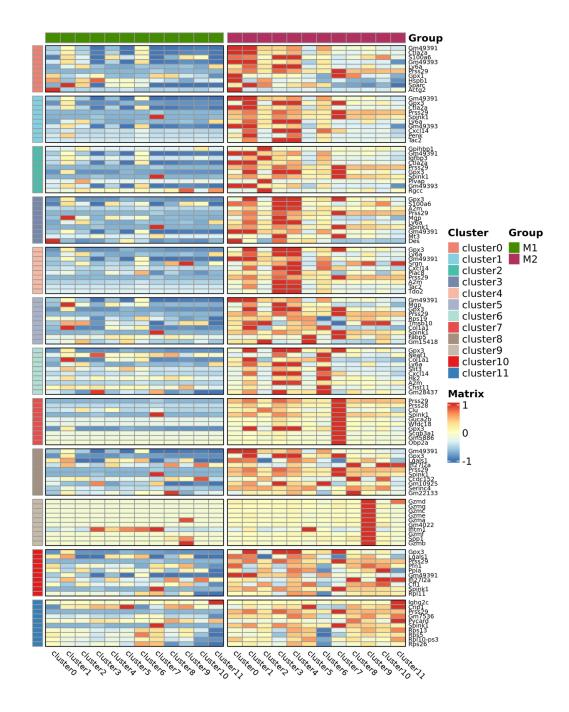
Análise intergrupal
Análise da expresión diferencial: Grupo DEG
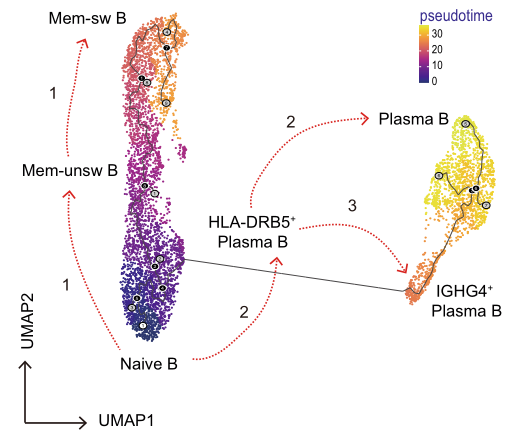
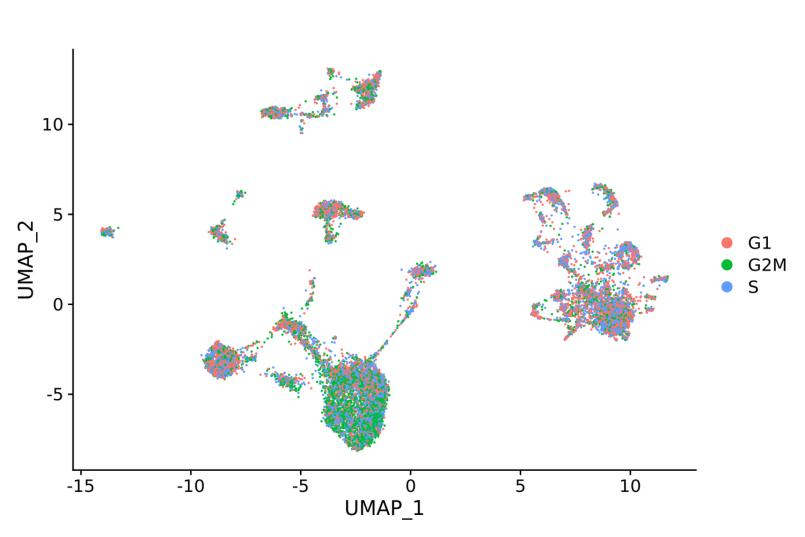
Análise avanzada:
Análise pseudotemporal:
Análise do ciclo celular:
Explore os avances facilitados polos servizos de secuenciación de ARN dun só núcleo de BMKGene por 10X Chromium nestas publicacións destacadas:
Wang, L. et al. (2021) "A análise transcriptómica unicelular revela a paisaxe inmune do pulmón na exacerbación da asma resistente aos esteroides",Actas da Academia Nacional de Ciencias dos Estados Unidos de América, 118(2), páx. e2005590118. doi: 10.1073/pnas.2005590118
Zheng, H. et al. (2022) "A Global Regulatory Network for Disregulated Gene Expression and Annormal Metabolic Signaling in Immune Cells in the Microenvironment of Graves' Disease and Hashimoto's Tiroiditis",Fronteiras en Inmunoloxía, 13, páx. 879824. doi: 10.3389/FIMMU.2022.879824/BIBTEX.
Tian, H. et al. (2023) "O transcriptoma unicelular descobre a heteroxeneidade e as respostas inmunitarias dos leucocitos despois da vacinación con Edwardsiella tarda inactivada na platija (Paralichthys olivaceus)".Acuicultura, 566, páx. 739238. doi: 10.1016/J.ACUICULTURA.2023.739238.
Yu, Y. et al. (2023) "A terapia fotodinámica mellora o resultado dos inhibidores do punto de control inmunitario mediante a remodelación da inmunidade antitumoral en pacientes con cancro gástrico".Cancro gástrico, 26 (5), páxinas 798–813. doi: 10.1007/S10120-023-01409-X/METRICS.