Representación reducida Secuenciación de bisulfito (RRBs)
Características do servizo
● Necesita un xenoma de referencia.
● O ADN lambda úsase para controlar a eficiencia de conversión de bisulfito.
● Tamén se monitoriza a eficiencia da dixestión MSPI.
● Dixestión dobre encima para mostras de plantas.
● Secuenciación en Illumina Novaseq.
Vantaxes do servizo
●Alternativa rendible e eficiente aos WGB: habilitando a análise a realizarse a un custo máis baixo e con menores requisitos de mostra.
●Plataforma completa:Ofrece un excelente servizo de procesamento de mostras, construción de bibliotecas e secuenciación á análise de bioinformática.
●Ampla experiencia: Con proxectos de secuenciación de RRBS completados con éxito en diversas especies, BMKGene trae máis dunha década de experiencia, un equipo de análise altamente cualificado, contido integral e excelente soporte post-venda.
Especificacións de servizo
| Biblioteca | Estratexia de secuenciación | Saída de datos recomendada | Control de calidade |
| MSPI dixerida e biblioteca tratada con bisulfito | Illumina PE150 | 8 GB | Q30 ≥ 85% Conversión de bisulfito> 99% Eficiencia de corte MSPI> 95% |
Requisitos da mostra
| Concentración (ng/µl) | Cantidade total (µg) |
| |
| ADN xenómico | ≥ 30 | ≥ 1 | Degradación ou contaminación limitada |
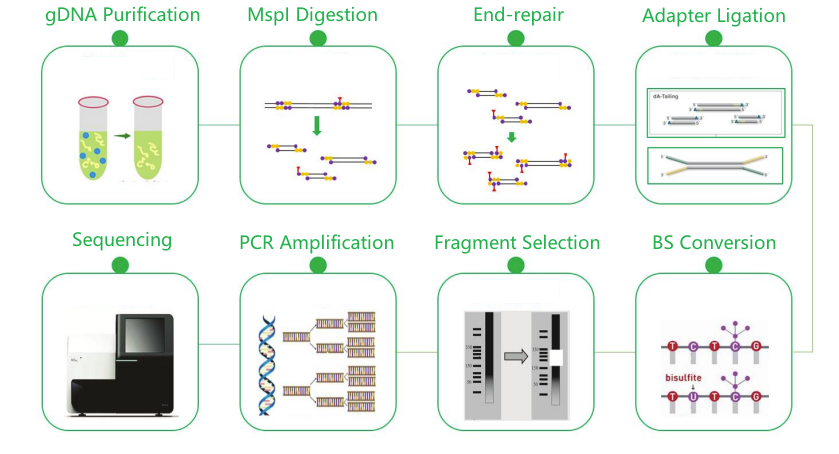
Fluxo de traballo de servizo

Entrega de mostra

Construción da biblioteca

Secuenciación

Análise de datos
Servizos despois da venda
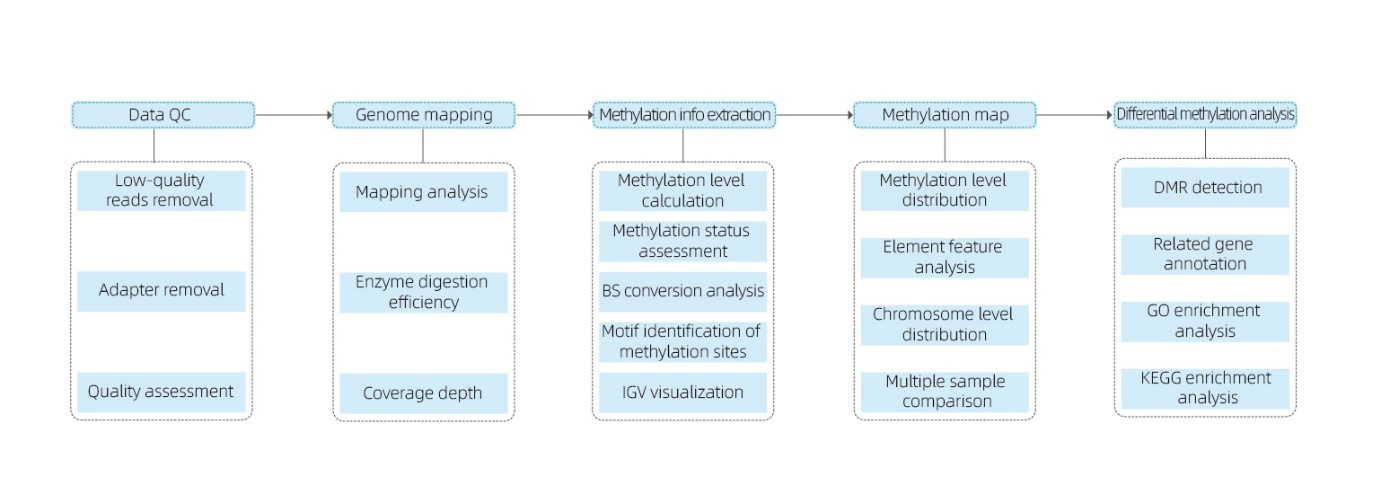
Inclúe a seguinte análise:
● Control de calidade de secuenciación en bruto;
● Mapeo para o xenoma de referencia;
● Detección de bases metiladas de 5mc e identificación de motivos;
● Análise da distribución de metilación e comparación de mostras;
● Análise de rexións metiladas diferencialmente (DMRs);
● anotación funcional de xenes asociados a DMRs.
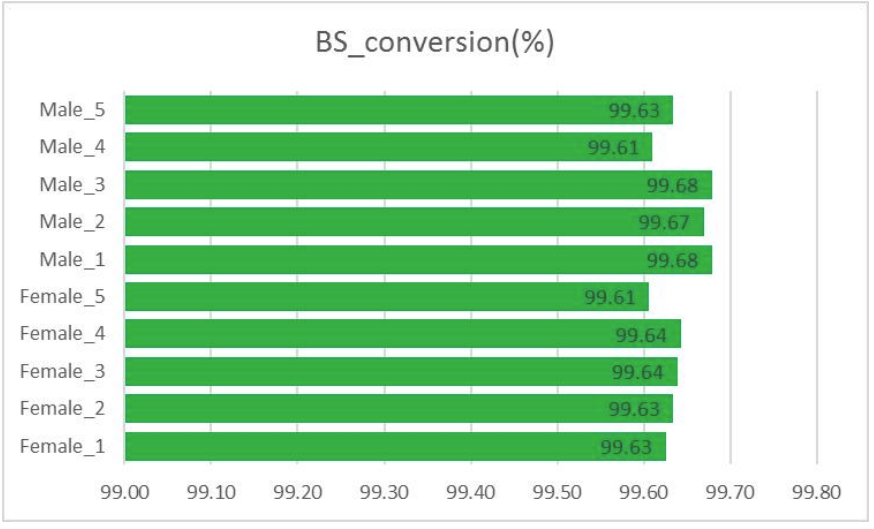
Control de calidade: eficiencia da dixestión (no mapeo do xenoma)
Control de calidade: conversión de bisulfito (en extracción de información de metilación)
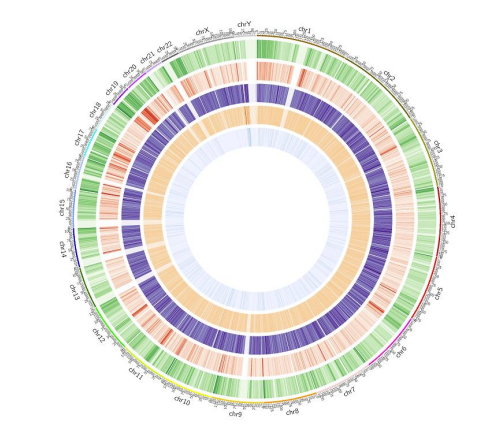
Mapa de metilación: Distribución de 5mc de metilación en todo o xenoma
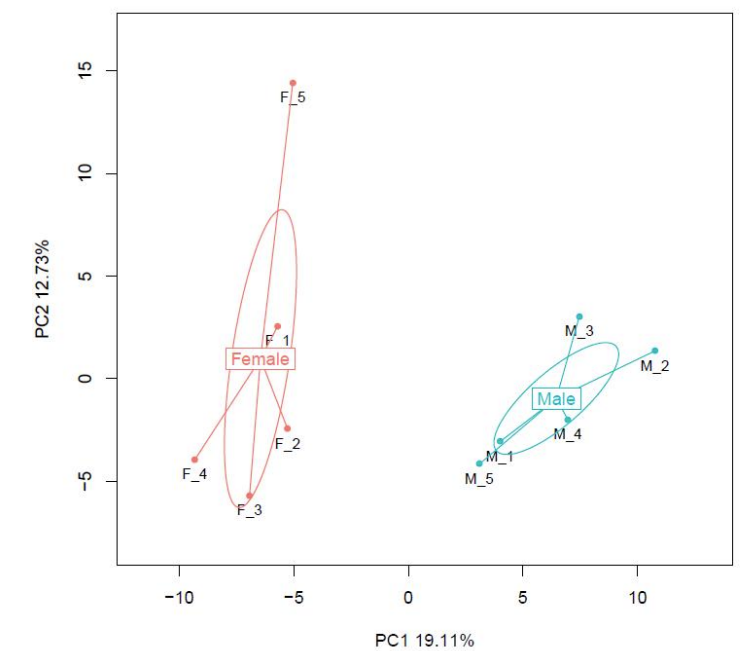
Comparación de mostras: análise de compoñentes principais
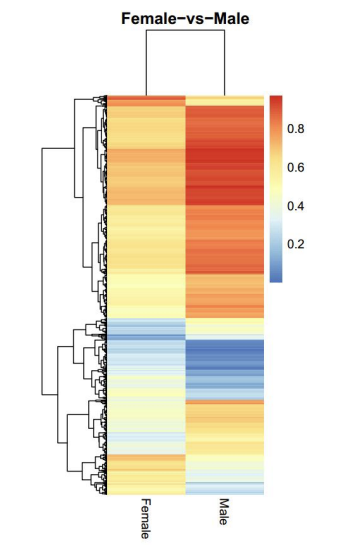
Análise de rexións metiladas de forma diferente (DMRS): Mapa de calor
Explora os avances da investigación facilitados polos servizos de secuenciación de bisulfitos do xenoma de BMKGene a través dunha colección curada de publicacións.
Li, Z. et al. (2022) "Reprogramación de alta fidelidade en células similares a Leydig mediante activación CRISPR e factores paracrinos",PNAS Nexus, 1 (4). doi: 10.1093/pnasnexus/pgac179.
Tian, H. et al. (2023) 'Análise de metilación de ADN en todo o xenoma da composición corporal nos xemelgos monozigotos chineses',Revista Europea de Investigación Clínica, 53 (11), p. E14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) 'Metilación do ADN e relación cintura-cadeira: un estudo de asociación en todo o epigenoma en xemelgos monozigotos chineses',Revista de investigación endocrinolóxica, 45 (12), pp. 2365–2376. doi: 10.1007/s40618-022-01878-4.