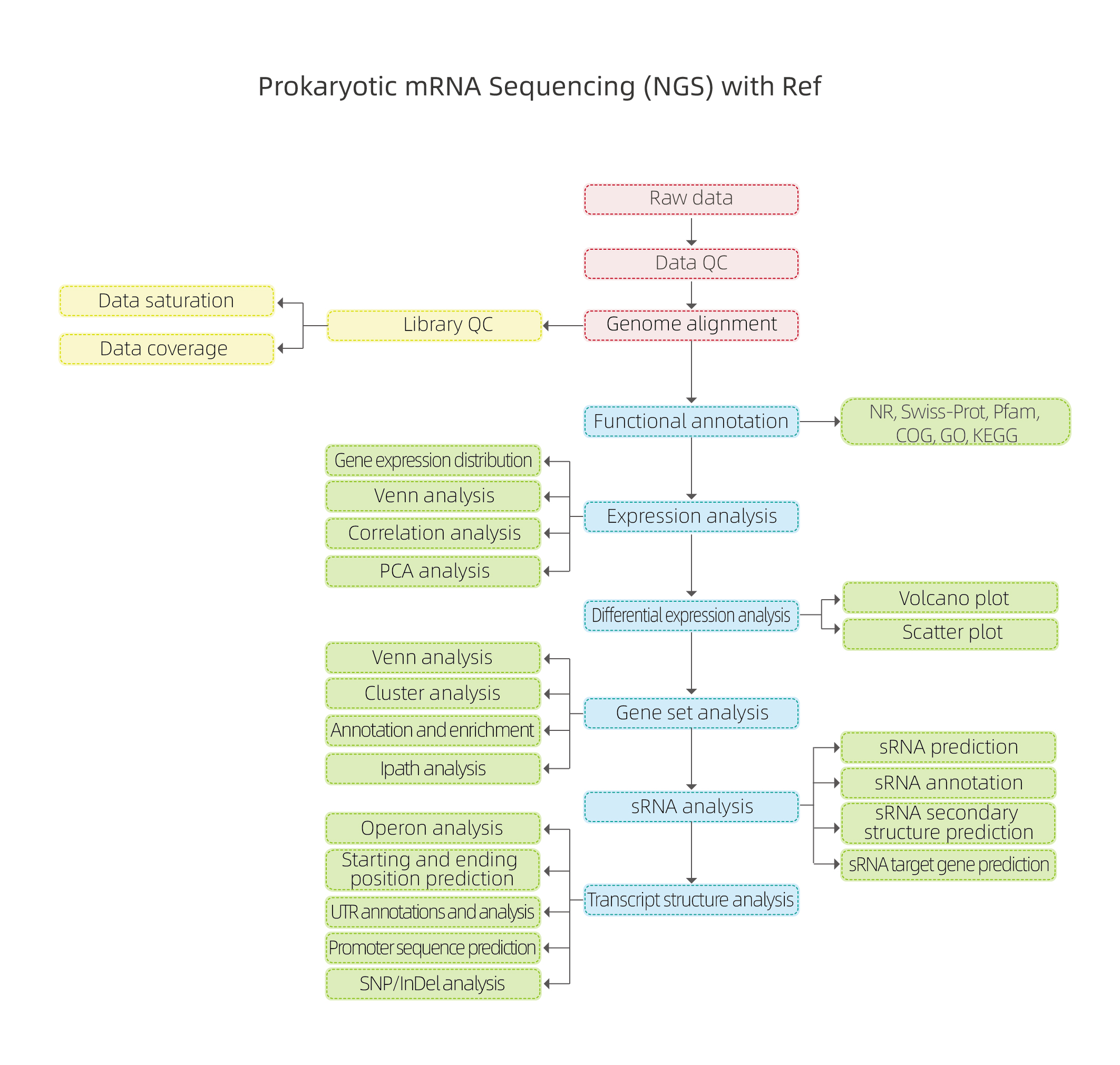
Secuenciación de ARN procariota
Características
● O procesamento da mostra de ARN implicou o esgotamento do ARNr seguido da preparación direccional da biblioteca de ARN.
● Análise bioinformática baseada no aliñamento a un xenoma de referencia
● A análise inclúe a expresión xénica e os DEG pero tamén a estrutura da transcrición e a análise de ARNs
Vantaxes do servizo
●Control de Calidade Riguroso: implementamos puntos de control básicos en todas as etapas, desde a preparación de mostras e bibliotecas ata a secuenciación e a bioinformática. Este seguimento minucioso garante a entrega de resultados de alta calidade constantemente.
●Datos de secuenciación específicos da cadea: debido a que a preparación da biblioteca de ARN é direccional, permitindo a identificación de transcricións antisentido.
●Análise completa adaptada aos transcriptomas procariotas: o pipeline bioinformático inclúe non só a análise da expresión xenética senón tamén a análise da estrutura da transcrición, incluíndo a identificación de operóns, UTR e promotores. Tamén inclúe análise de sRNAs, é dicir, anotación e predición da estrutura secundaria e obxectivos.
●Soporte posvenda: o noso compromiso vai máis aló da finalización do proxecto cun período de servizo posvenda de 3 meses. Durante este tempo, ofrecemos seguimento do proxecto, asistencia para a resolución de problemas e sesións de preguntas e respostas para resolver calquera dúbida relacionada cos resultados.
Requisitos de mostra e entrega
| Biblioteca | Estratexia de secuenciación | Datos recomendados | Control de calidade |
| Biblioteca direccional esgotada de ARNr | Illumina PE150 | 1-2 Gb | Q30≥85% |
Requisitos de mostra:
| Conc. (ng/μl) | Cantidade (μg) | Pureza | Integridade |
| ≥ 50 | ≥ 1 | OD260/280=1,8-2,0 OD260/230=1,0-2,5 Contaminación limitada ou nula de proteínas ou ADN no xel. | RIN ≥ 6,5 |
Entrega de mostra recomendada
Recipiente: tubo de centrífuga de 2 ml (non se recomenda papel de aluminio)
Etiquetaxe da mostra: grupo+réplica, por exemplo, A1, A2, A3; B1, B2, B3.
Envío:
1. Xeo seco: as mostras deben ser embaladas en bolsas e enterradas en xeo seco.
2. Tubos RNAstable: as mostras de ARN pódense secar nun tubo de estabilización de ARN (por exemplo, RNAstable®) e enviarse a temperatura ambiente.
Fluxo de traballo do servizo

Entrega da mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
Fluxo de traballo de análise bioinformática
Inclúe a seguinte análise:
● Control de calidade dos datos brutos
● Aliñación co xenoma de referencia
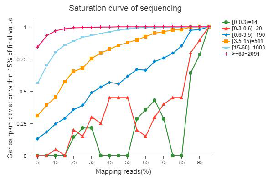
● Avaliación da calidade da biblioteca: aleatoriedade da fragmentación do ARN, tamaño da inserción e saturación da secuenciación
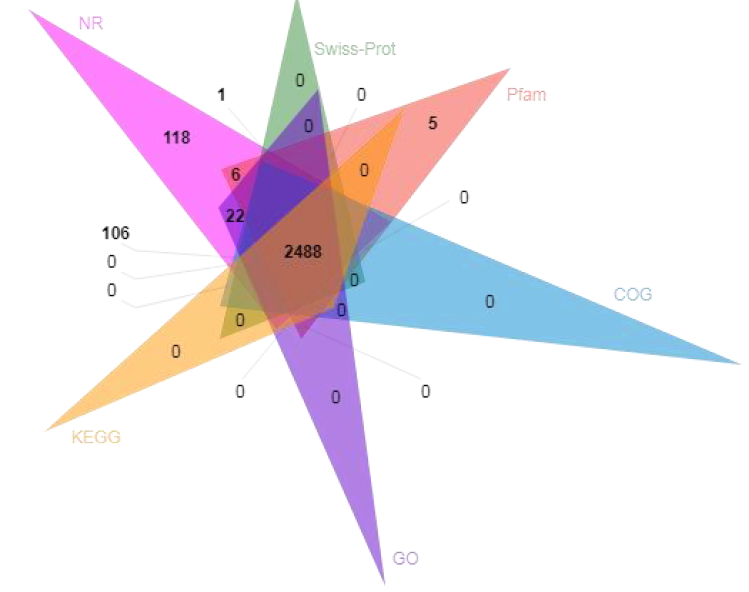
● Anotación funcional dos xenes codificantes previstos
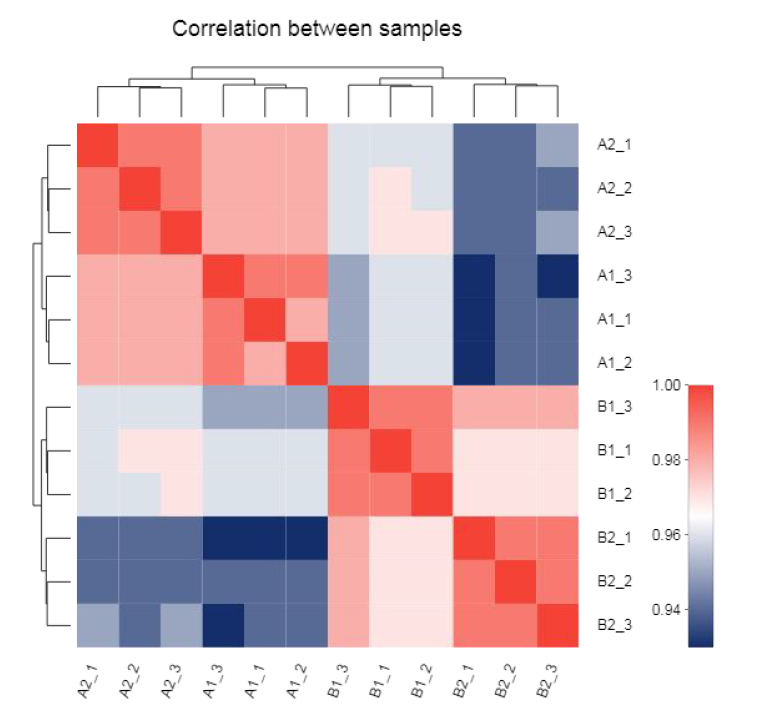
● Análise da expresión: correlación e análise de compoñentes principais (PCA)
● Expresión xenética diferencial (DEG)
● Anotación funcional e enriquecemento dos DEG
● Análise de sRNA: predición, anotación, obxectivo e predición da estrutura secundaria
● Análise da estrutura da transcrición: operóns, posicións iniciais e finais, rexión non traducida (UTS), promotor e análise SNP/InDel
Saturación de secuencias
Anotación funcional de xenes codificantes
Correlación entre mostras
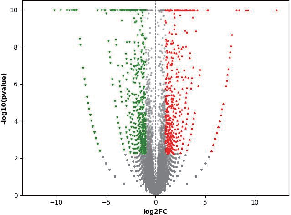
Análise de xenes expresados diferenciais (DEG).
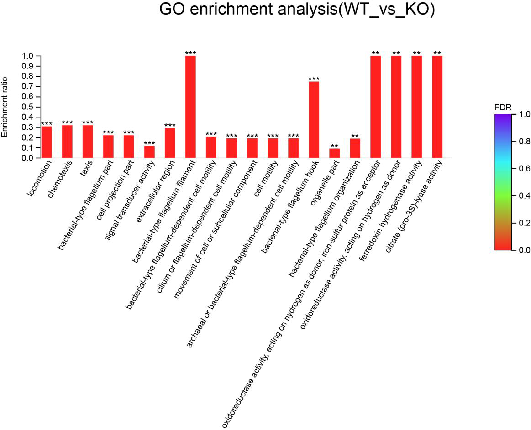
Análise do enriquecemento funcional
anotación de sRNA
Explore os avances facilitados polos servizos de secuenciación de ARNm de lonxitude completa Nanopore de BMKGene nesta publicación destacada.
Guan, CP et al. (2018) "Cambios globais do transcriptoma de Staphylococcus epidermidis formador de biopelículas que responden aos alcaloides totais de Sophorea alopecuroides",Revista polaca de microbioloxía, 67(2), páx. 223. doi: 10.21307/PJM-2018-024.