-

Secuenciación metaxenómica -NGS
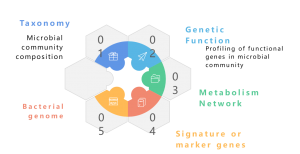
Un metaxenoma é unha colección do material xenético total dunha comunidade mixta de organismos, como os metaxenomas ambientais e humanos. Contén xenomas de microorganismos cultivables e non cultivables. A secuenciación metaxenómica de escopeta con NGS permite o estudo destas paisaxes xenómicas intrincadas incrustadas en mostras ambientais proporcionando algo máis que un perfís taxonómicos, proporcionando tamén información granular sobre a diversidade de especies, a dinámica da abundancia e as estruturas poboacionais complexas. Ademais dos estudos taxonómicos, a metaxenómica de escopetas tamén ofrece unha perspectiva da xenómica funcional, que permite a exploración de xenes codificados e os seus papeis putativos nos procesos ecolóxicos. Finalmente, o establecemento de redes de correlación entre elementos xenéticos e factores ambientais contribúe a unha comprensión holística da intrincada interacción entre as comunidades microbianas e os seus antecedentes ecolóxicos. En conclusión, a secuenciación metaxenómica eríxese como un instrumento fundamental para desvelar as complejidades xenómicas de diversas comunidades microbianas, iluminando as relacións multifacéticas entre a xenética e a ecoloxía dentro destes ecosistemas complexos.
Plataformas: Illumina NovaSeq e DNBSEQ-T7
-

Secuenciación metagenómica-TGS
Un metaxenoma é unha colección de material xenético dunha comunidade mixta de organismos, como os metaxenomas ambientais e humanos. Contén xenomas de microorganismos cultivables e non cultivables. A secuenciación metaxenómica permite o estudo destas intrincadas paisaxes xenómicas incrustadas en mostras ecolóxicas proporcionando algo máis que un perfil taxonómico. Tamén ofrece unha perspectiva da xenómica funcional explorando os xenes codificados e os seus papeis putativos nos procesos ambientais. Aínda que os enfoques tradicionais de escopeta con secuenciación Illumina foron amplamente utilizados nos estudos metaxenómicos, a chegada da secuenciación de lectura longa de Nanopore e PacBio cambiou o campo. A tecnoloxía Nanopore e PacBio melloran as análises bioinformáticas posteriores, especialmente a ensamblaxe do metaxenoma, garantindo montaxes máis continuas. Os informes indican que a metaxenómica baseada en Nanopore e PacBio xeraron con éxito xenomas bacterianos completos e pechados a partir de microbiomas complexos (Moss, EL, et al., Nature Biotech, 2020). A integración das lecturas de Nanopore coas lecturas de Illumina proporciona un enfoque estratéxico para a corrección de erros, mitigando a baixa precisión inherente de Nanopore. Esta combinación sinérxica aproveita os puntos fortes de cada plataforma de secuenciación, ofrecendo unha solución robusta para superar as limitacións potenciais e avanzar na precisión e fiabilidade das análises metaxenómicas.
Plataforma: Nanopore PromethION 48, Illumia e PacBio Revio
-

16S/18S/ITS Amplicon Sequencing-PacBio
Os xenes de ARNr 16S e 18S, xunto coa rexión Internal Transcribed Spacer (ITS), serven como marcadores de pegadas moleculares fundamentais debido á súa combinación de rexións altamente conservadas e hipervariables, o que os converte en ferramentas inestimables para caracterizar organismos procariotas e eucariotas. A amplificación e secuenciación destas rexións ofrecen un enfoque libre de illamento para investigar a composición e diversidade microbiana en varios ecosistemas. Aínda que a secuenciación de Illumina adoita dirixirse a rexións hipervariables curtas como V3-V4 de 16S e ITS1, demostrouse que se pode conseguir unha anotación taxonómica superior secuenciando a lonxitude total de 16S, 18S e ITS. Este enfoque integral dá lugar a maiores porcentaxes de secuencias clasificadas con precisión, conseguindo un nivel de resolución que se estende á identificación de especies. A plataforma de secuenciación en tempo real de moléculas únicas (SMRT) de PacBio destaca por ofrecer lecturas longas (HiFi) moi precisas que cobren os amplicóns de lonxitude total, rivalizando coa precisión da secuenciación de Illumina. Esta capacidade permite aos investigadores acadar unha vantaxe inigualable: unha visión panorámica da paisaxe xenética. A cobertura estendida eleva significativamente a resolución na anotación de especies, especialmente dentro das comunidades bacterianas ou fúngicas, o que permite unha comprensión máis profunda das complejidades das poboacións microbianas.
-

16S/18S/ITS Amplicon Sequencing-NGS
A secuenciación de amplicóns coa tecnoloxía Illumina, dirixida específicamente aos marcadores xenéticos 16S, 18S e ITS, é un método poderoso para descubrir a filoxenia, a taxonomía e a abundancia de especies dentro das comunidades microbianas. Este enfoque implica a secuenciación das rexións hipervariables dos marcadores xenéticos domésticos. Orixinalmente introducido como unha pegada dixital molecular porWoeses et alen 1977, esta técnica revolucionou o perfil do microbioma ao permitir análises sen illamento. A través da secuenciación de 16S (bacterias), 18S (fungos) e Internal Transcrited Spacer (ITS, fungos), os investigadores poden identificar non só especies abundantes senón tamén raras e non identificadas. Amplamente adoptada como unha ferramenta fundamental, a secuenciación de amplicóns converteuse en fundamental para discernir composicións microbianas diferenciais en diversos ambientes, incluíndo a boca humana, os intestinos, as feces e máis aló.
-

Re-secuenciación do xenoma total de bacterias e fungos
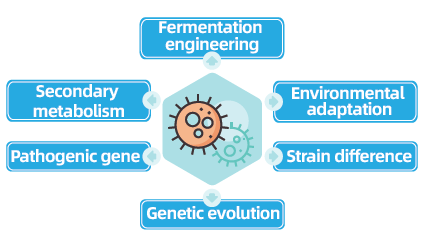
Os proxectos de resecuenciación do xenoma completo de bacterias e fungos son fundamentais para avanzar na xenómica microbiana ao permitir a realización e a comparación de xenomas microbianos. Isto facilita a enxeñaría de fermentación, a optimización dos procesos industriais e a exploración das vías secundarias do metabolismo. Ademais, a re-secuenciación de fungos e bacterias é fundamental para comprender a adaptación ambiental, optimizar as cepas e revelar a dinámica da evolución xenética, con amplas implicacións na medicina, a agricultura e a ciencia ambiental.
-

Secuenciación de ARN procariota
A secuenciación de ARN permite o perfil completo de todos os transcritos de ARN dentro das células en condicións específicas. Esta tecnoloxía de vangarda serve como unha potente ferramenta, revelando perfís de expresión xénica complexos, estruturas xenéticas e mecanismos moleculares asociados a diversos procesos biolóxicos. Amplamente adoptada na investigación fundamental, diagnóstico clínico e desenvolvemento de fármacos, a secuenciación de ARN ofrece información sobre as complejidades da dinámica celular e da regulación xenética. O noso procesamento de mostras de ARN procariota está adaptado para transcriptomas procariotas, que implica o esgotamento do ARNr e a preparación de bibliotecas direccionais.
Plataforma: Illumina NovaSeq
-

Secuenciación de metatranscriptomas
Aproveitando a tecnoloxía de secuenciación de Illumina, o servizo de secuenciación de metatranscriptoma de BMKGENE revela a expresión xenética dinámica dunha gran variedade de microbios, que abarca eucariotas ata procariotas e virus, dentro de ambientes naturais como o chan, a auga, o mar, as feces e o intestino. O noso servizo integral permite aos investigadores afondar nos perfís completos de expresión xénica das comunidades microbianas complexas. Ademais da análise taxonómica, o noso servizo de secuenciación de metatranscriptoma facilita a exploración do enriquecemento funcional, arroxando luz sobre os xenes expresados de forma diferencial e os seus papeis. Descubra unha gran cantidade de coñecementos biolóxicos mentres navega polas complexas paisaxes da expresión xénica, a diversidade taxonómica e as dinámicas funcionais dentro destes diversos nichos ambientais.
-

Asemblea do xenoma de fungos de novo
BMKGENE ofrece solucións versátiles para os xenomas de fungos, atendendo a diversas necesidades de investigación e a integridade do xenoma desexado. Utilizar só a secuenciación de Illumina de lectura curta permite a xeración dun xenoma borrador. As lecturas curtas e a secuenciación de lecturas longas mediante Nanopore ou Pacbio combínanse para obter un xenoma de fungos máis refinado con contigs máis longos. Ademais, a integración da secuenciación Hi-C mellora aínda máis as capacidades, permitindo a obtención dun xenoma completo a nivel cromosómico.
-

Ensamble de xenoma bacteriano de novo
Ofrecemos un completo servizo de montaxe de xenoma bacteriano, garantindo 0 lagoas. Isto é posible integrando tecnoloxías de secuenciación de lectura longa, como Nanopore e PacBio para a montaxe e secuenciación de lectura curta con Illumina para a validación da montaxe e a corrección de erros das lecturas ONT. O noso servizo ofrece o fluxo de traballo bioinformático completo desde a montaxe, a anotación funcional e a análise bioinformática avanzada, cumprindo obxectivos de investigación específicos. Este servizo permite o desenvolvemento de xenomas de referencia precisos para diversos estudos xenéticos e xenómicos. Ademais, constitúe a base para aplicacións como a optimización de cepas, a enxeñaría xenética e o desenvolvemento de tecnoloxía microbiana, garantindo datos xenómicos fiables e libres de lagoas cruciais para avanzar nos coñecementos científicos e na innovación biotecnolóxica.