Secuenciación de exomas enteiros humanos
Características do servizo
● Dous paneis exomas dispoñibles en función do enriquecemento de obxectivos con sondas: seguro que selecciona Human All Exon V6 (Agilent) e XGEN Exome Hibridation Panel V2 (IDT).
● Secuenciación en Illumina Novaseq.
● Pipeline bioinformático dirixido á análise da enfermidade ou á análise do tumor.
Vantaxes do servizo
●Obxectivo a rexión de codificación de proteínas: Ao capturar e secuenciar rexións de codificación de proteínas, as HWEs utilízanse para revelar variantes relacionadas coa estrutura proteica.
●Custo eficaz:As HWS producen aproximadamente o 85% das mutacións asociadas á enfermidade humana do 1% do xenoma humano.
●Alta precisión: Con alta profundidade de secuenciación, HWES facilita a detección de variantes comúns e variantes raras con frecuencias inferiores ao 1%.
●Control de calidade rigoroso: Implementamos cinco puntos de control básicos en todas as etapas, desde a preparación da mostra e da biblioteca ata a secuenciación e a bioinformática. Este minucioso control asegura a entrega de resultados de alta calidade constantemente.
●Análise bioinformática completa: O noso pipeline vai máis alá de identificar variacións ao xenoma de referencia, xa que incorpora unha análise avanzada deseñada para abordar específicamente as cuestións de investigación relacionadas con aspectos xenéticos de enfermidades ou análise do tumor.
●Soporte post-venda:O noso compromiso esténdese máis alá da finalización do proxecto cun período de servizo posterior a 3 meses. Durante este tempo, ofrecemos un seguimento do proxecto, asistencia de solución de problemas e sesións de Q&A para abordar as consultas relacionadas cos resultados.
Especificacións de mostra
| Estratexia de captura de exon | Estratexia de secuenciación | Saída de datos recomendada |
| SEGURO SELECT HUMANO All Exon V6 (Agilent) ou XGEN Exome Hibridation Panel V2 (IDT)
| Illumina Novaseq PE150 | 5-10 GB Para trastornos mendelianos/enfermidades raras:> 50x Para mostras de tumor: ≥ 100x |
Requisitos da mostra
| Tipo de mostra
| Cantidade(Qubit®)
| Concentración | Volume
| Pureza (Nanodrop ™) |
|
ADN xenómico
| ≥ 50 ng | ≥ 6 ng/µl | ≥ 15 µl | OD260/280 = 1,8-2,0 sen degradación, sen contaminación
|
Bioinformática

A análise bioinformática de mostras de enfermidade HWES inclúe:
● Datos de secuenciación QC
● Aliñamento do xenoma de referencia
● Identificación de SNPs e Indels
● Anotación funcional de SNPs e Indels

A análise bioinformática das mostras de tumor inclúe:
● Datos de secuenciación QC
● Aliñamento do xenoma de referencia
● Identificación de SNPs, indels e variacións somáticas
● Identificación de variantes de liña xerminal
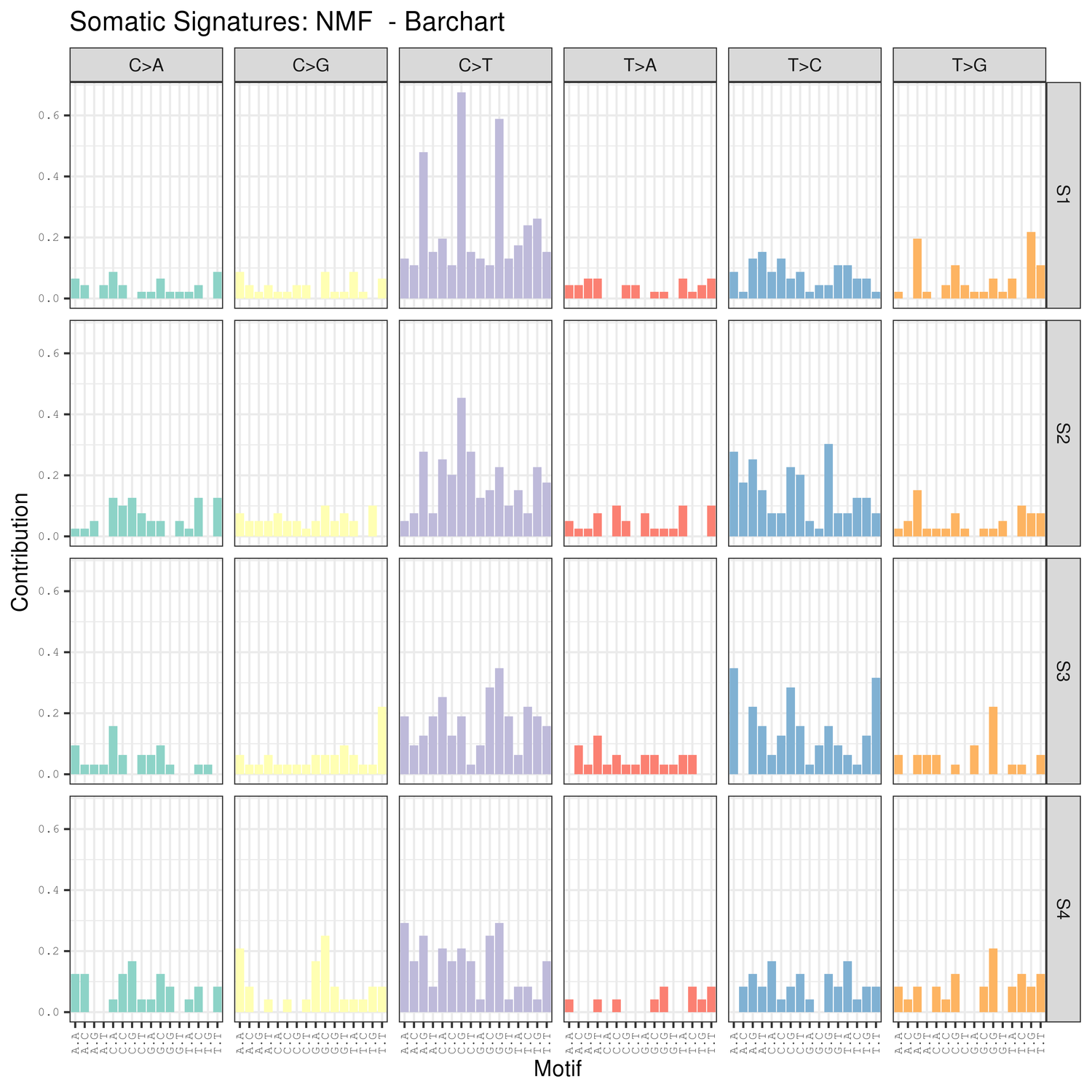
● Análise de firmas de mutación
● Identificación de xenes de unidade baseados en mutacións de ganancia de función
● anotación de mutación a nivel de susceptibilidade á droga
● Análise de heteroxeneidade - cálculo da pureza e da ploidía
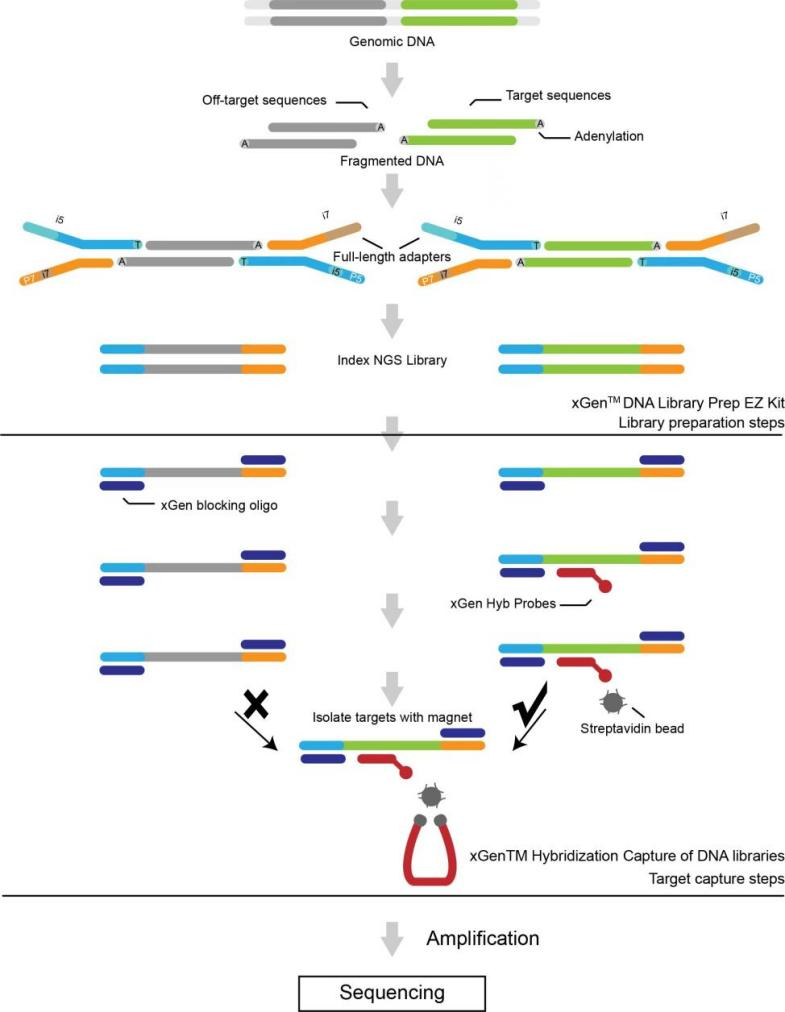
Fluxo de traballo de servizo

Entrega de mostra

Extracción de ADN

Construción da biblioteca

Secuenciación

Análise de datos

Entrega de datos

Servizos despois da venda
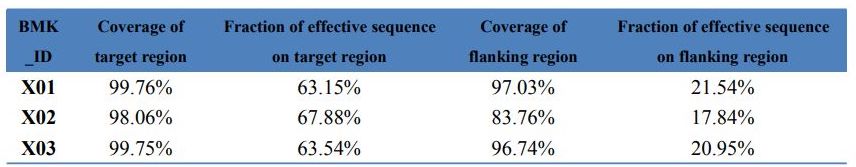
Datos QC - Estatísticas da captura de Exome
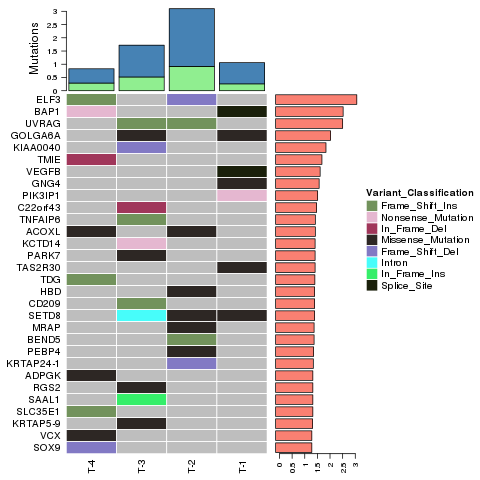
Identificación da variante - Indels
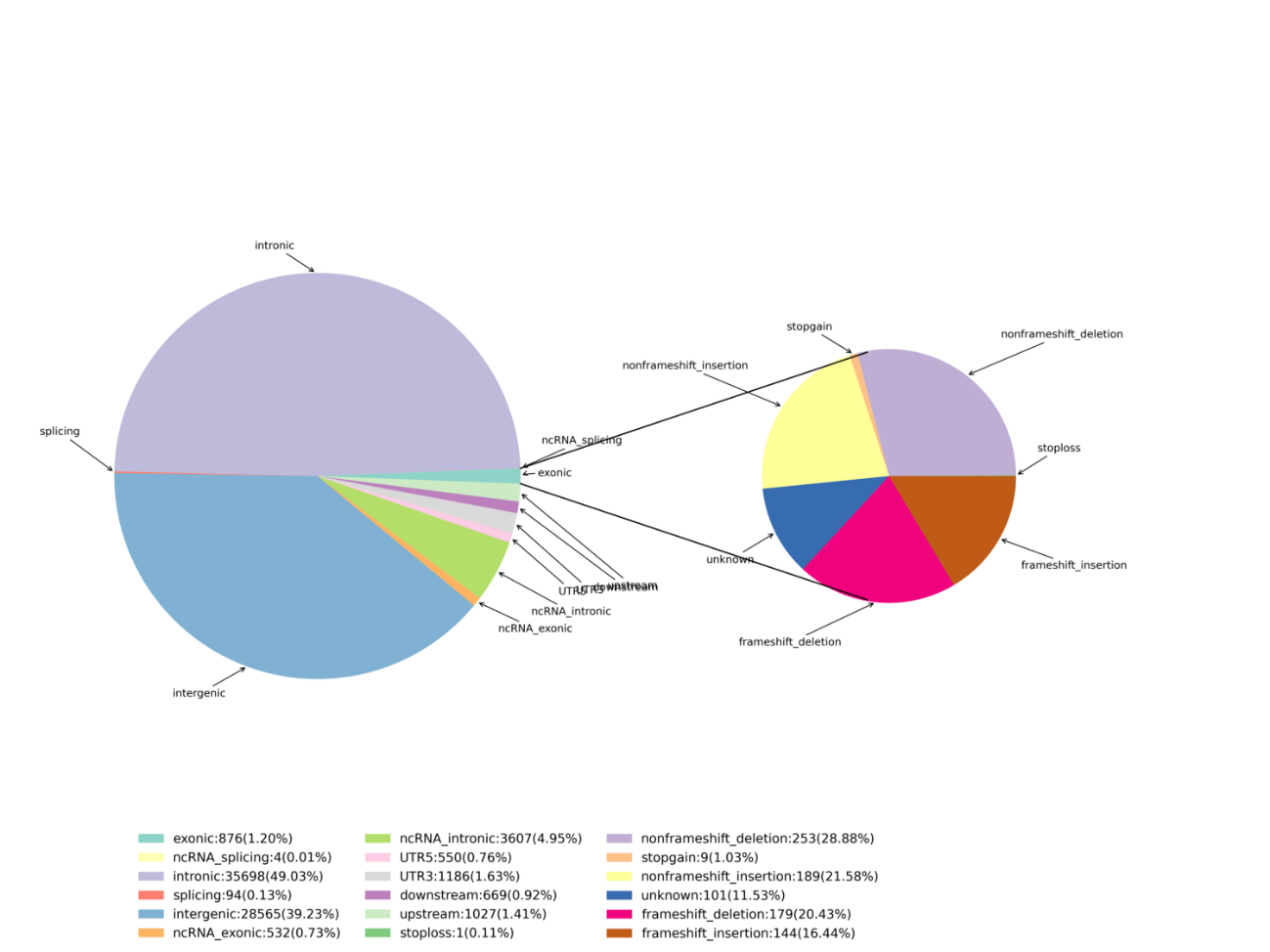
Análise avanzada: Identificación e distribución de SNPs/Indels nocivos - Circos Plot
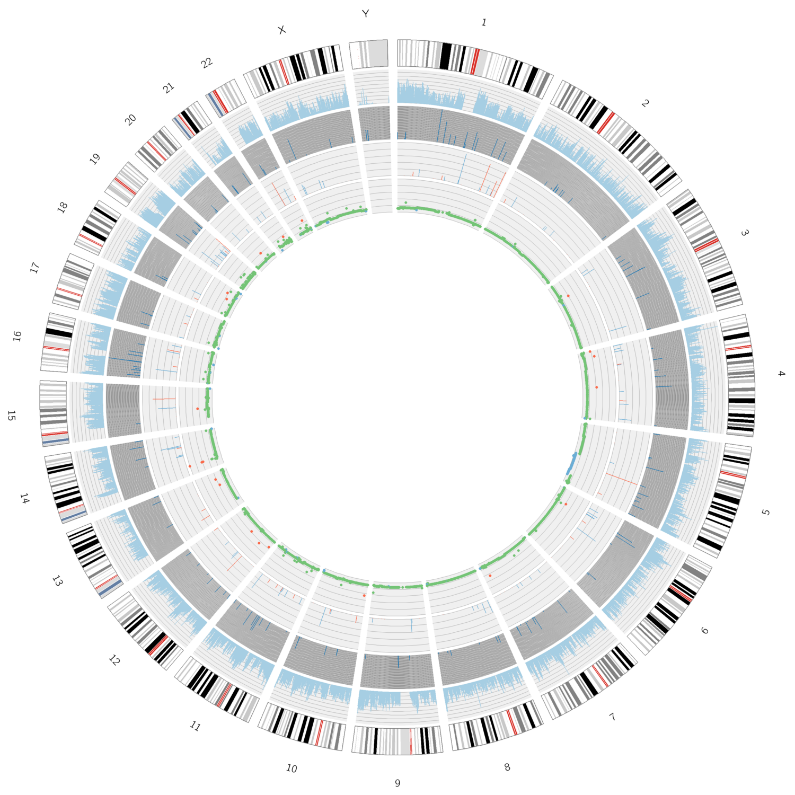
Análise do tumor: identificación e distribución de mutacións somáticas - trama de circos
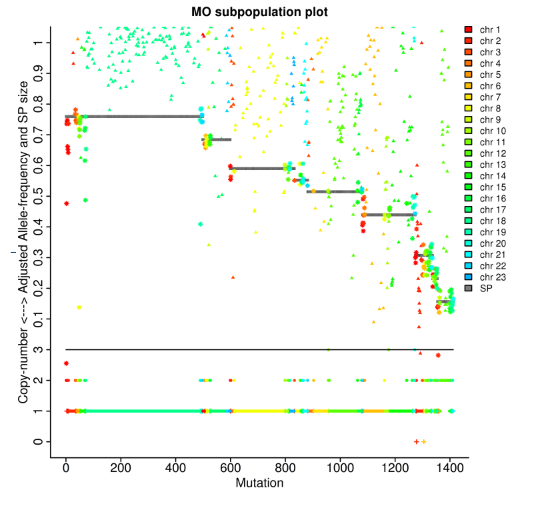
Análise do tumor: liñaxes clonais