Montaxe de xenoma baseado en HI-C
Características do servizo
● Secuenciación en Illumina Novaseq con PE150.
● O servizo require mostras de tecido, en lugar de ácidos nucleicos extraídos, para enlace reticulado con formaldehído e conserva as interaccións ADN-proteínas.
● O experimento Hi-C implica restrición e reparación final dos extremos pegajosos con biotina, seguido da circularización dos extremos contundentes resultantes conservando as interaccións. O ADN é entón tirado con perlas de estreptavidina e purificado para a preparación da biblioteca posterior.
Vantaxes do servizo

Visión xeral de Hi-C
(Lieberman-Aiden E et al.,Ciencia, 2009)
●Eliminando a necesidade de datos xenéticos da poboación:Hi-C substitúe a información esencial necesaria para a anclaxe de contig.
●Alta densidade de marcadores:dando lugar a unha alta relación de anclaxe de contigación superior ao 90%.
●Extensos coñecementos e rexistros de publicación:BMKGene ten unha ampla experiencia con máis de 2000 casos de montaxe do xenoma de Hi-C de 1000 especies diferentes e diversas patentes. Máis de 200 casos publicados teñen un factor de impacto acumulativo de máis de 2000.
●Equipo de bioinformática altamente cualificado:Con patentes internas e dereitos de autor de software para experimentos de Hi-C e análise de datos, o software de datos de visualización autodenvolvido permite que o bloque manual se mova, reverteran, revogan e refaceran.
●Soporte post-venda:O noso compromiso esténdese máis alá da finalización do proxecto cun período de servizo posterior a 3 meses. Durante este tempo, ofrecemos un seguimento do proxecto, asistencia de solución de problemas e sesións de Q&A para abordar as consultas relacionadas cos resultados.
●Anotación completa: Empregamos varias bases de datos para anotar funcionalmente os xenes con variacións identificadas e realizar a correspondente análise de enriquecemento, proporcionando información sobre múltiples proxectos de investigación.
Especificacións de servizo
| Preparación da biblioteca | Estratexia de secuenciación | Saída de datos recomendada | Control de calidade |
| Biblioteca Hi-C | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Requisitos da mostra
| Tecido | Cantidade requirida |
| Vísceras animais | ≥ 2 g |
| Músculo animal | |
| Sangue de mamíferos | ≥ 2 ml |
| Avicultura/sangue de peixe | |
| Planta- folla fresca | ≥ 3 g |
| Células cultivadas | ≥ 1x107 |
| Insecto | ≥ 2 g |
Fluxo de traballo de servizo

Deseño de experimentos

Entrega de mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos despois da venda
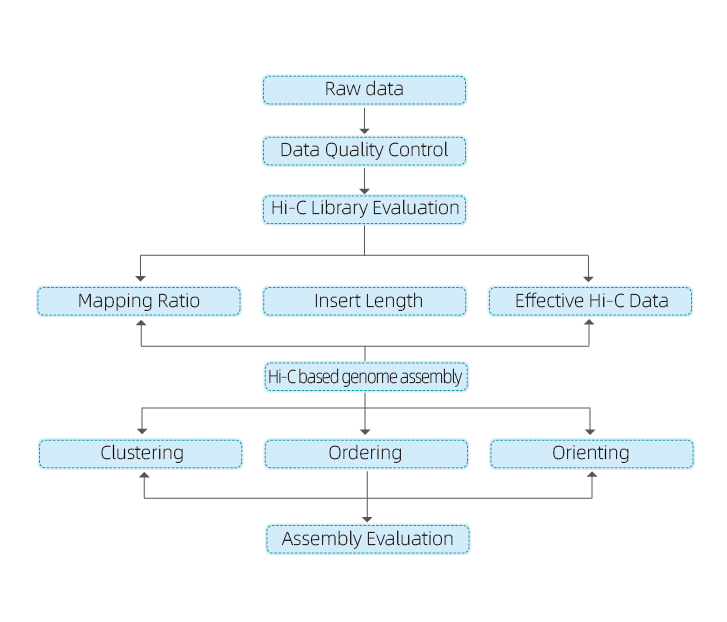
1) Datos brutos QC
2) Biblioteca Hi-C QC: Estimación de interaccións Hi-C válidas
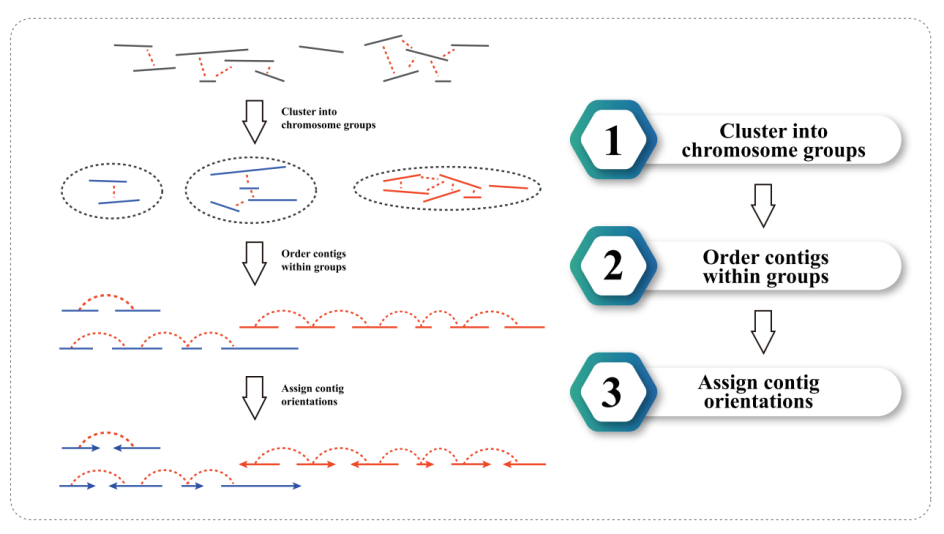
3) Montaxe Hi-C: agrupación de contigs en grupos, seguida da orde de contig dentro de cada grupo e asignando orientación contig
4) Avaliación Hi-C
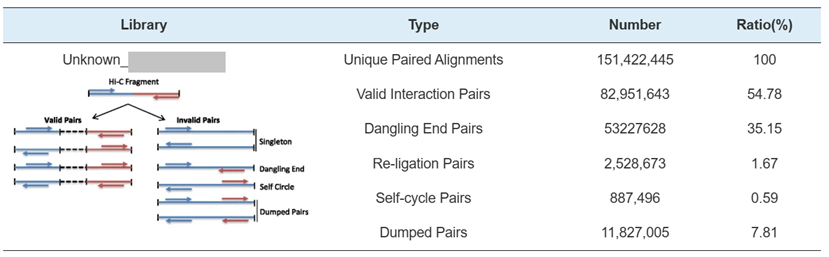
Biblioteca Hi-C QC-Estimación de pares de interacción válidos de Hi-C
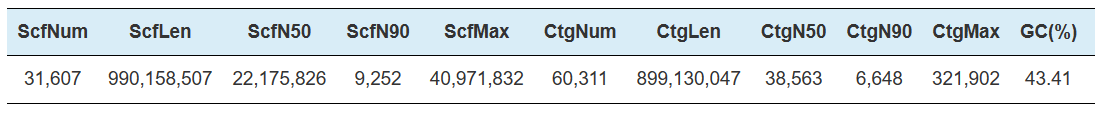
Asemblea HI-C-Estatísticas
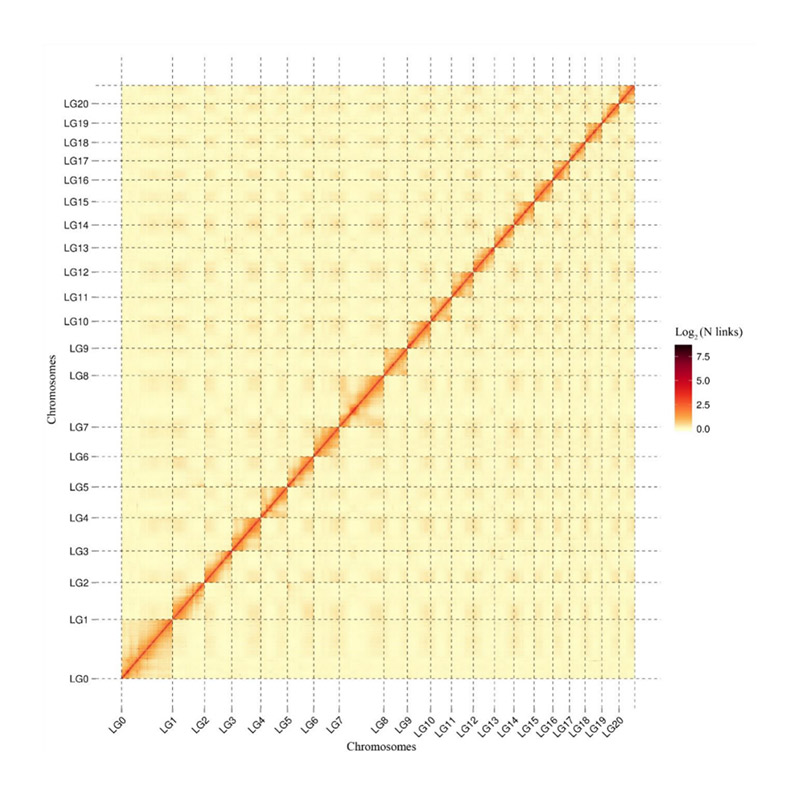
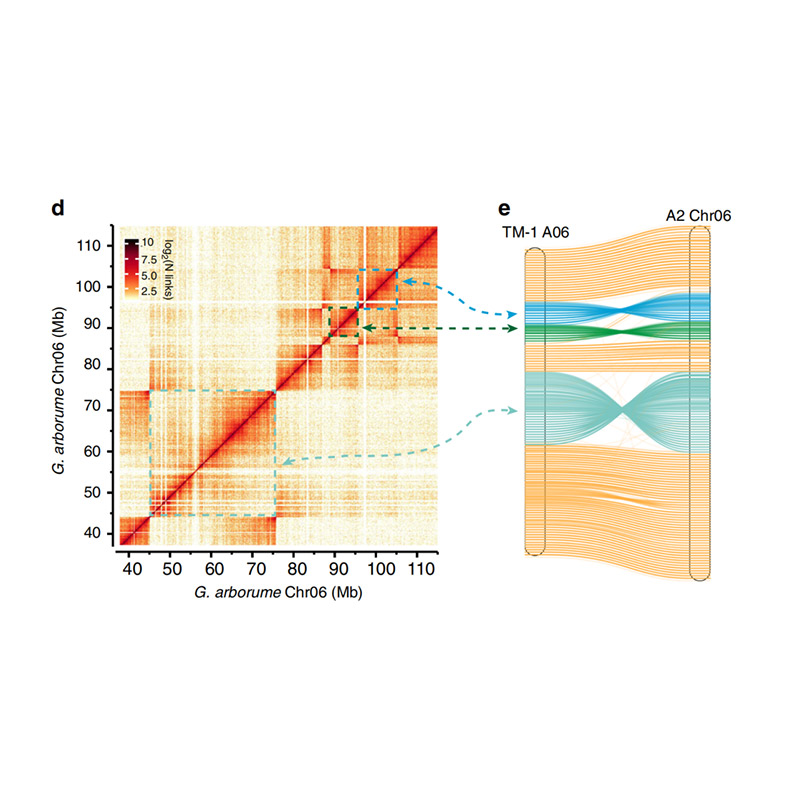
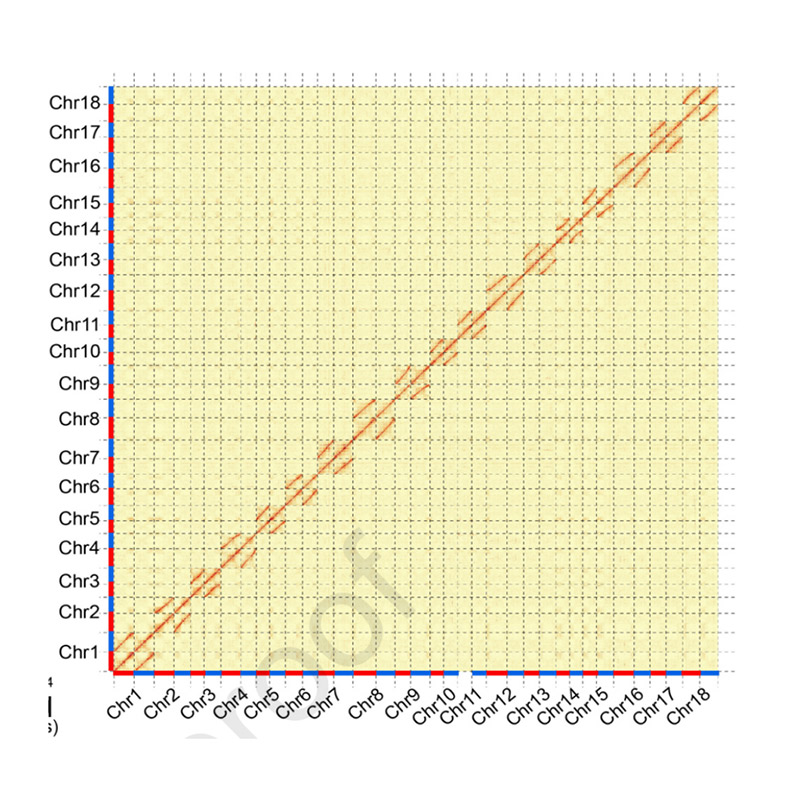
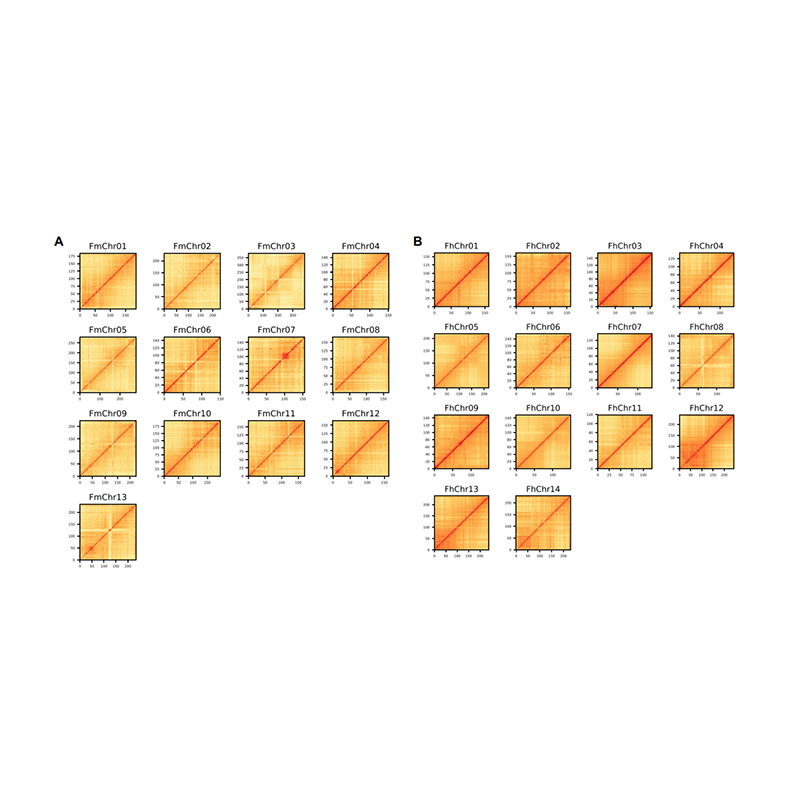
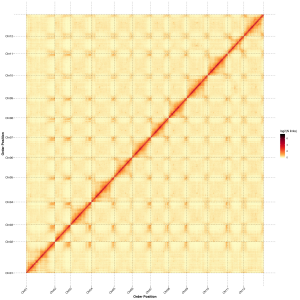
Avaliación post-ensamblaxe: mapa de calor da intensidade do sinal entre papeleiras
Explora os avances facilitados polos servizos de montaxe Hi-C de BMKGene a través dunha colección curada de publicacións.
Tian, T. et al. (2023) "Montaxe do xenoma e disección xenética dun destacado xermoplasma de millo resistente á seca", Nature Genetics 2023 55: 3, 55 (3), pp. 496-506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) "A Asemblea a escala cromosoma do xenoma de Apis Cerana de abella asiática", Frontiers in Genetics, 11, p. 524140. Doi: 10.3389/fgene.2020.00279/bibtex.
Zhang, F. et al. (2023) "Evolución reveladora da biosíntese alcaloide de Tropane analizando dous xenomas na familia Solanaceae", Nature Communications 2023 14: 1, 14 (1), pp. 1-18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) "Os xenomas da árbore banyan e a avispa polinizadora proporcionan información sobre a coevolución da fig-wasp", Cell, 183 (4), pp. 875-889.E17. doi: 10.1016/j.cell.2020.09.043