Interacción con cromatina baseada en HI-C
Características do servizo
● Secuenciación en Illumina Novaseq con PE150.
● O servizo require mostras de tecido, en lugar de ácidos nucleicos extraídos, para enlace reticulado con formaldehído e conserva as interaccións ADN-proteínas.
● O experimento Hi-C implica restrición e reparación final dos extremos pegajosos con biotina, seguido da circularización dos extremos contundentes resultantes conservando as interaccións. O ADN é entón tirado con perlas de estreptavidina e purificado para a preparación da biblioteca posterior.
Vantaxes do servizo
●Deseño de enzimas de restrición óptima: Para garantir unha alta eficiencia Hi-C en diferentes especies con pares de interacción válidos ata un 93%.
●Extensos coñecementos e rexistros de publicación:BMKGene ten unha ampla experiencia con> 2000 proxectos de secuenciación de Hi-C de 800 especies diferentes e diversas patentes. Máis de 100 casos publicados cun factor de impacto acumulativo de máis de 900.
●Equipo de bioinformática altamente cualificado:con patentes internos e dereitos de autor de software para experimentos de Hi-C e análise de datos e un software de datos de visualización autodenvolvido.
●Soporte post-venda:O noso compromiso esténdese máis alá da finalización do proxecto cun período de servizo posterior a 3 meses. Durante este tempo, ofrecemos un seguimento do proxecto, asistencia de solución de problemas e sesións de Q&A para abordar as consultas relacionadas cos resultados.
●Anotación completa: Empregamos varias bases de datos para anotar funcionalmente os xenes con variacións identificadas e realizar a correspondente análise de enriquecemento, proporcionando información sobre múltiples proxectos de investigación.
Especificacións de servizo
| Biblioteca | Estratexia de secuenciación | Saída de datos recomendada | Resolución de sinal HI-C |
| Biblioteca Hi-C | Illumina PE150 | Loop de cromatina: 150x Tad: 50x | Loop de cromatina: 10kb Tad: 40kb |
Requisitos de servizo
| Tipo de mostra | Cantidade requirida |
| Tecido animal | ≥2g |
| Sangue enteiro | ≥2ml |
| Fungos | ≥1g |
| Planta- tecido novo | 1g/alícuota, 2-4 aliquotas recomendadas |
| Células cultivadas | ≥1x107 |
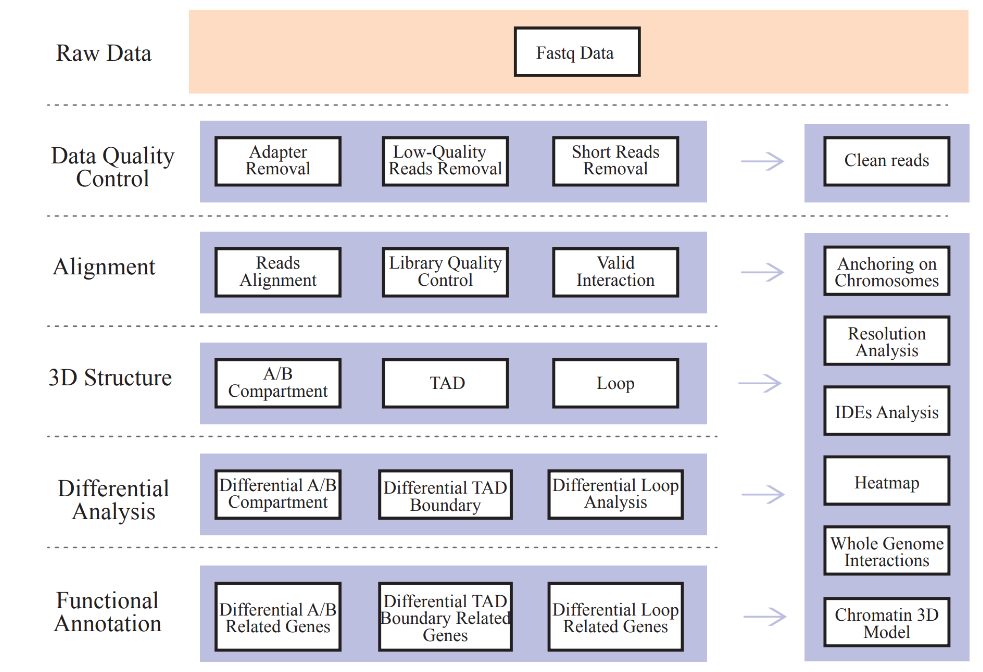
Inclúe a seguinte análise:
● Datos brutos QC;
● Mapping and Hi-C Library QC: pares de interacción válidos e interacción Exponentes de decadencia (IDES);
● Perfil de interacción en todo o xenoma: análise cis/trans e mapa de interacción Hi-C;
● Análise da distribución do compartimento A/B;
● Identificación de TADs e bucles de cromatina;
● Análise diferencial sobre elementos de estrutura de cromatina 3D entre mostras e anotación funcional correspondente de xenes asociados.
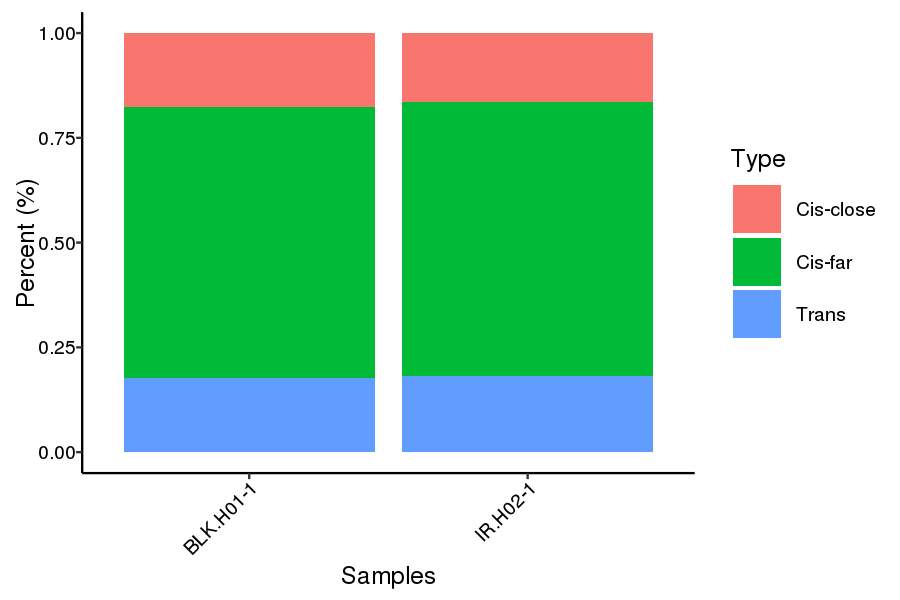
Distribución de proporcións cis e trans
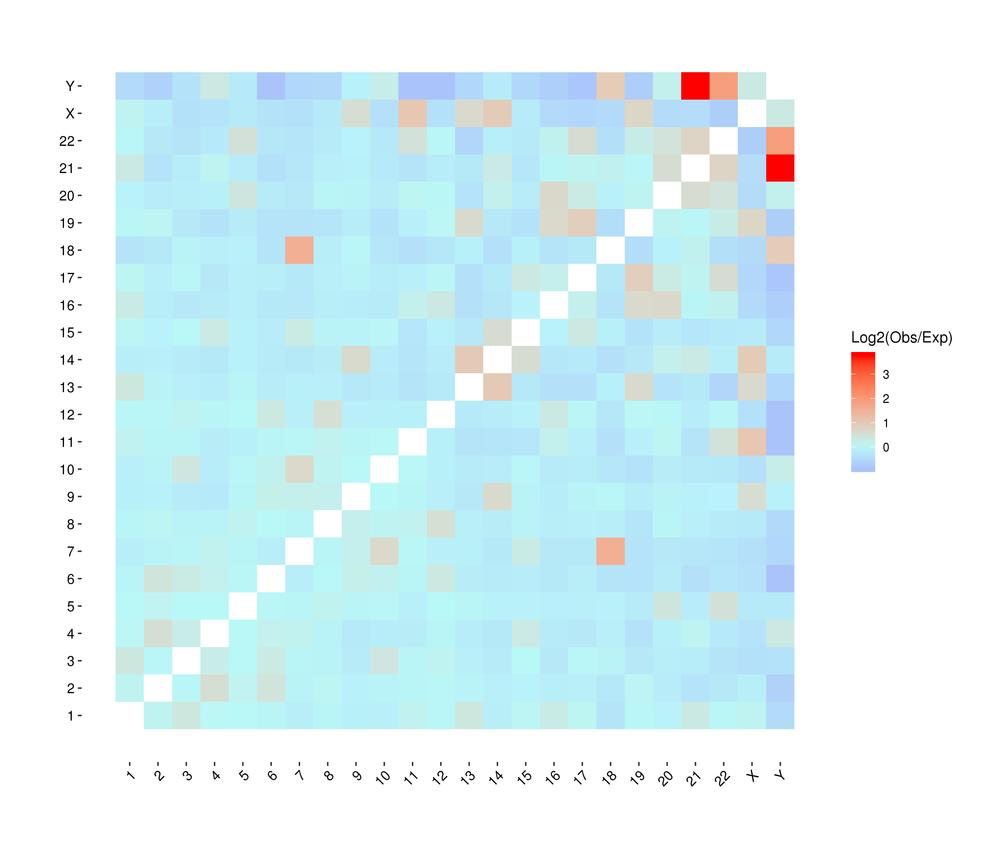
Mapa de calor das interaccións cromosómicas entre mostras
Distribución en todo o xenoma de compartimentos A/B
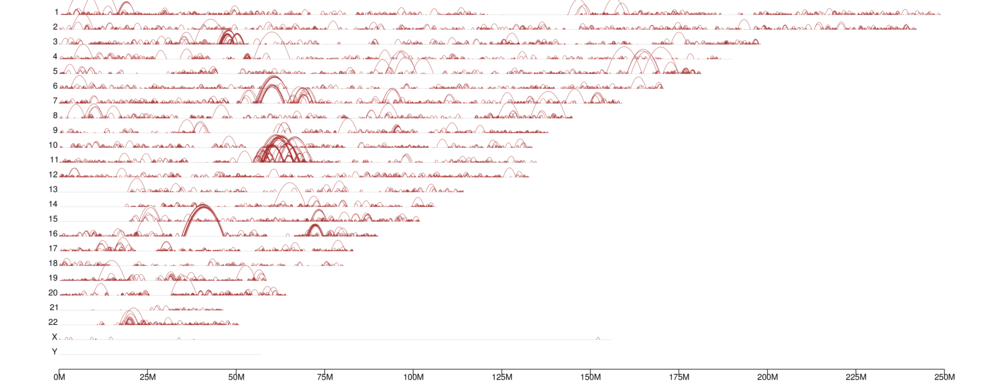
Distribución en todo o xenoma de bucles de cromatina
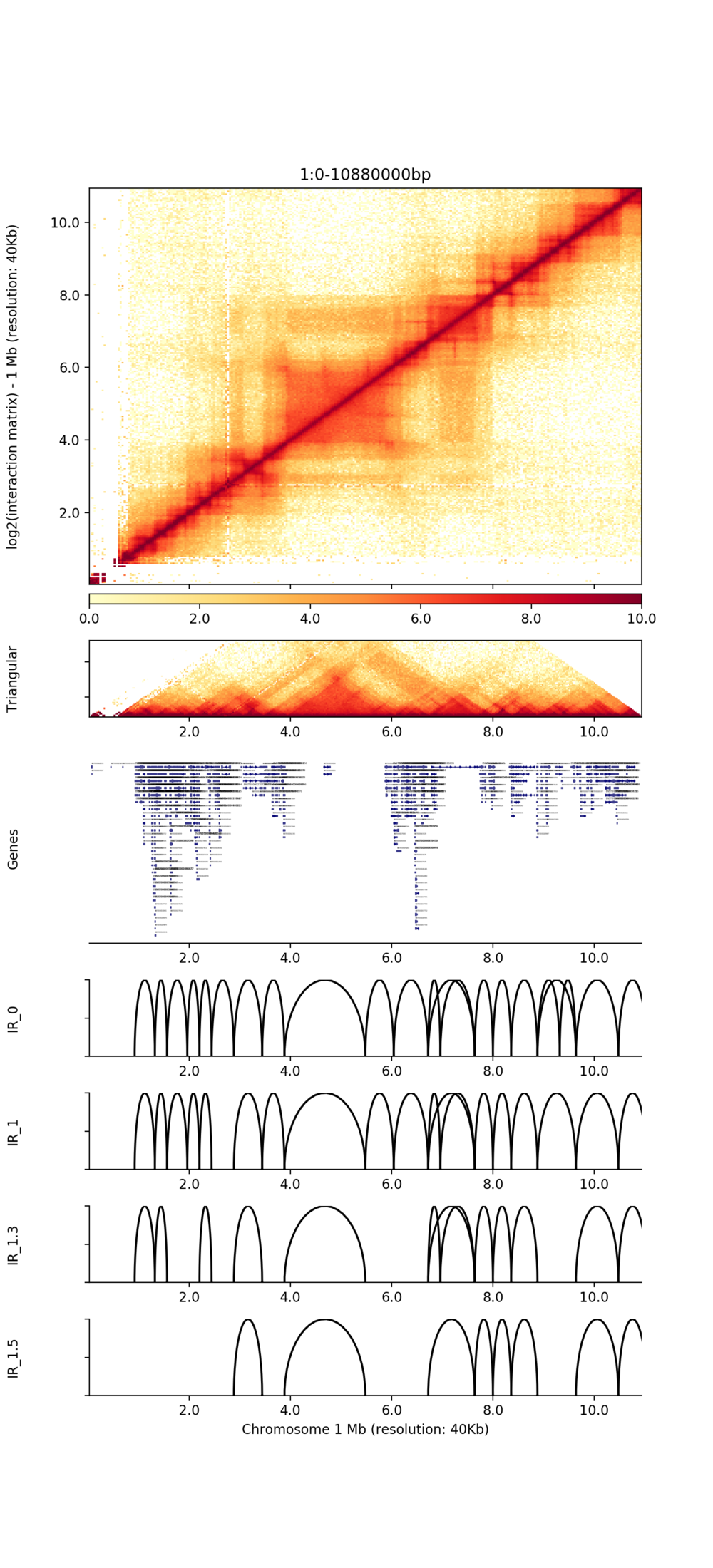
Visualización de TADS
Explora os avances da investigación facilitados polos servizos de secuenciación Hi-C de BMKGene a través dunha colección curada de publicacións.
Meng, T. et al. (2021) "Unha análise integrada de multiomics integrada identifica a CA2 como un obxectivo novo para o acordoma",Neuro-oncoloxía, 23 (10), pp. 1709-1722. doi: 10.1093/neuonc/noab156.
Xu, L. et al. (2021) "Desorganización e reordenación do xenoma 3D proporcionan información sobre a patoxénese do NAFLD por Hi-C integrada, nanopor e secuenciación de ARN ',Acta Pharmaceutica Sinica B, 11 (10), pp. 3150–3164. doi: 10.1016/j.apsb.2021.03.022.