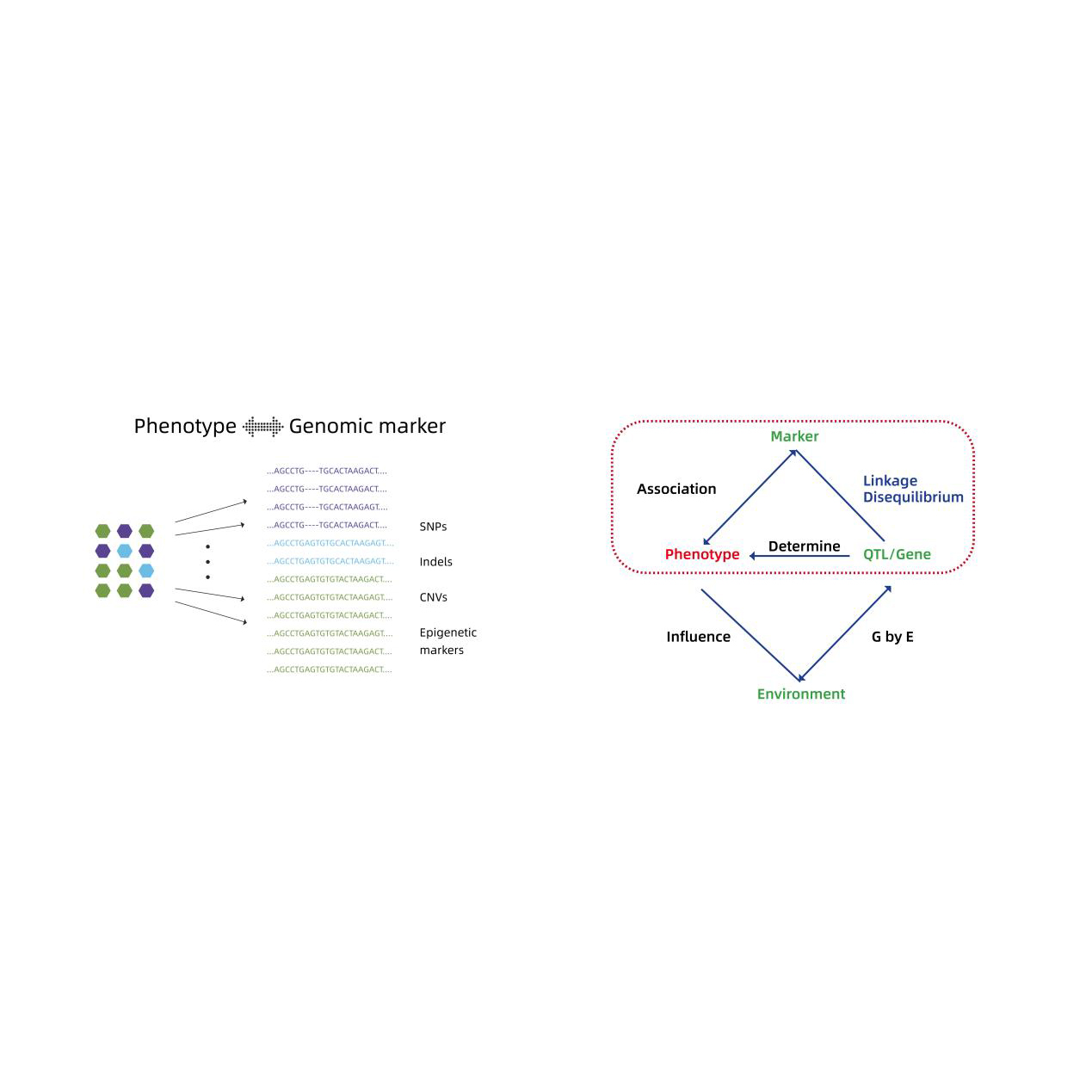
Análise de asociacións en todo o xenoma
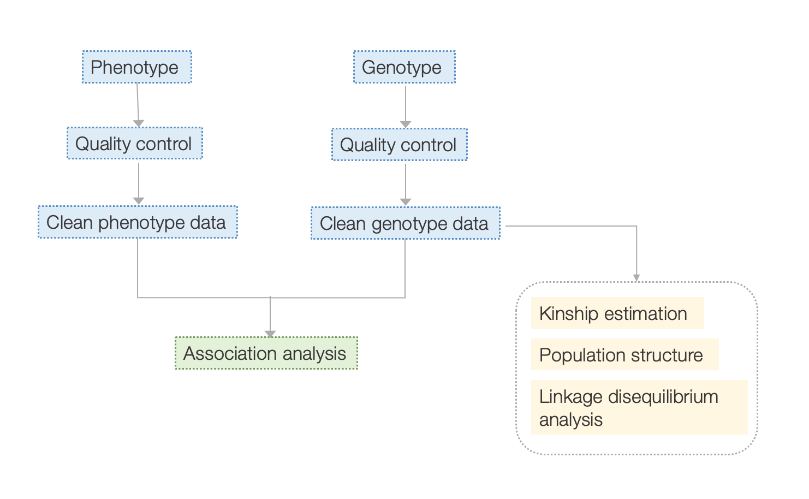
Fluxo de traballo

Vantaxes do servizo
●Extensos coñecementos e rexistros de publicación: Con experiencia acumulada en GWAS, BMKGene completou centos de proxectos de especies na investigación de GWAS de poboación, axudou aos investigadores a publicar máis de 100 artigos e o factor acumulativo de impacto alcanzou os 500.
● Análise bioinformática completa: O fluxo de traballo inclúe a análise da Asociación SNP-Trait, a entrega dun conxunto de xenes candidatos e a súa correspondente anotación funcional.
●Equipo de bioinformática altamente cualificado e ciclo de análise curta: Con gran experiencia na análise avanzada de xenómica, o equipo de BMKGene ofrece análises completas cun tempo rápido.
●Soporte post-venda:O noso compromiso esténdese máis alá da finalización do proxecto cun período de servizo posterior a 3 meses. Durante este tempo, ofrecemos un seguimento do proxecto, asistencia de solución de problemas e sesións de Q&A para abordar as consultas relacionadas cos resultados.
Especificacións e requisitos do servizo
| Tipo de secuenciación | Escala de poboación recomendada | Estratexia de secuenciación | Requisitos de nucleótidos |
| Secuenciación de xenomas enteiros | 200 mostras | 10x | Concentración: ≥ 1 ng/ µl Cantidade total≥ 30ng Degradación ou contaminación limitada ou sen |
| Fragmento amplificado do-locus específico (SLAF) | Profundidade de etiqueta: 10x Número de etiquetas: <400 MB: recoméndase WGS <1 GB: etiquetas 100k 1 GB > 2 GB: etiquetas de 300k Etiquetas máximas de 500k | Concentración ≥ 5 ng/µl Cantidade total ≥ 80 ng Nanodrop OD260/280 = 1,6-2,5 Xel de agarosa: non degradación ou contaminación limitada
|
Selección de materiais



Diferentes variedades, subespecies, landraces/genebanks/familias mixtas/recursos salvaxes
Diferentes variedades, subespecies, terras
Familia de medio sib/familia completa/recursos salvaxes
Fluxo de traballo de servizo

Deseño de experimentos

Entrega de mostra

Extracción de ARN

Construción da biblioteca

Secuenciación

Análise de datos

Servizos despois da venda
Inclúe a seguinte análise:
- Análise de asociacións en todo o xenoma: LM, LMM, Emmax, Modelo FastLMM
- Anotación funcional de xenes candidatos
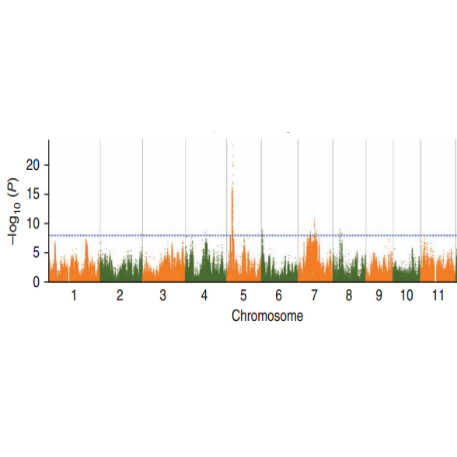
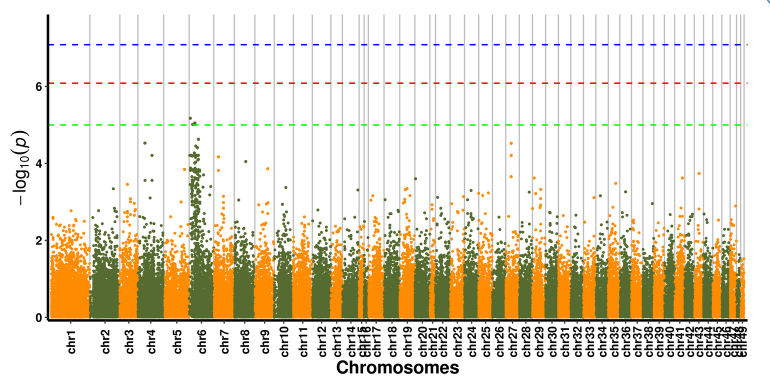
Análise da Asociación SNP-Trait-Manhattan Plot
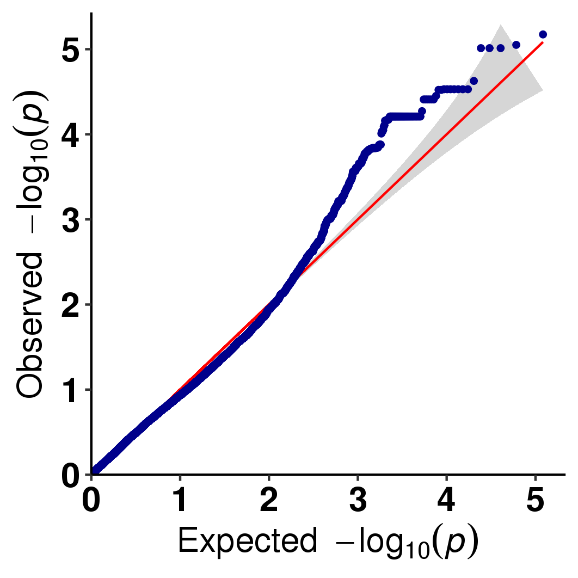
Análise da Asociación SNP-Trait-Plot QQ
Explora os avances facilitados polos servizos de GWAS de BMKGene a través dunha colección curada de publicacións:
LV, L. et al. ")Acuicultura, 569, p. 739351. Doi: 10.1016/j.aquaculture.2023.739351.
Li, X. et al. (2022) 'As análises multi-Tomics de 398 accesións de millo de foxtail revelan rexións xenómicas asociadas á domesticación, trazos de metabolitos e efectos antiinflamatorios ",Planta molecular, 15 (8), pp. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) "mapeo de asociacións en todo o xenoma de hulless apenas fenotipos no ambiente de seca",Fronteiras na ciencia das plantas, 13, p. 924892. Doi: 10.3389/fpls.2022.924892/bibtex.
Zhao, X. et al. (2021) 'GMST1, que codifica unha sulfotransferase, confire resistencia ás cepas de virus de mosaico de soia G2 e G3',Planta, células e ambiente, 44 (8), pp. 2777–2792. doi: 10.1111/pce.14066.