Secuenciación de ARNm de lonxitude completa
Características
● Captura de ARNm poli-A seguido de síntese de ADNc e preparación da biblioteca
● Secuenciación das transcricións de lonxitude completa
● Análise bioinformática baseada no aliñamento dun xenoma de referencia
● A análise bioinformática inclúe non só a expresión a nivel de xenes e isoforma senón tamén análise de lncRNA, fusións xénicas, poli-adenilación e estrutura xénica
Vantaxes do servizo
●Cuantificación da expresión a nivel de isoforma: habilitando unha análise de expresión detallada e precisa, revelando un cambio que se pode enmascarar ao analizar toda a expresión xénica
●Demandas de datos reducidas:En comparación coa secuenciación de última xeración (NGS), a secuenciación de nanopore presenta requisitos de datos máis baixos, permitindo niveis equivalentes de saturación de cuantificación da expresión xénica con datos máis pequenos.
●Maior precisión da cuantificación da expresión: tanto a nivel de xenes como isoforma
●Identificación de información transcriptómica adicional: Poliadenilación alternativa, xenes de fusión e LCNRNA e os seus xenes de destino
●Ampla experiencia: O noso equipo achega unha ampla experiencia a todos os proxectos, completando máis de 850 proxectos de transcriptoma de lonxitude completa e procesaron máis de 8.000 mostras.
●Soporte post-venda: O noso compromiso esténdese máis alá da finalización do proxecto cun período de servizo posterior a 3 meses. Durante este tempo, ofrecemos un seguimento do proxecto, asistencia de solución de problemas e sesións de Q&A para abordar as consultas relacionadas cos resultados.
Requisitos de mostra e entrega
| Biblioteca | Estratexia de secuenciación | Datos recomendados | Control de calidade |
| Poly a enriquecido | Illumina PE150 | 6/12 GB | Puntuación media de calidade: Q10 |
Requisitos da mostra:
Nucleótidos:
| Conc. (Ng/µl) | Cantidade (μg) | Pureza | Integridade |
| ≥ 100 | ≥ 1,0 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Contaminación limitada ou sen proteínas ou ADN mostrada no xel. | Para plantas: rin≥7,0; Para animais: rin≥7,5; 5.0≥28s/18s≥1,0; elevación de base limitada ou sen base |
● Plantas:
Raíz, talo ou pétalo: 450 mg
Folla ou semente: 300 mg
Froito: 1,2 g
● Animal:
Corazón ou intestino: 300 mg
Vísceras ou cerebro: 240 mg
Músculo: 450 mg
Ósos, pelo ou pel: 1g
● Artrópodos:
Insectos: 6g
Crustacea: 300 mg
● sangue enteiro: 1 tubo
● Células: 106 células
Entrega de mostra recomendada
Recipiente: tubo de centrifuga de 2 ml (non se recomenda a folla de lata)
Etiquetaxe de mostra: grupo+replicar por exemplo A1, A2, A3; B1, B2, B3.
Envío:
1. Ice seco: as mostras deben ser embaladas en bolsas e enteradas en xeo seco.
2. Tubos RNastable: As mostras de ARN pódense secar no tubo de estabilización do ARN (por exemplo, RNastable®) e enviadas a temperatura ambiente.
Fluxo de traballo de servizo
Nucleótidos:

Entrega de mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos despois da venda
Fluxo de traballo de servizo
Tecido:

Deseño de experimentos

Entrega de mostra

Extracción de ARN

Construción da biblioteca

Secuenciación

Análise de datos

Servizos despois da venda
● Procesamento de datos en bruto
● Identificación da transcrición
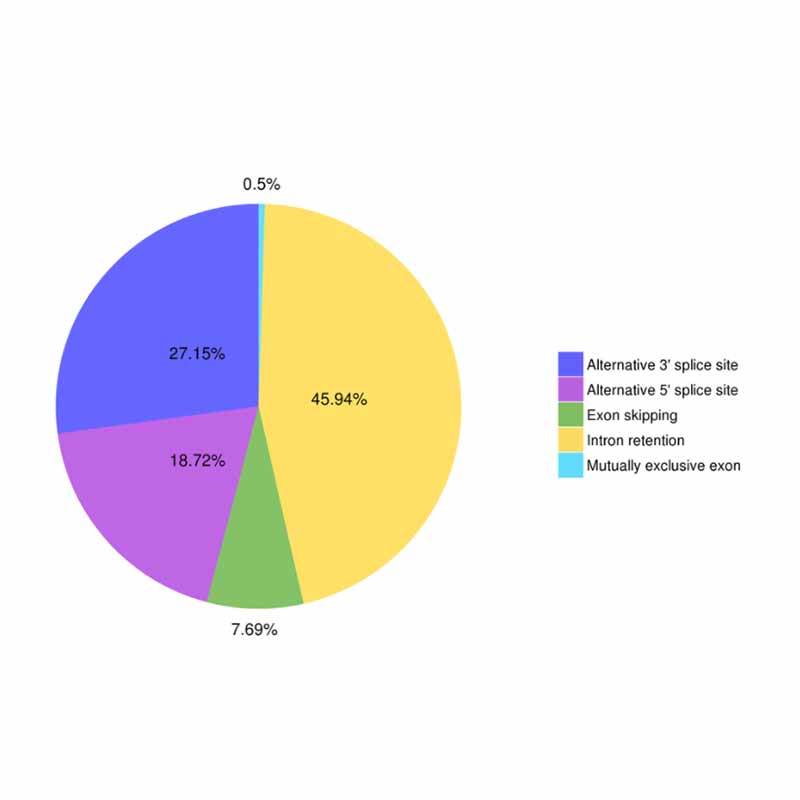
● Splicing alternativo
● Cuantificación da expresión no nivel de xenes e no nivel de isoforma
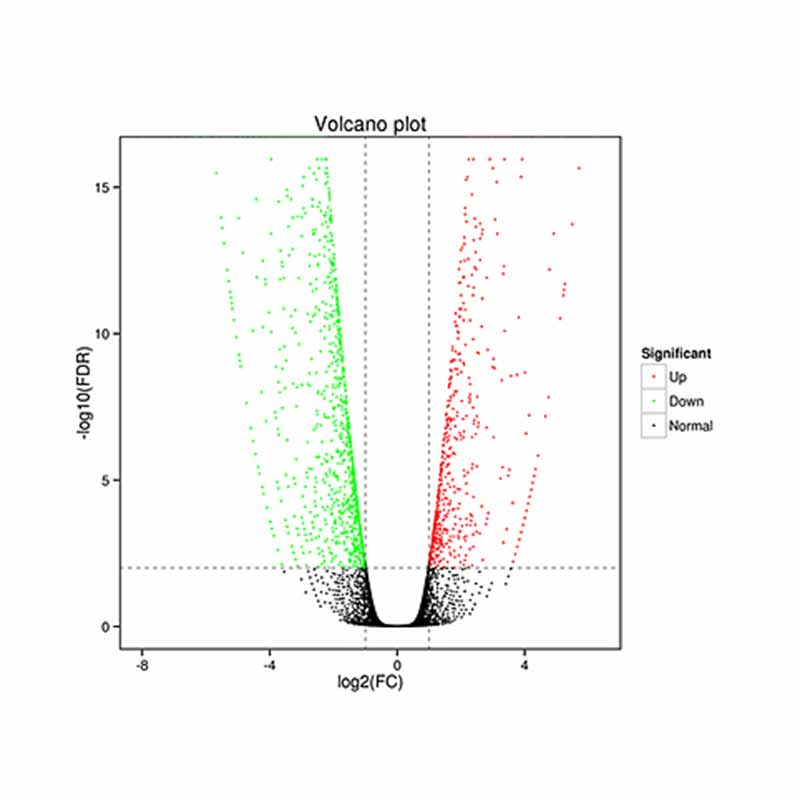
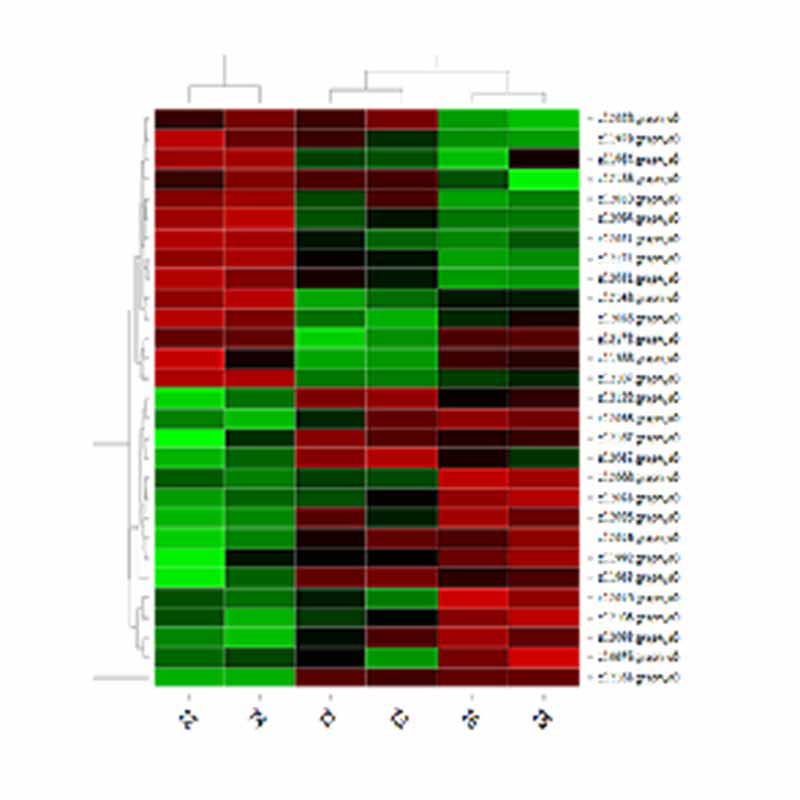
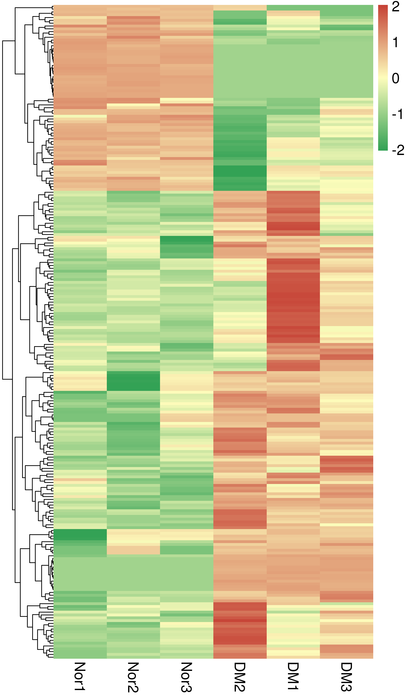
● Análise de expresión diferencial
● Anotación e enriquecemento da función (DEGS e Det)
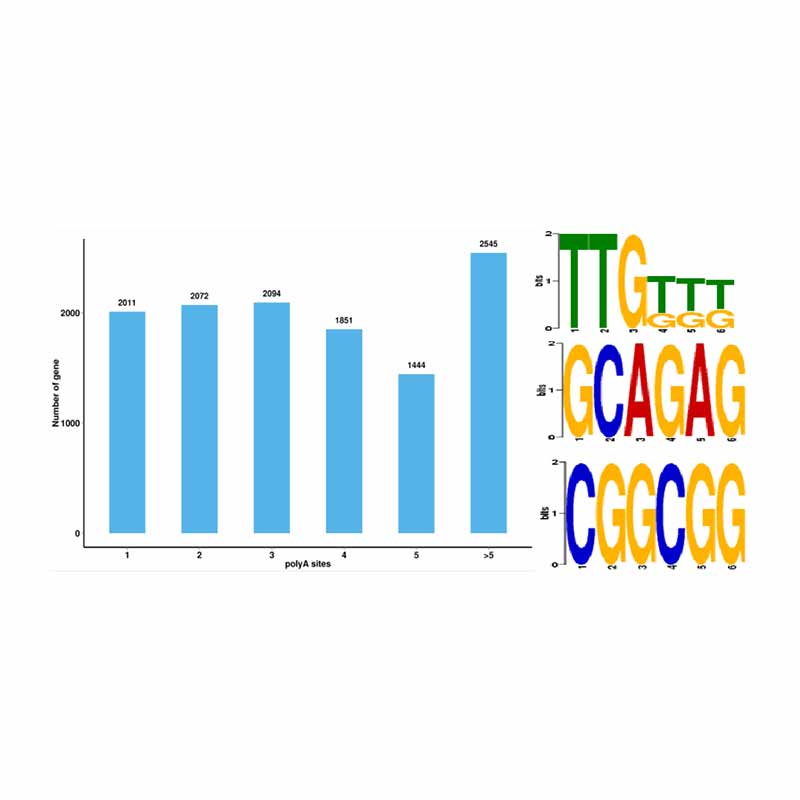
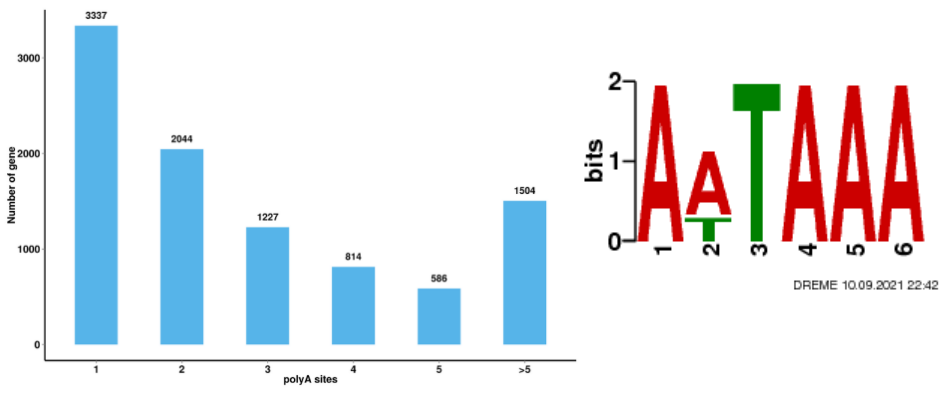
Análise alternativa de empalme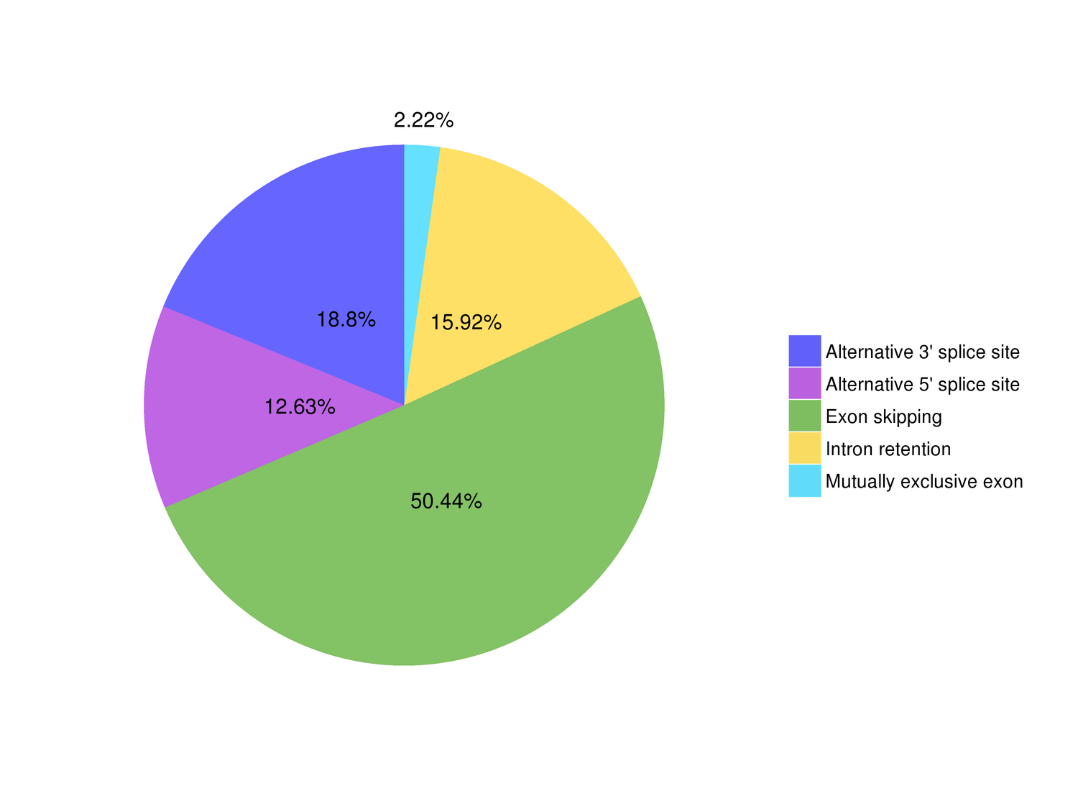 Análise alternativa de poliadenilación (APA)
Análise alternativa de poliadenilación (APA)
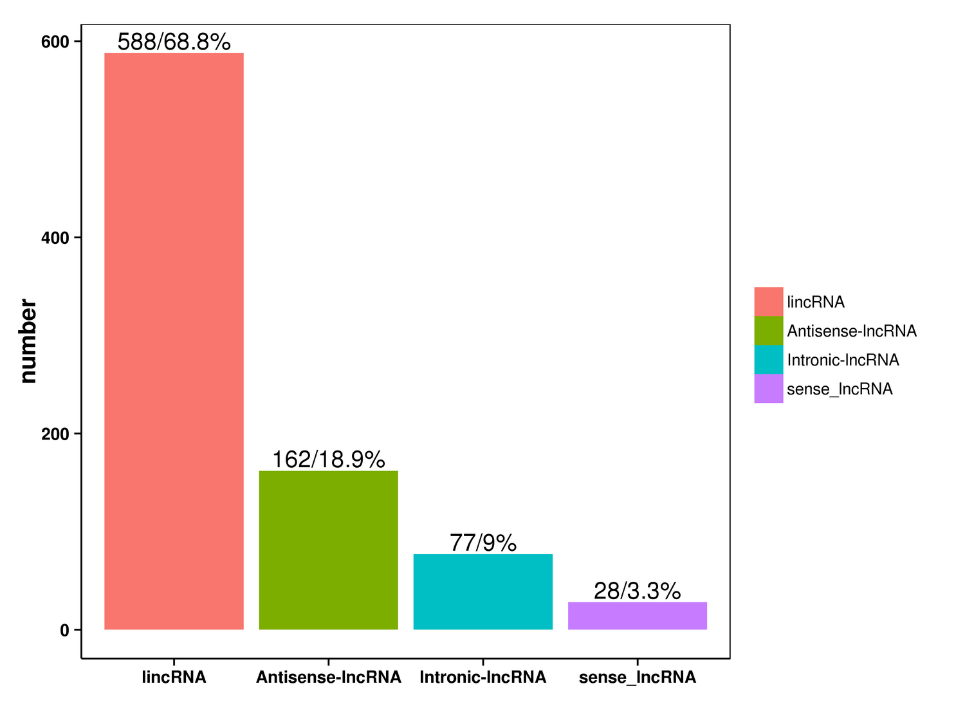
Predición de LNCRNA
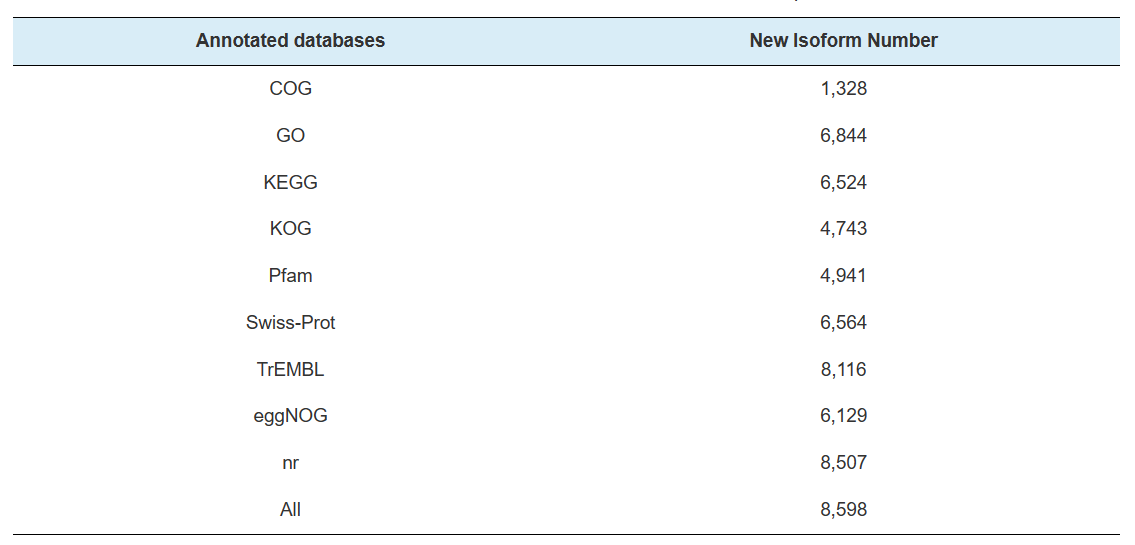
Anotación de xenes novidosos
Agrupación de dets
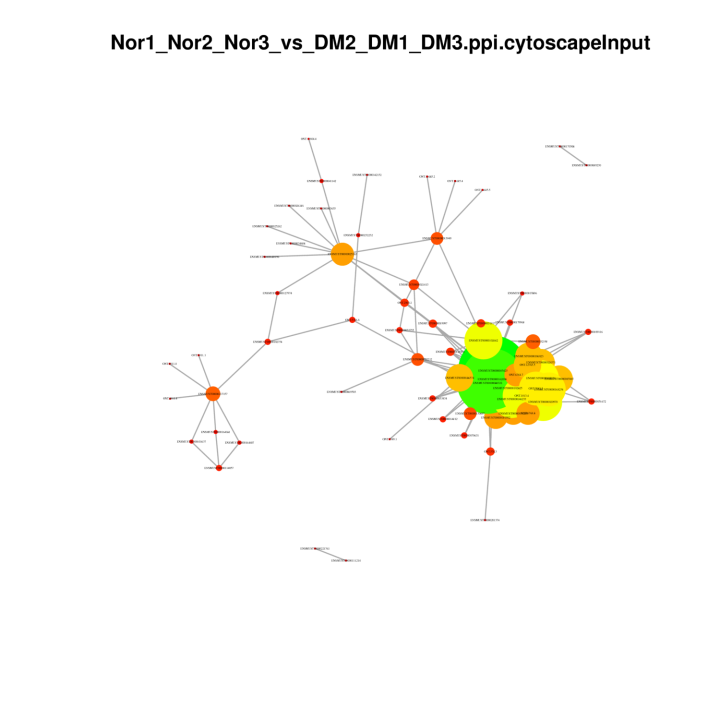
Redes de proteínas proteínas en DEG
Explora os avances facilitados polos servizos de secuenciación de ARNm de lonxitude completa de BMKGene a través dunha colección curada de publicacións.
Gong, B. et al. (2023) 'Activación epixenética e transcripcional da Secretara quinase FAM20C como oncóxeno no glioma', Journal of Genetics and Genomics, 50 (6), pp. 422-433. doi: 10.1016/j.jgg.2023.01.008.
El, Z. et al. (2023) 'A secuenciación de transcriptoma de lonxitude completa de linfocitos responde a IFN-γ revela unha resposta inmune de th1 en flounder (Paralichthys olivaceus)', Inmunoloxía de peixe e marisco, 134, p. 108636. Doi: 10.1016/j.fsi.2023.108636.
Ma, Y. et al. (2023) 'Análise comparativa de métodos de secuenciación de ARN PACBIO e ONT para identificación de veneno Nemopilema Nomurai', Genomics, 115 (6), p. 110709. DOI: 10.1016/j.ygeno.2023.110709.
Yu, D. et al. (2023) 'A análise nano-seq revela unha tendencia funcional diferente entre exosomas e microvesículas derivadas de Humsc', investigación e terapia de células nai, 14 (1), pp. 1-13. doi: 10.1186/s13287-023-03491-5/táboas/6.