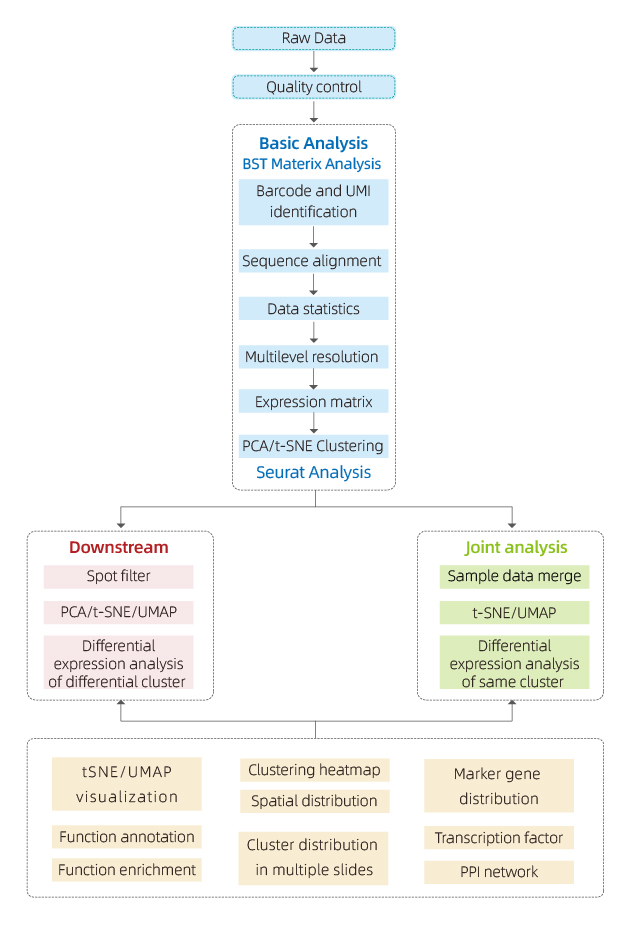
Transcriptoma espacial BMKMANU S1000
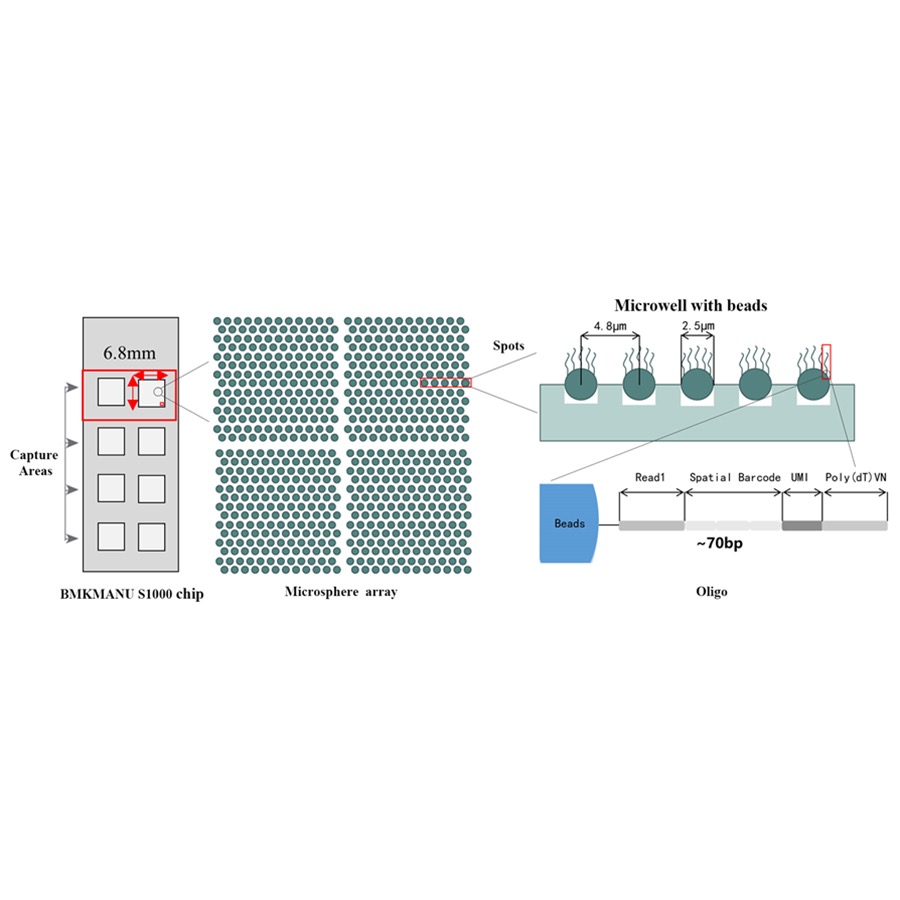
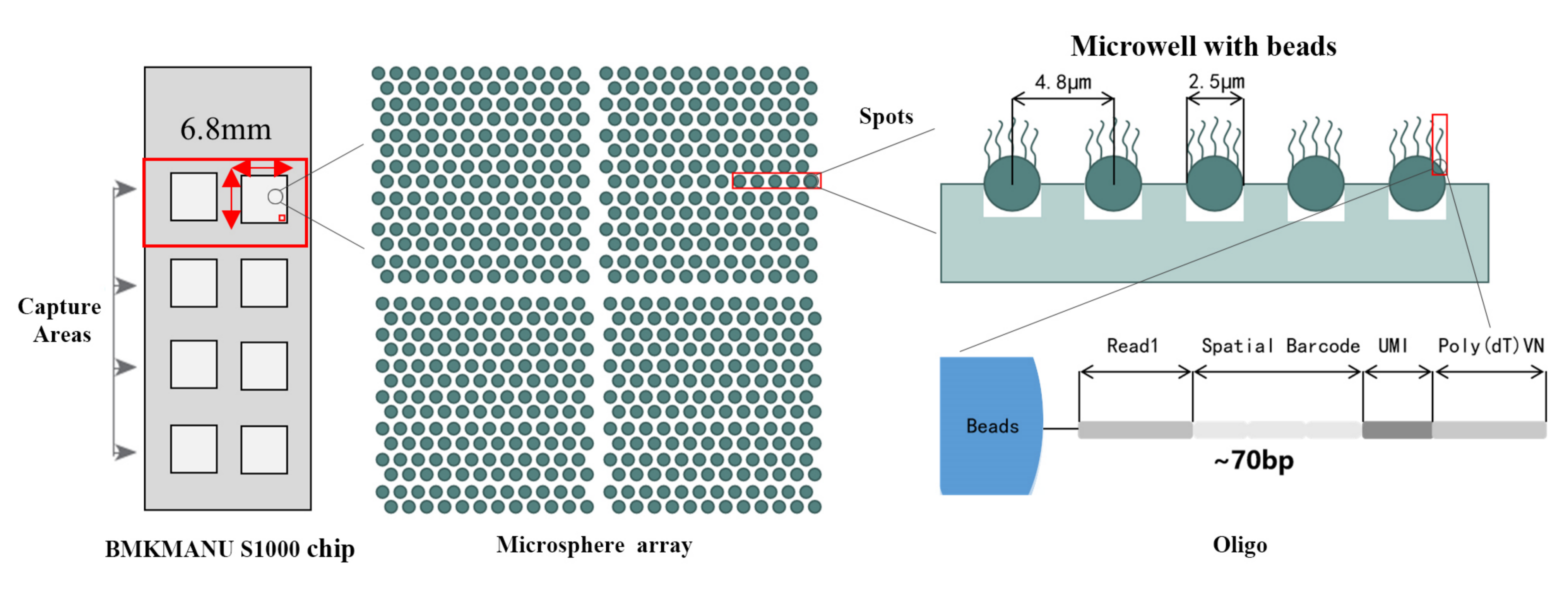
Esquema técnico do transcriptoma espacial BMKMANU S1000
Características
● Resolución: 5 µM
● Diámetro do punto: 2,5 µM
● Número de spots: aproximadamente 2 millóns
● 3 posibles formatos de área de captura: 6,8 mm * 6,8 mm, 11 mm * 11 mm ou 15 mm * 20 mm
● Cada perla con código de barras está cargada con imprimacións compostas por 4 seccións:
cola poli(dT) para cebado de ARNm e síntese de ADNc
Identificador molecular único (UMI) para corrixir o sesgo de amplificación
Código de barras espacial
Secuencia de unión do cebador de secuenciación de lectura parcial 1
● Tinción H&E e fluorescente de seccións
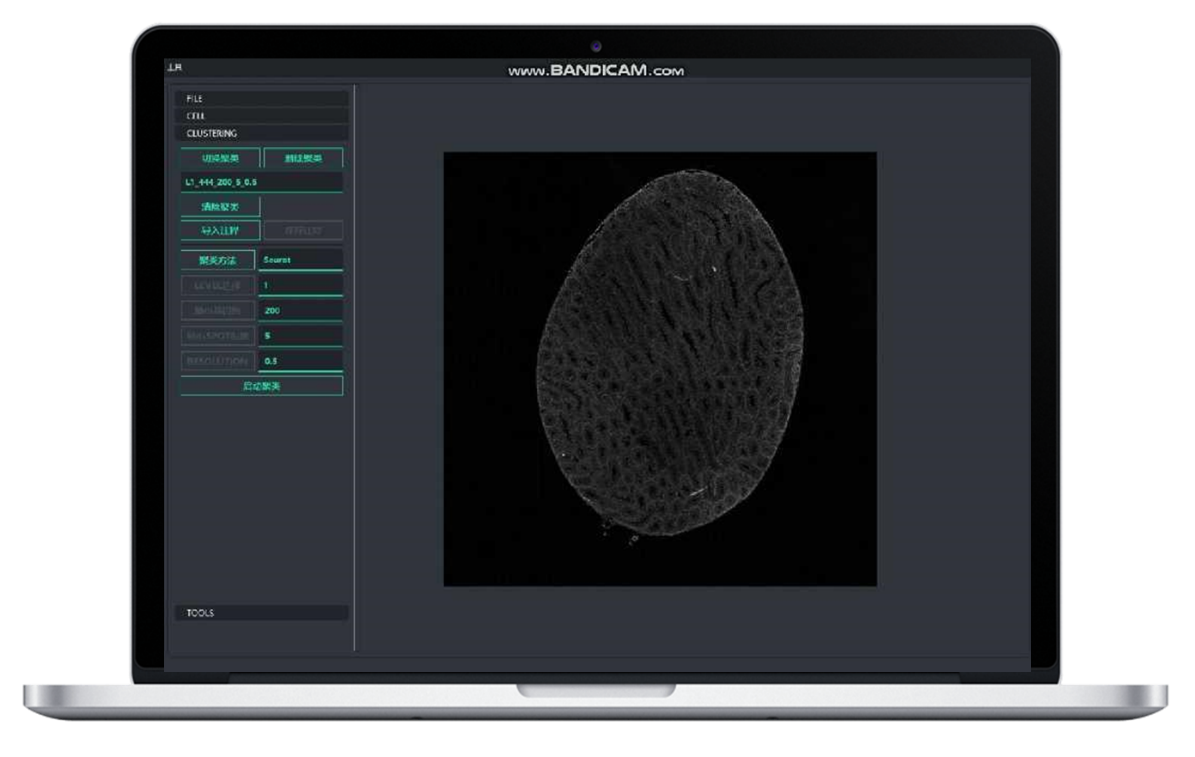
● Posibilidade de usotecnoloxía de segmentación celular: integración da tinción H&E, a tinción fluorescente e a secuenciación de ARN para determinar os límites de cada célula e asignar correctamente a expresión xénica a cada célula.
Vantaxes do BMKMANU S1000
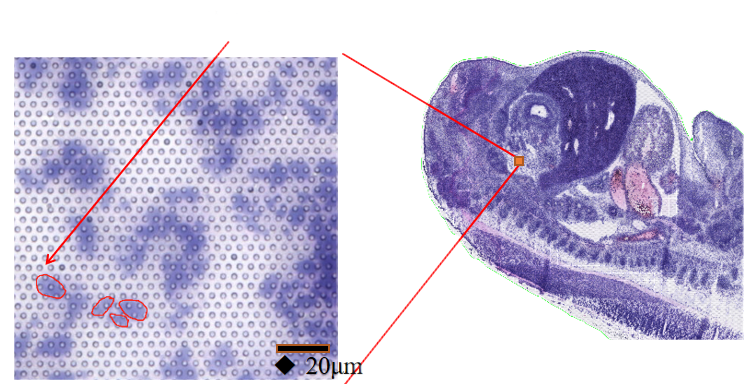
●Resolución subcelular: Cada área de captura contiña > 2 millóns de puntos espaciais con código de barras cun diámetro de 2,5 µm e un espazamento de 5 µm entre os centros de puntos, o que permitía a análise espacial do transcriptoma con resolución subcelular (5 µm).
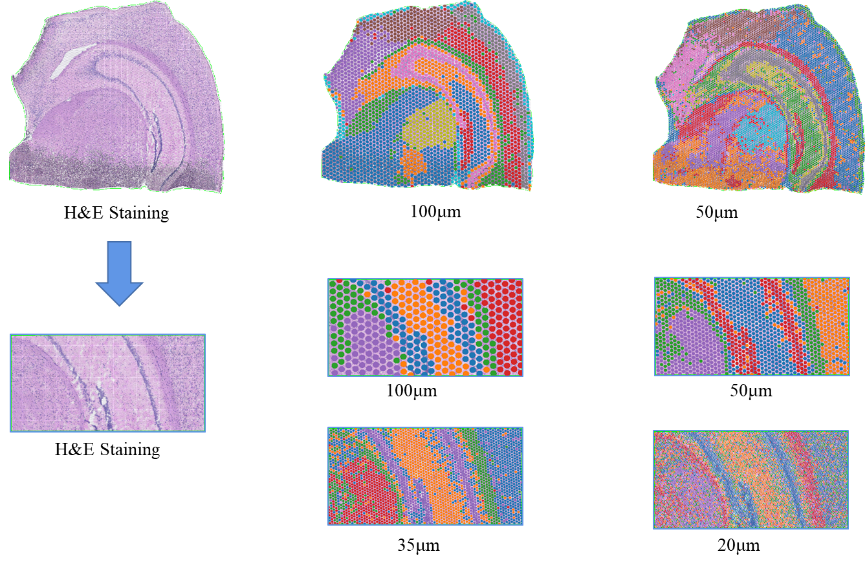
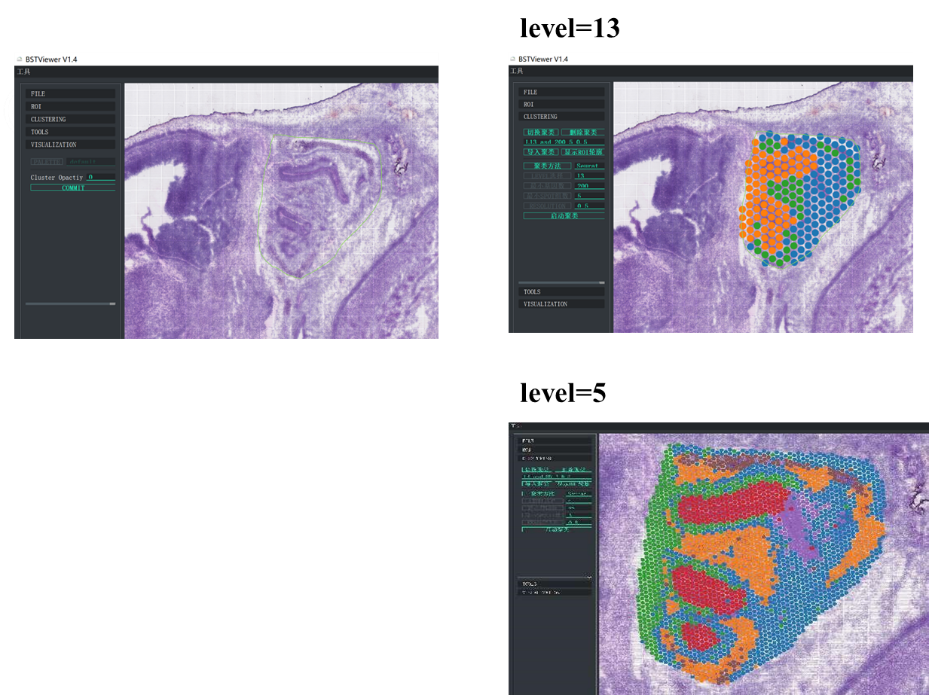
●Análise de resolución multinivel:Análise flexible multinivel que varía de 100 μm a 5 μm para resolver diversas características do tecido cunha resolución óptima.
●Posibilidade de utilizar a tecnoloxía de segmentación celular "Tres nunha diapositiva":Combinando a tinción de fluorescencia, a tinción de H&E e a secuenciación de ARN nunha única diapositiva, o noso algoritmo de análise "tres en un" permite a identificación dos límites celulares para a posterior transcriptómica baseada en células.
●Compatible con múltiples plataformas de secuenciación: Dispoñible tanto NGS como secuenciación de lectura longa.
●Deseño flexible de 1-8 áreas de captura activa: O tamaño da zona de captura é flexible, sendo posible utilizar 3 formatos (6,8 mm * 6,8 mm., 11 mm * 11 mm e 15 mm * 20 mm)
●Servizo de portelo único: Integra todos os pasos baseados na experiencia e na habilidade, incluíndo a criosección, a tinción, a optimización de tecidos, a codificación de barras espaciais, a preparación de bibliotecas, a secuenciación e a bioinformática.
●Bioinformática integral e visualización amigable dos resultados:O paquete inclúe 29 análises e máis de 100 cifras de alta calidade, combinadas co uso de software desenvolvido na casa para visualizar e personalizar a división celular e a agrupación de puntos.
●Análise e visualización de datos personalizados: dispoñible para diferentes solicitudes de investigación
●Equipo técnico altamente cualificado: con experiencia en máis de 250 tipos de tecidos e máis de 100 especies, incluíndo humanos, ratos, mamíferos, peixes e plantas.
●Actualizacións en tempo real de todo o proxecto: con control total do progreso experimental.
●Análise conxunta opcional con secuenciación de ARNm unicelular
Especificacións do servizo
|
Mostra Requisitos
| Biblioteca |
Estratexia de secuenciación
| Datos recomendados | Control de calidade |
| Criomostras incrustadas en OCT, 3 bloques por mostra | Biblioteca de ADNc S1000 | Illumina PE150 (outras plataformas dispoñibles) | 100K lecturas de PE por 100 uM (60-150 Gb) | RIN>7 |
Para obter máis detalles sobre a orientación de preparación de mostras e o fluxo de traballo do servizo, non dubide en falar con aExperto en BMKGENE
Fluxo de traballo do servizo
Na fase de preparación da mostra, realízase un ensaio inicial de extracción de ARN a granel para garantir que se pode obter un ARN de alta calidade. Na fase de optimización do tecido téñense e visualízanse as seccións e optimízanse as condicións de permeabilización para a liberación do ARNm do tecido. O protocolo optimizado aplícase despois durante a construción da biblioteca, seguido da secuenciación e análise de datos.
O fluxo de traballo completo do servizo implica actualizacións en tempo real e confirmacións do cliente para manter un bucle de feedback receptivo, garantindo a execución do proxecto sen problemas.
Os datos xerados por BMKMANU S1000 analízanse mediante o software "BSTMatrix", que está deseñado de forma independente por BMKGENE, xerando unha matriz de expresión xenética. A partir de aí, xérase un informe estándar que inclúe o control da calidade dos datos, a análise de mostras internas e a análise entre grupos.
● Control de calidade dos datos:
Saída de datos e distribución da puntuación de calidade
Detección de xenes por punto
Cobertura tisular
● Análise da mostra interna:
Riqueza xenética
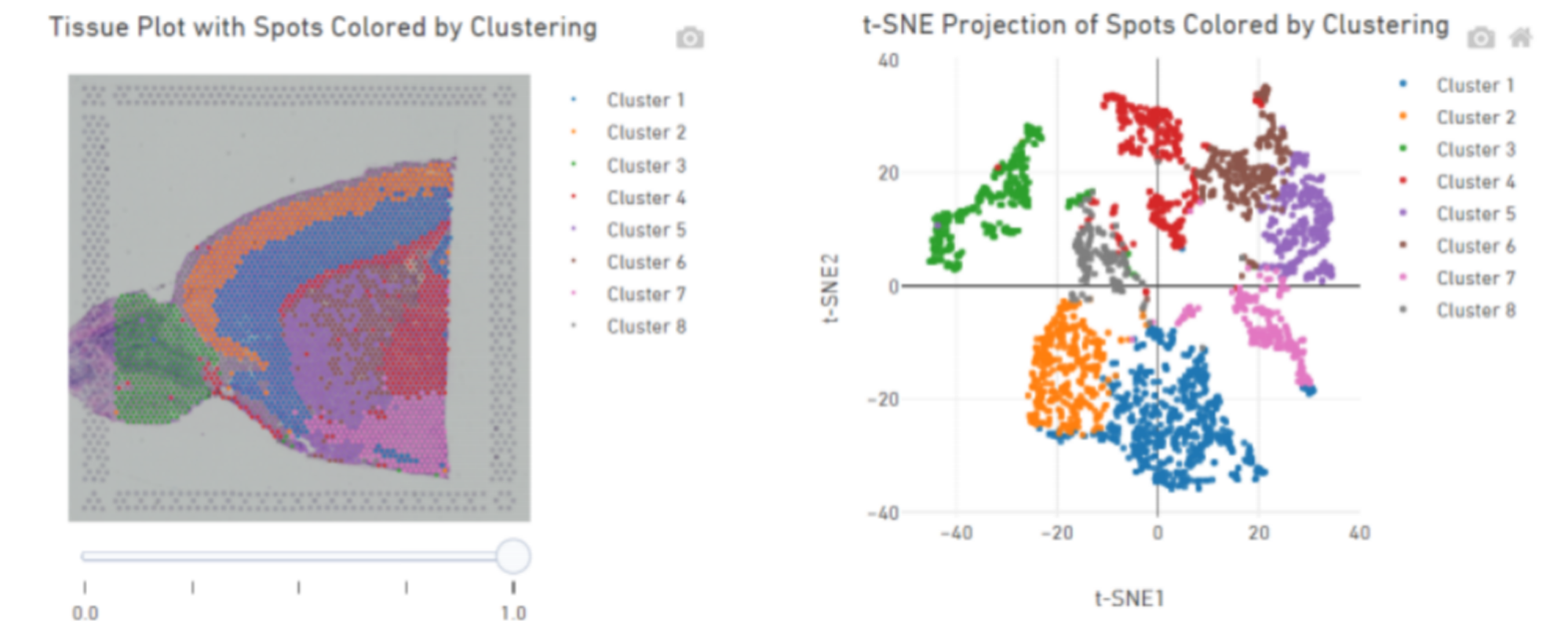
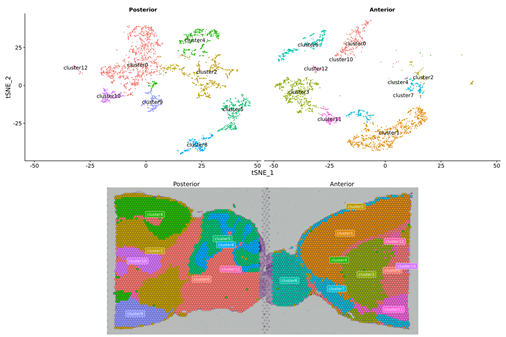
Agrupación puntual, incluída a análise de dimensións reducidas
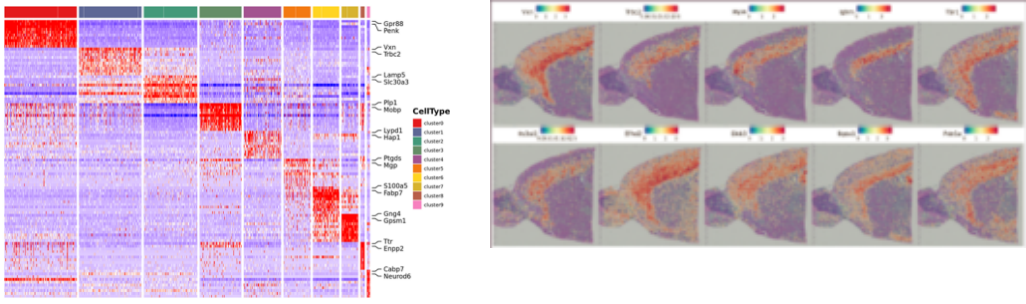
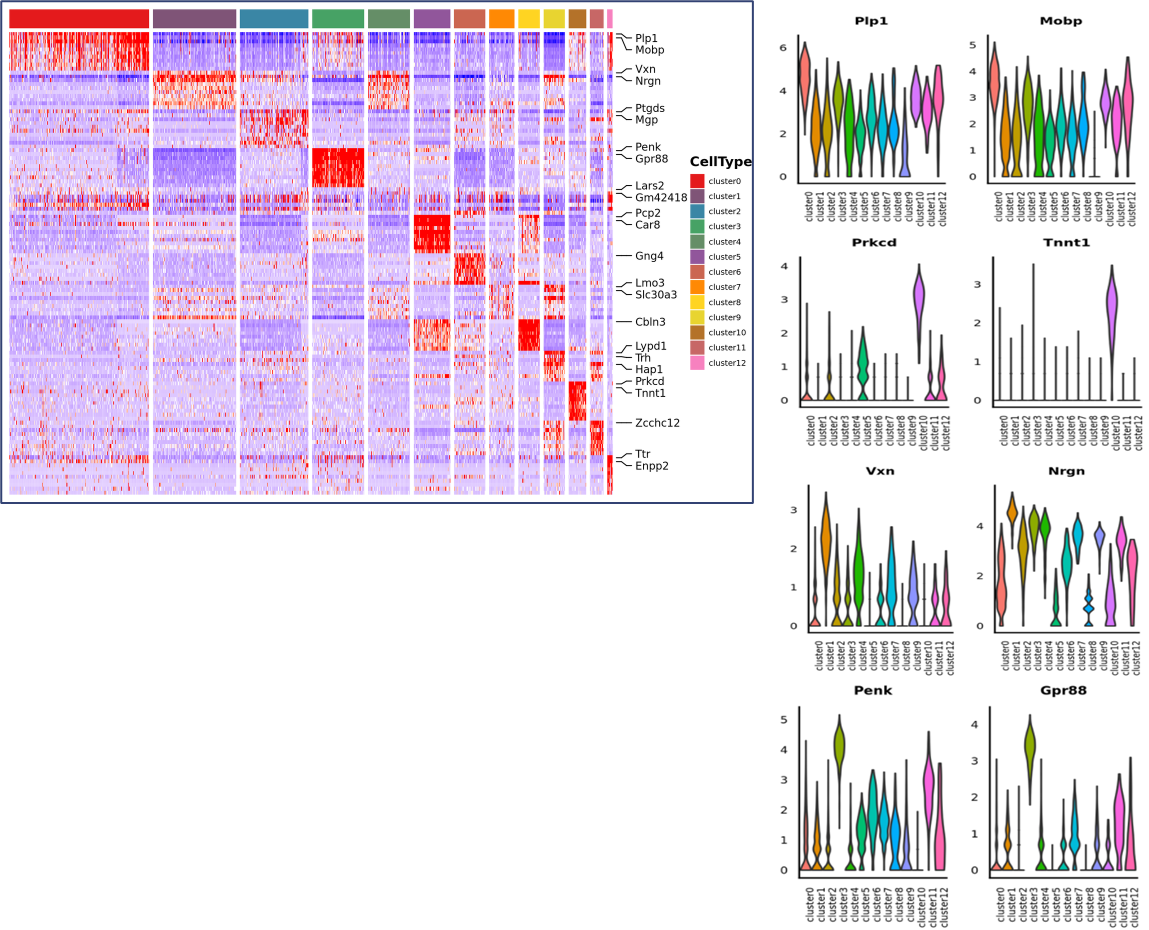
Análise de expresión diferencial entre clusters: identificación de xenes marcadores
Anotación funcional e enriquecemento de xenes marcadores
● Análise intergrupal:
Recombinación de puntos de ambas as mostras (por exemplo, enfermos e control) e recombinación
Identificación de xenes marcadores para cada cluster
Anotación funcional e enriquecemento de xenes marcadores
Expresión diferencial dun mesmo clúster entre grupos
Ademais, "BSTViewer" desenvolvido por BMKGENE é unha ferramenta fácil de usar que permite ao usuario visualizar a expresión xenética e a agrupación de puntos en diferentes resolucións.
BMKGene desenvolveu un software para unha visualización amigable
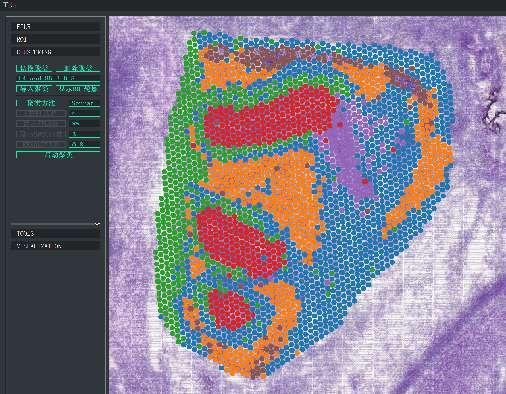
Agrupación de puntos BSTViewer con resolución multinivel
BSTCellViewer: división de células automática e manual
Análise da mostra interna
Agrupación puntual:
Identificación de xenes marcadores e distribución espacial:
Análise intergrupal
Combinación de datos de ambos grupos e recluster:
Xenes marcadores de novos clusters:
Explore os avances facilitados polos servizos de transcriptómica espacial de BMKGene coa tecnoloxía BMKManu S1000 nesta publicación destacada:
Song, X. et al. (2023) "A transcriptómica espacial revela células de clorenquima inducidas pola luz implicadas na promoción da rexeneración de brotes no callo do tomate".Actas da Academia Nacional de Ciencias dos Estados Unidos de América, 120 (38), páx. e2310163120. doi: 10.1073/pnas.2310163120
Ti, Y. et al. (2023) "Comparación sistemática de métodos transcriptómicos espaciais baseados en secuenciación",bioRxiv, páx. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.