Ensamble de xenoma bacteriano de novo
Características do servizo
● Con dúas opcións posibles para escoller en función do grao de integridade do xenoma desexado.
● Borrador de opción de xenoma: secuenciación de lectura curta con Illumina NovaSeq PE150.
● Completa o xenoma 0 gap.
● Secuenciación de lectura longa en Nanopore PromethION 48 ou PacBio Revio para a ensamblaxe do xenoma.
● Secuenciación de lectura curta en Illumina NovaSeq para a validación do xenoma e a corrección de erros (Nanopore) ou para xerar un borrador de xenoma.
Vantaxes do servizo
●Xenoma Zero-Gap garantido: Isto débese á integración da secuenciación Illumina coa secuenciación de lectura longa (Nanopore ou PacBio).
●Fluxo de traballo completo de bioinformática:Isto inclúe a montaxe do xenoma e a predición de múltiples elementos xenómicos, a anotación de xenes funcionais e visualizacións do xenoma como o diagrama de Circos.
●Amplia experiencia: Con máis de 20.000 xenomas microbianos reunidos, BMKGENE aporta máis dunha década de experiencia, un equipo de análise altamente cualificado, contido completo e excelente soporte posvenda.
●Soporte posvenda:O noso compromiso vai máis aló da finalización do proxecto cun período de servizo posvenda de 3 meses. Durante este tempo, ofrecemos seguimento do proxecto, asistencia para a resolución de problemas e sesións de preguntas e respostas para resolver calquera dúbida relacionada cos resultados.
●Múltiples estratexias de secuenciación dispoñibles:Para diferentes obxectivos de investigación e requisitos de integridade do xenoma.
Especificacións do servizo
| Servizo | Estratexia de secuenciación |
| Borrador do xenoma | Illumina PE150 100x |
| 0 Xenoma Gap | Nanopore 100x + Illumina PE150 100x Or Pacbio HiFi 30x + Illumina PE150 100x (opcional) |
Requisitos de mostra:
| Concentración (ng/µL) | Cantidade total (µg) | Volume (µL) | OD260/280 | OD260/230 | |
| PacBio | ≥20 | ≥1,2 | ≥20 | 1.7-2.2 | ≥1,0 |
| Nanoporo | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1,0-3,0 |
| Ilumina | ≥1 | ≥0,06 | ≥20 | - | - |
· Bacterias: ≥3,5x1010 células
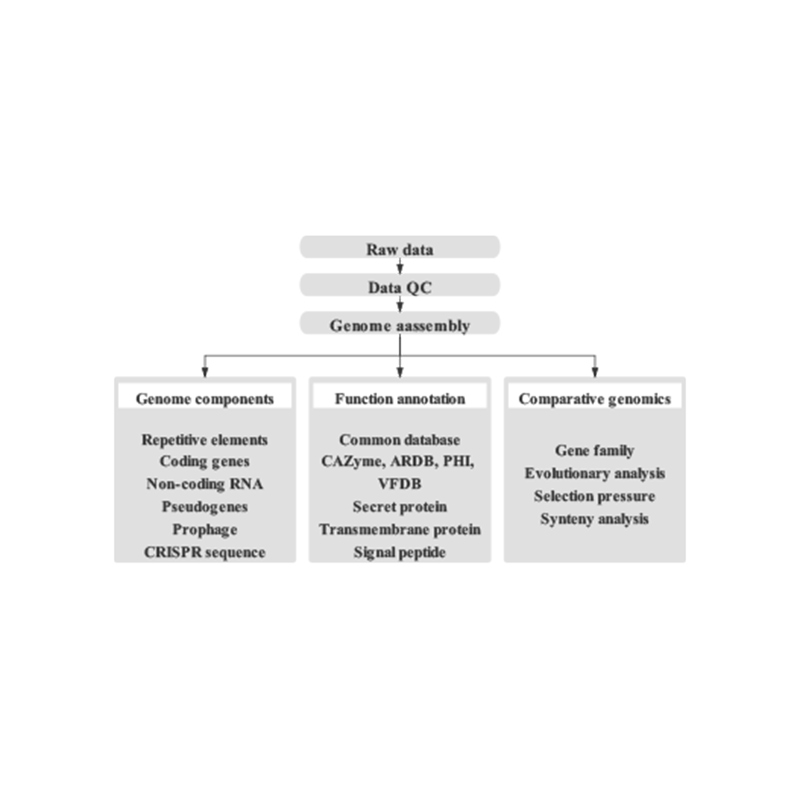
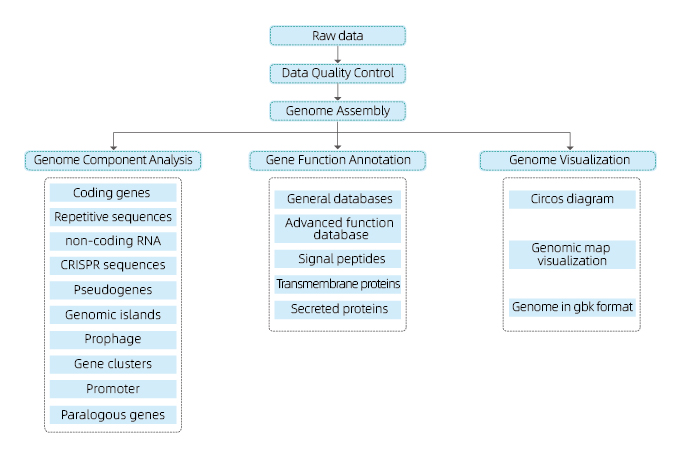
Fluxo de traballo do servizo

Entrega da mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
Inclúe a seguinte análise:
● Control de calidade de datos de secuenciación
● Asemblea do xenoma
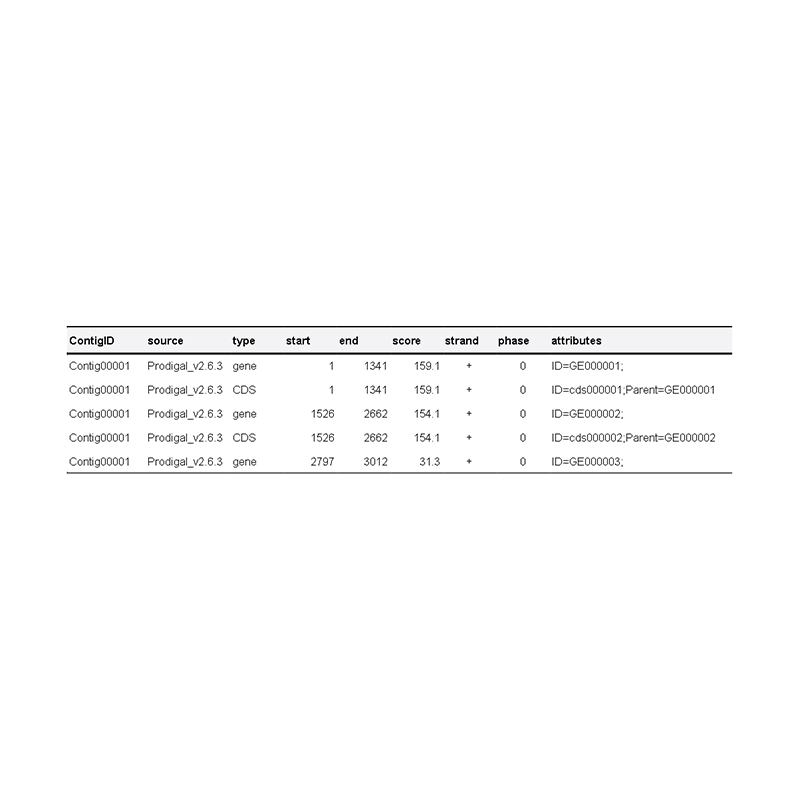
● Análise de compoñentes xenómicos: predición de CDS e elementos xenómicos múltiples
● Anotación funcional con múltiples bases de datos xerais (GO, KEGG, etc.) e bases de datos avanzadas (CARD, VFDB, etc.)
● Visualización do xenoma
Ofrecemos o xenoma nun formato fasta de fácil acceso e o ficheiro de anotación do xenoma (gff).
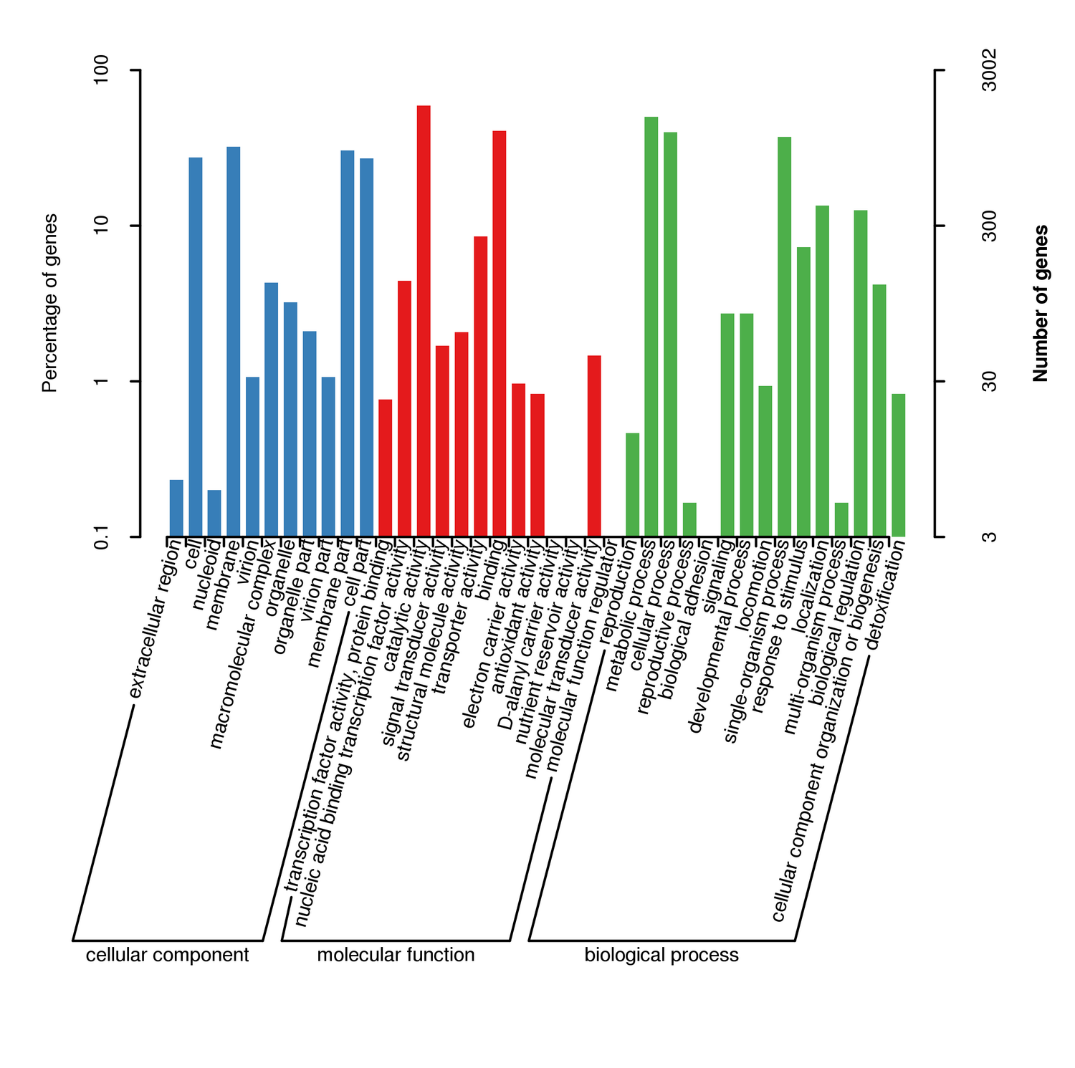
Anotación xenética - GO
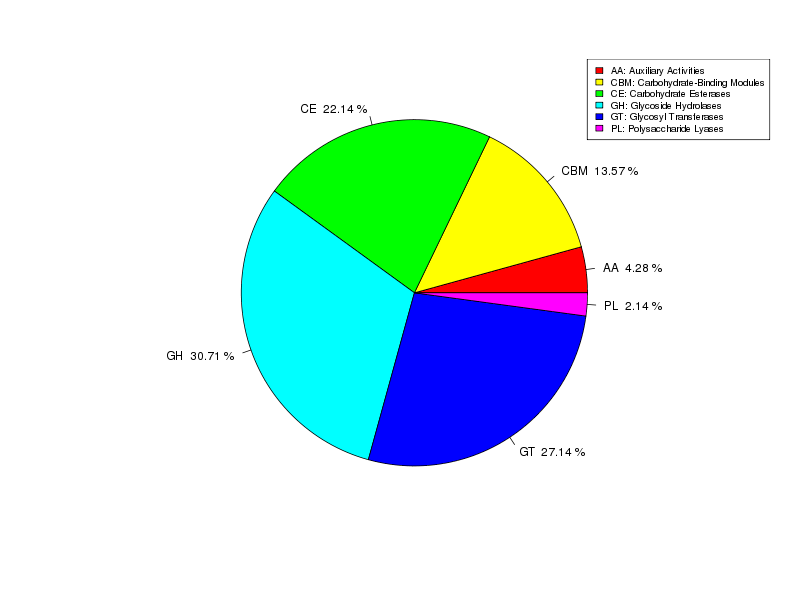
 Anotación xenética: carbohidratos CAZY
Anotación xenética: carbohidratos CAZY
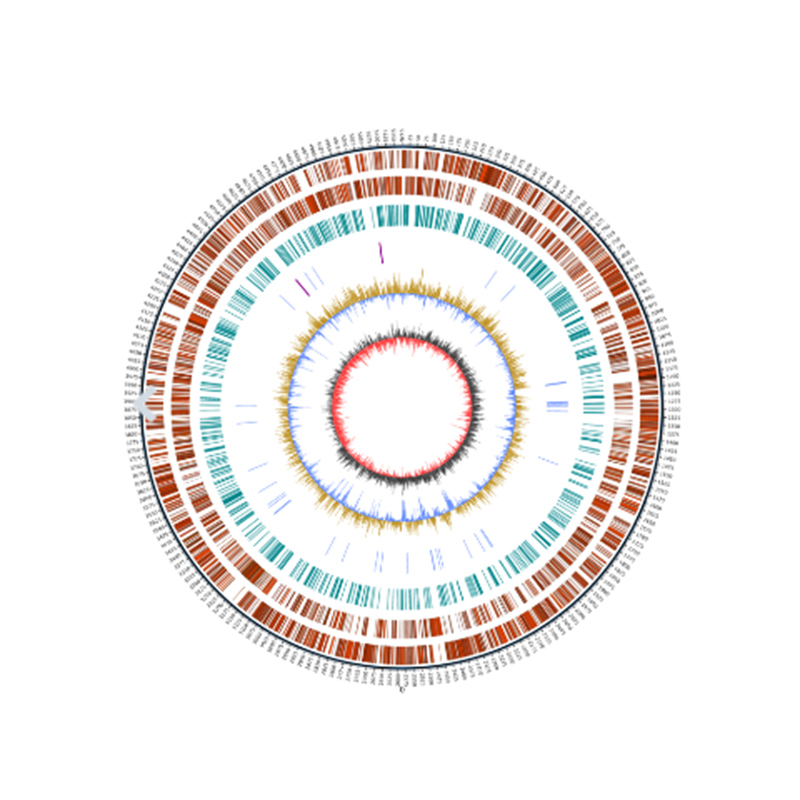
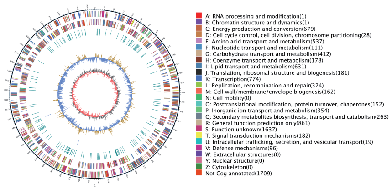
Visualización do xenoma – Trama Circos
Explore os avances facilitados polos servizos de ensamblaxe do xenoma bacteriano de BMKGene a través dunha colección de publicacións seleccionada.
Dai, W. et al. (2023) "Descubrimento de Bacteroides uniformis F18-22 como unha bacteria probiótica segura e nova para o tratamento da colite ulcerosa do colon humano saudable",Revista Internacional de Ciencias Moleculares, 24 (19), páx. 14669. doi: 10.3390/IJMS241914669/S1.
Kang, Q. et al. (2021) "Illados de Proteus mirabilis multirresistentes que transportan blaOXA-1 e blaNDM-1 da vida salvaxe en China: aumento do risco para a saúde pública",Zooloxía Integrativa, 16 (6), páxinas 798–809. doi: 10.1111/1749-4877.12510.
Wang, TT et al. (2017) "A secuencia do xenoma completa do endófito Bacillus flexus KLBMP 4941 revela o seu mecanismo de promoción do crecemento das plantas e a base xenética para a tolerancia á sal".Revista de Biotecnoloxía, 260, páxinas 38–41. doi: 10.1016/J.JBIOTEC.2017.09.001.
Wang, X. et al. (2021) "Multiple-Replicon Resistance Plasmids of Klebsiella Mediate Extensive Dissemination of Antimicrobial Genes",Fronteiras en Microbioloxía, 12, páx. 754931. doi: 10.3389/FMICB.2021.754931/BIBTEX.