10X Genomics Visium Transcriptoma espacial
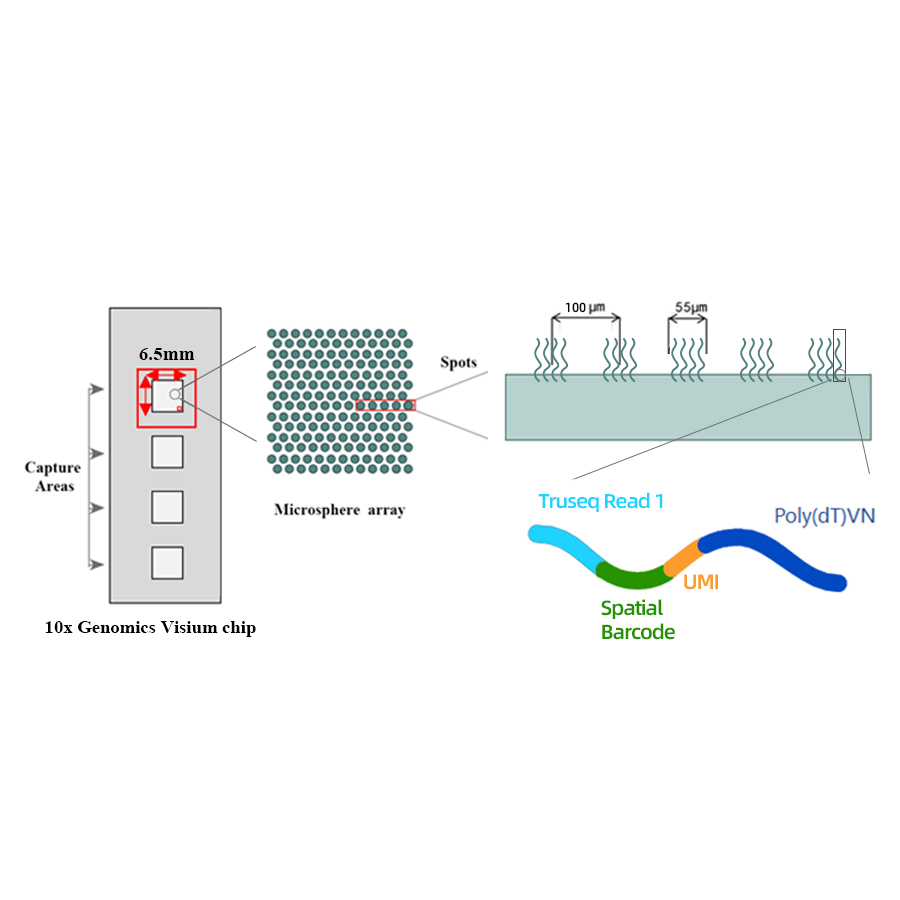
Esquema técnico

Características
● Resolución: 100 µm
● Diámetro da mancha: 55 µm
● Número de manchas: 4992
● Área de captura: 6,5 x 6,5 mm
● Cada mancha codificada está cargada con cebadores compostos por 4 seccións:
- cola poli (dt) para a cebada de ARNm e a síntese de ADNc
- Identificador molecular único (UMI) para corrixir o sesgo de amplificación
- Código de barras espacial
- Secuencia de vinculación de Ler parcial 1 Primador de secuenciación
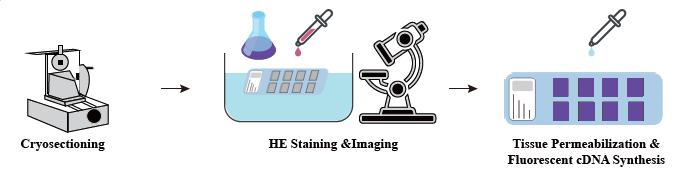
● Tinción de H&E de seccións
Vantaxes
●Servizo único: Integra toda a experiencia e os pasos baseados en habilidades, incluíndo sección de crio, tinción, optimización de tecidos, codificación espacial, preparación da biblioteca, secuenciación e bioinformática.
● Equipo técnico altamente cualificado: con experiencia en máis de 250 tipos de tecidos e máis de 100 especies, incluíndo humanos, ratos, mamíferos, peixes e plantas.
●Actualización en tempo real sobre todo o proxecto: con control total do progreso experimental.
●Bioinformática estándar integral:O paquete inclúe 29 análises e 100+ cifras de alta calidade.
●Análise e visualización de datos personalizados: Dispoñible para diferentes solicitudes de investigación.
●Análise de articulacións opcionais con secuenciación de ARNm de células monocelulares
Especificacións
| Requisitos da mostra | Biblioteca | Estratexia de secuenciación | Datos recomendados | Control de calidade |
| Mostras de crio incrustadas en OCT (Diámetro óptimo: aproximadamente 6x6x6 mm³) 2 bloques por mostra | Biblioteca de ADNc de 10x Visium | Illumina PE150 | 50k PE Reads por punto (60 GB) | Rin> 7 |
Para obter máis detalles sobre a preparación da mostra e o fluxo de traballo do servizo, non dubide en falar cunBMKGENE Expert
Fluxo de traballo de servizo
Na fase de preparación da mostra, realízase un ensaio inicial de extracción de ARN a granel para garantir que se poida obter un ARN de alta calidade. Na fase de optimización de tecidos as seccións son manchadas e visualizadas e optimízanse as condicións de permeabilización para a liberación de ARNm do tecido. A continuación, aplícase o protocolo optimizado durante a construción da biblioteca, seguido de secuenciación e análise de datos.
O fluxo de traballo completo de servizo implica actualizacións en tempo real e confirmacións do cliente para manter un bucle de retroalimentación sensible, garantindo unha boa execución do proxecto.

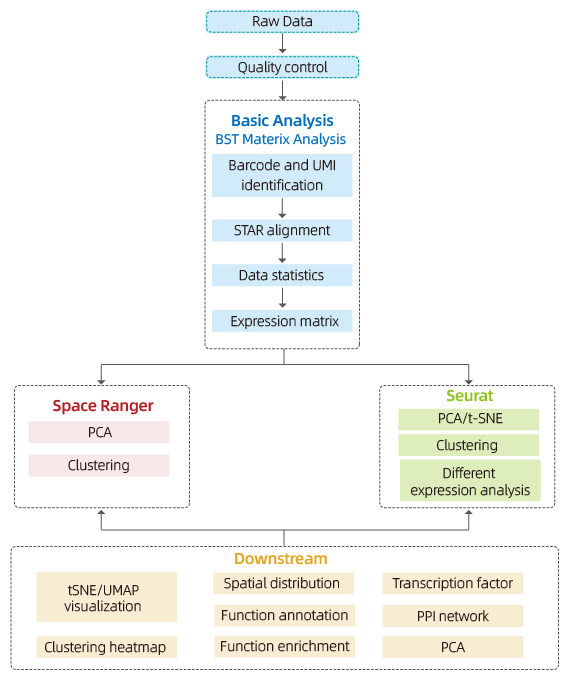
Inclúe a seguinte análise:
Control de calidade de datos:
o Saída de datos e distribución de puntuación de calidade
o Detección de xenes por punto
o Cobertura do tecido
Análise de mostras interiores:
o riqueza xénica
o Clustering Spot, incluída a análise de dimensións reducidas
o Análise de expresión diferencial entre clústers: identificación de xenes marcadores
o Anotación funcional e enriquecemento dos xenes marcadores
Análise entre grupos
o Re-combinación de manchas de ambas mostras (por exemplo, enfermo e control) e re-cluster
o Identificación de xenes marcadores para cada clúster
o Anotación funcional e enriquecemento dos xenes marcadores
o Expresión diferencial do mesmo clúster entre grupos
Análise de mostras interiores
Clustering Spot

Identificación de xenes marcadores e distribución espacial


Análise entre grupos
Combinación de datos de ambos grupos e re-cluster
Xenes marcadores de novos clústers
Explora os avances facilitados polo servizo de transcriptomics espaciais de BMKGene por 10X Visium nestas publicacións destacadas:
Chen, D. et al. (2023) 'MTHL1, un potencial homólogo de Drosophila de GPCRs de adhesión de mamíferos, está implicado en reaccións antitumorales ás células oncogénicas inxectadas nas moscas ",Actas da Academia Nacional de Ciencias dos Estados Unidos de América, 120 (30), p. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) "O aceiro permite a delineación de alta resolución de datos transcriptómicos espatiotemporais",Informes en bioinformática, 24 (2), pp. 1-10. doi: 10.1093/bib/bbad068.
Liu, C. et al. (2022) "Un atlas espatiotemporal da organoxénese no desenvolvemento de flores de orquídeas",Investigación de ácidos nucleicos, 50 (17), pp. 9724–9737. doi: 10.1093/nar/gkac773.
Wang, J. et al. (2023) "Integrar a transcriptómica espacial e a secuenciación de ARN dun só núcleo revela as potenciais estratexias terapéuticas para o leiomioma uterino",Revista Internacional de Ciencias Biolóxicas, 19 (8), pp. 2515-2530. doi: 10.7150/ijbs.83510.