-

Metagenomic Sequencing -NGS
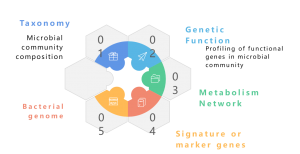
In metagenoom is in samling fan it totale genetysk materiaal fan in mingde mienskip fan organismen, lykas miljeu- en minsklike metagenomen. It befettet genomen fan sawol kultiverbere as net te kultivearjende mikroorganismen. Metagenomyske folchoarder fan Shotgun mei NGS makket de stúdzje fan dizze yngewikkelde genomyske lânskippen ynbêde yn miljeumonsters mooglik troch mear te leverjen dan taksonomyske profilearring, en jouwe ek korrelige ynsjoch yn soarten ferskaat, oerfloeddynamyk en komplekse populaasjestruktueren. Beyond taksonomyske stúdzjes, shotgun metagenomics biedt ek in funksjoneel genomics perspektyf, wêrtroch it ferkennen fan kodearre genen en harren putative rollen yn ekologyske prosessen. Uteinlik draacht de oprjochting fan korrelaasjenetwurken tusken genetyske eleminten en omjouwingsfaktoaren by oan in holistysk begryp fan 'e yngewikkelde ynteraksje tusken mikrobiele mienskippen en har ekologyske eftergrûn. Ta beslút, metagenomic sequencing stiet as in pivotal ynstrumint foar it unraveling de genomyske intricacies fan ferskate mikrobiële mienskippen, ferljochtsje de mearsidige relaasjes tusken genetika en ekology binnen dizze komplekse ekosystemen.
Platfoarms: Illumina NovaSeq en DNBSEQ-T7
-

Metagenomic Sequencing-TGS
In metagenoom is in samling fan it genetysk materiaal fan in mingde mienskip fan organismen, lykas miljeu- en minsklike metagenomen. It befettet genomen fan sawol kultiverbere as net te kultivearjende mikroorganismen. Metagenomyske sequencing makket de stúdzje fan dizze yngewikkelde genomyske lânskippen ynbêde yn ekologyske samples mooglik troch mear te leverjen dan taksonomyske profilearring. It biedt ek in funksjoneel genomika-perspektyf troch it ferkennen fan de kodearre genen en har putative rollen yn miljeuprosessen. Wylst tradisjonele shotgun-oanpak mei Illumina-sequencing in protte brûkt binne yn metagenomyske stúdzjes, hat de komst fan Nanopore en PacBio langlêzen sequencing it fjild feroare. Nanopore- en PacBio-technology ferbetterje streamôfwerts bioinformatyske analyzes, benammen metagenoom-assemblage, en soargje foar mear trochgeande gearkomsten. Rapporten jouwe oan dat Nanopore-basearre en PacBio-basearre metagenomics mei súkses folsleine en sletten baktearjele genomen hawwe generearre fan komplekse mikrobiomen (Moss, EL, et al., Nature Biotech, 2020). It yntegrearjen fan Nanopore-lêzen mei Illumina-lêzen biedt in strategyske oanpak foar flaterkorreksje, wat de ynherinte lege krektens fan Nanopore fermindert. Dizze synergistyske kombinaasje benut de sterkte fan elk sequencing-platfoarm, en biedt in robúste oplossing om potinsjele beheiningen te oerwinnen en de krektens en betrouberens fan metagenomyske analyzes te befoarderjen.
Platfoarm: Nanopore PromethION 48, Illumia en PacBio Revio
-

Whole genome bisulfite sequencing (WGBS)
Whole Genome Bisulfite Sequencing (WGBS) stiet as de gouden-standertmetodology foar yngeande ferkenning fan DNA-methylaasje, spesifyk de fyfde posysje yn cytosine (5-mC), in pivotale regulator fan geneekspresje en sellulêre aktiviteit. It prinsipe ûnderlizzende WGBS omfettet bisulfite-behanneling, wêrtroch de konverzje fan unmethylearre cytosines nei uracil (C nei U) feroarsake wurdt, wylst methylearre cytosines ûnferoare litte. Dizze technyk biedt resolúsje op ien basis, wêrtroch ûndersikers it methylome wiidweidich kinne ûndersykje en abnormale methylaasjepatroanen ûntdekke ferbûn mei ferskate betingsten, benammen kanker. Troch WGBS te brûken, kinne wittenskippers ongeëvenaarde ynsjoch krije yn genome-brede methylaasjelânskippen, en jouwe in nuansearre begryp fan 'e epigenetyske meganismen dy't ferskate biologyske prosessen en sykten ûnderlizze.
-

Assay foar transposase-tagonklik chromatine mei hege trochfier-sekwinsje (ATAC-seq)
ATAC-seq is in sequencingtechnyk mei hege trochset dy't brûkt wurdt foar analyse fan genoombrede chromatine tagonklikens. It gebrûk leveret djipper begryp fan 'e komplekse meganismen fan globale epigenetyske kontrôle oer geneekspresje. De metoade brûkt in hyperaktive Tn5-transposase om tagelyk iepen chromatine-regio's te fragmintearjen en te markearjen troch sequencing-adapters yn te setten. Folgjende PCR-amplifikaasje resulteart yn 'e skepping fan in sequencing-bibleteek, dy't de wiidweidige identifikaasje fan iepen chromatine-regio's mooglik makket ûnder spesifike romte-tiid-betingsten. ATAC-seq jout in holistyske werjefte fan tagonklike chromatine lânskippen, yn tsjinstelling ta metoaden dy't allinnich rjochtsje op transkripsje faktor binende sites of spesifike histon-modifisearre regio. Troch dizze iepen chromatine-regio's te foltôgjen, ûntbleatet ATAC-seq regio's dy't mear kâns hawwe op aktive regeljouwingssekwinsjes en potinsjele transkripsjefaktor-binende siden, en biedt weardefolle ynsjoch yn 'e dynamyske modulaasje fan gen-ekspresje oer it genoom.
-

16S / 18S / ITS Amplicon Sequencing-PacBio
De 16S en 18S rRNA genen, tegearre mei de Internal Transcribed Spacer (ITS) regio, tsjinje as pivotale molekulêre fingerprinting markers fanwege harren kombinaasje fan tige konservearre en hyper-fariabele regio, wêrtroch't se ûnskatbere wearde ark foar karakterisearjen prokaryotic en eukaryotic organismen. Amplifikaasje en sekwinsje fan dizze regio's biede in isolaasjefrije oanpak foar it ûndersykjen fan de mikrobiële gearstalling en ferskaat oer ferskate ekosystemen. Wylst Illumina sequencing typysk rjochte is op koarte hyperfariabele regio's lykas V3-V4 fan 16S en ITS1, is it oantoand dat superieure taksonomyske annotaasje te berikken is troch it foltôgjen fan 'e folsleine lingte fan 16S, 18S en ITS. Dizze wiidweidige oanpak resulteart yn hegere persintaazjes fan sekuer klassifisearre sekwinsjes, it berikken fan in nivo fan resolúsje dat útwreidet nei soarten identifikaasje. PacBio's Single-Molecule Real-Time (SMRT) sequencing-platfoarm opfalt troch it leverjen fan heul krekte lange lêzings (HiFi) dy't de amplicons fan 'e folsleine lingte dekke, en konkurrearje mei de presyzje fan Illumina-sequencing. Dizze mooglikheid lit ûndersikers in ongeëvenaard foardiel berikke - in panoramysk werjefte fan it genetyske lânskip. De útwreide dekking ferheft signifikant de resolúsje yn soarten annotaasje, benammen binnen baktearjele as skimmelmienskippen, wêrtroch in djipper begryp fan 'e yngewikkeldheden fan mikrobiele populaasjes mooglik is.
-

16S / 18S / ITS Amplicon Sequencing-NGS
Amplicon-sekwinsje mei Illumina-technology, spesifyk rjochte op 'e 16S, 18S, en ITS genetyske markers, is in krêftige metoade foar it unraveljen fan de fylogeny, taksonomy en soarten oerfloed binnen mikrobiele mienskippen. Dizze oanpak omfettet sekwinsje fan 'e hyperfariabele regio's fan genetyske markers foar húshâlding. Oarspronklik yntrodusearre as in molekulêre fingerprint trochWoese et alyn 1977, dizze technyk hat revolutionized microbiome profilearring troch it ynskeakeljen fan isolaasje-frije analyzes. Troch de sekwinsje fan 16S (baktearjes), 18S (skimmels), en ynterne transkribearre spacer (ITS, fungi), kinne ûndersikers net allinich oerfloedige soarten identifisearje, mar ek seldsume en net identifisearre. Wiid oannommen as in pivotal ark, is amplicon-sekwinsje ynstruminteel wurden yn it ûnderskieden fan differinsjaal mikrobiële komposysjes yn ferskate omjouwings, ynklusyf de minsklike mûle, darmen, kruk, en fierder.
-

Bakteriële en fungal hiele genome re-sequence
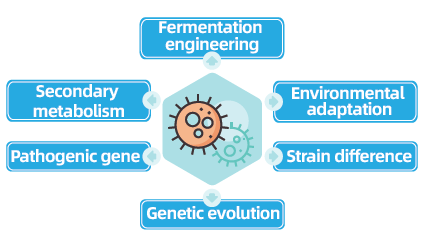
Projekten foar opnij foltôgjen fan baktearjes en skimmels fan gehiel genomen binne pivotal foar it fuortsterkjen fan mikrobiële genomika troch it ynskeakeljen fan it foltôgjen en fergelykjen fan mikrobiële genomen. Dit fasilitearret fermentaasjetechnyk, de optimalisaasje fan yndustriële prosessen, en it ferkennen fan sekundêre metabolismepaden. Fierder is skimmel- en baktearjele re-sekwinsje krúsjaal foar it begripen fan miljeu-oanpassing, it optimalisearjen fan stammen en it iepenbierjen fan genetyske evolúsjedynamyk, mei brede gefolgen yn medisinen, lânbou en miljeuwittenskip.
-

PacBio-folsleine 16S/18S/ITS Amplicon Sequencing
Amplicon (16S / 18S / ITS) platfoarm is ûntwikkele mei jierrenlange ûnderfining yn mikrobieel ferskaat projektanalyse, dy't standerdisearre basisanalyse en personaliseare analyse befettet: basisanalyse beslacht de mainstream-analyze-ynhâld fan hjoeddeistige mikrobieel ûndersyk, de analyse-ynhâld is ryk en wiidweidich, en analyseresultaten wurde presintearre yn 'e foarm fan projektrapporten; De ynhâld fan personaliseare analyze is ferskaat. Samples kinne wurde selekteare en parameters kinne fleksibel ynsteld wurde neffens it basisanalyserapport en ûndersyksdoel, om personaliseare easken te realisearjen. Windows bestjoeringssysteem, ienfâldich en fluch.
-

PacBio-folsleine transkriptoom (net-referinsje)
Troch Pacific Biosciences (PacBio) Isoform-sekwinsjegegevens as ynfier te nimmen, is dizze app yn steat om transkripsjesekwinsjes fan folsleine lingte te identifisearjen (sûnder gearstalling). Troch sekwinsjes fan folsleine lingte yn kaart te bringen tsjin referinsjegenoom, kinne transkripsjes optimalisearre wurde troch bekende genen, transkripsjes, kodearringregio's, ensfh. Yn dit gefal kin krektere identifikaasje fan mRNA-struktueren, lykas alternative splicing, ensfh. Mienskiplike analyze mei NGS-transkriptom-sekwinsjegegevens makket wiidweidigere annotaasje en krekter kwantifikaasje yn ekspresje op transkripsjenivo mooglik, wat foar in grut part profitearret downstream differinsjaal ekspresje en funksjonele analyse.
-

Reduced Representation Bisulfite Sequencing (RRBS)
Reduced Representation Bisulfite Sequencing (RRBS) is ûntstien as in kosten-effektyf en effisjint alternatyf foar Whole Genome Bisulfite Sequencing (WGBS) yn DNA-methylaasjeûndersyk. Wylst WGBS wiidweidige ynsjoggen leveret troch it heule genoom te ûndersiikjen by resolúsje fan ien basis, kinne de hege kosten in beheinende faktor wêze. RRBS fermindert dizze útdaging strategysk troch selektyf in represintatyf diel fan it genoom te analysearjen. Dizze metodyk fertrout op de ferriking fan CpG-eilân-rike regio's troch MspI-spjalting folge troch grutte seleksje fan 200-500/600 bps fragminten. Dêrtroch wurde allinich regio's proximaal foar CpG-eilannen sequearre, wylst dy mei fiere CpG-eilannen wurde útsletten fan 'e analyse. Dit proses, kombinearre mei bisulfite-sekwinsje, makket it mooglik foar detectie fan hege resolúsje fan DNA-methylaasje, en de sequencing-oanpak, PE150, rjochtet him spesifyk op 'e úteinen fan' e ynserts yn stee fan 'e midden, wêrtroch't de effisjinsje fan methylaasjeprofilearring ferheget. De RRBS is in ûnskatbere wearde ark dat kosten-effektyf DNA-methylaasjeûndersyk mooglik makket en kennis fan epigenetyske meganismen foarútgong.
-

Prokaryotyske RNA-sekwinsje
RNA-sekwinsje makket de wiidweidige profilearring fan alle RNA-transkrippen binnen sellen ûnder spesifike omstannichheden mooglik. Dizze avansearre technology tsjinnet as in krêftich ark, ûntbleatet yngewikkelde gene-ekspresjeprofilen, genstruktueren en molekulêre meganismen ferbûn mei ferskate biologyske prosessen. Wiid oannommen yn fûnemintele ûndersyk, klinyske diagnoaze, en medisynûntwikkeling, RNA-sekwinsje biedt ynsjoch yn 'e kompleksjes fan sellulêre dynamyk en genetyske regeling. Us prokaryotyske RNA-monsterferwurking is ôfstimd foar prokaryotyske transkriptomen, wêrby't rRNA-útputting en direksjonele biblioteektarieding belutsen is.
Platfoarm: Illumina NovaSeq
-

Metatranskriptoom-sekwinsje
It brûken fan Illumina-sekwinsjetechnology, BMKGENE's metatranscriptome-sekwinsjetsjinst ûntbleatet de dynamyske gen-ekspresje fan in ferskaat array fan mikroben, oerspant eukaryoten oant prokaryoten en firussen, binnen natuerlike omjouwings lykas boaiem, wetter, see, kruk, en de darm. Us wiidweidige tsjinst stelt ûndersikers yn steat om te ferdjipjen yn 'e folsleine geneekspresjeprofilen fan komplekse mikrobiele mienskippen. Beyond taksonomyske analyse, fasilitearret ús tsjinst foar metatranskriptoomsekwinsje ferkenning nei funksjonele ferriking, en smyt ljocht op differinsjaal útdrukte genen en har rollen. Untdek in skat oan biologyske ynsjoch as jo navigearje troch de komplekse lânskippen fan genekspresje, taksonomyske ferskaat en funksjonele dynamyk binnen dizze ferskate miljeu-nissen.
-

De novo Fungal Genome Assembly
BMKGENE biedt alsidige oplossingen foar fungal genomes, catering oan ferskate ûndersyk behoeften en winske genome folsleinens. It brûken fan koarte-lêzen Illumina-sekwinsje allinich lit de generaasje fan in konseptgenoom mooglik meitsje. Koarte lêzen en langlêzen sequencing mei Nanopore of Pacbio wurde kombinearre foar in mear ferfine skimmelgenoom mei langere contigs. Boppedat fersterket it yntegrearjen fan Hi-C-sekwinsje de mooglikheden fierder, wêrtroch it berikken fan in folslein genoom op chromosoomnivo mooglik is.