Metatranskriptoom-sekwinsje
Service Features
● rRNA útputting folge troch directional mRNA bibleteek tarieding.
● Sequencing op Illumina NovaSeq.
Service Foardielen
●Studearje de feroaringen fan komplekse mikrobiele mienskippen:Dit bart op it transkripsjenivo en ferkenne potensjele nije genen.
●Ferklearje mikrobyske mienskip ynteraksjes mei de host of omjouwing.
●Wiidweidige Bioinformatic Analysis: Dit jout ynsjoch oer mienskip taksonomyske en funksjonele komposysjes, likegoed as differinsjaaloperator gene ekspresje analyze.
●Wiidweidige geneannotaasje:It brûken fan up-to-date genfunksjedatabases foar ynformative geneekspresjeynformaasje fan mikrobiele mienskippen.
●Stipe nei ferkeap:Us ynset giet fierder as projektfoltôging mei in 3-moanne tsjinstperioade nei ferkeap. Yn dizze tiid biede wy projektopfolging, help by it oplossen fan problemen, en Q&A-sesjes om alle fragen oan te pakken yn ferbân mei de resultaten.
Service Spesifikaasjes
| Sequencing platfoarm | Sequencing Strategy | Gegevens oanrikkemandearre | Data Quality Control |
| Illumina NovaSeq | PE150 | 12 gb | Q30≥85% |
Sample Requirements
| Konsintraasje (ng/µL) | Totaal bedrach (µg) | Volume (µL) | OD260/280 | OD260/230 | RIN |
| ≥50 | ≥1.0 | ≥20 | 1,8-2,0 | 1,0-2,5 | ≥6,5 |
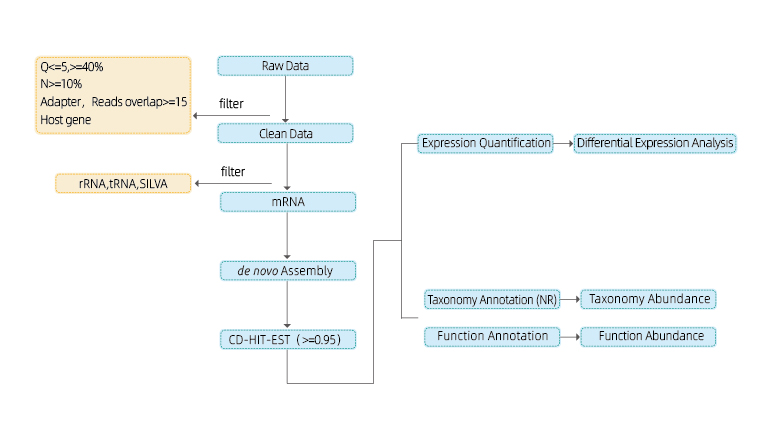
Service Work Flow

Sample levering

Biblioteekbou

Sequencing

Data analyze

Nei-ferkeap tsjinsten
Omfettet de folgjende analyze:
● Sequencing Data Quality Control
● Transkripsje Gearkomste
● Taksonomyske annotaasje en oerfloed
● Funksjonele annotaasje en oerfloed
● Ekspresje kwantifikaasje en differinsjaal analyze
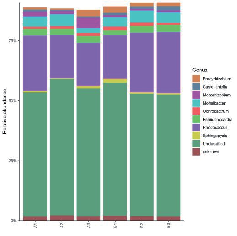
Taksonomyske ferdieling fan elke stekproef:
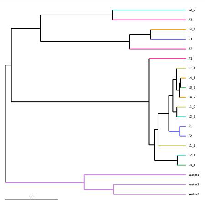
Beta ferskaat analyse: UPGMA
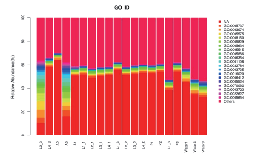
Funksjonele annotaasje - GO oerfloed
Differinsjaal taksonomy oerfloed - LEFSE
Ferkenne de foarútgong fasilitearre troch BMKGene's meta transcriptomics sequencing tsjinsten fia in gearstalde kolleksje fan publikaasjes.
Lu, Z. et al. (2023) 'Soertolerânsje fan lactate-gebrûkende baktearjes fan' e oarder Bacteroidales draacht by oan previnsje fan ruminal acidosis yn geiten oanpast oan in dieet mei hege konsintraasje.Animal Nutrition, 14, s. 130–140. doi: 10.1016/J.ANINU.2023.05.006.
Song, Z. et al. (2017) 'Unraveling kearnfunksjonele mikrobiota yn tradysjonele fermentaasje fan fêste steat troch hege-throughput amplicons en metatranscriptomics sequencing',Frontiers in Microbiology, 8 (JUL). doi: 10.3389 / FMICB.2017.01294 / FULL.
Wang, W. et al. (2022) 'Novel Mycoviruses Discovered from a Metatranscriptomics Survey of the Phytopathogenic Alternaria Fungus',Firussen, 14(11), s. 2552. doi: 10.3390/V14112552/S1.
Wei, J. et al. (2022) 'Parallelle metatranskriptoomanalyze lit degradaasje sjen fan sekundêre plantmetaboliten troch kevers en har darmsymbionten',Molekulêr Ekology, 31(15), s. 3999–4016. doi: 10.1111/MEC.16557.