Buldel Segregant Analyse
Tsjinstbewusteens

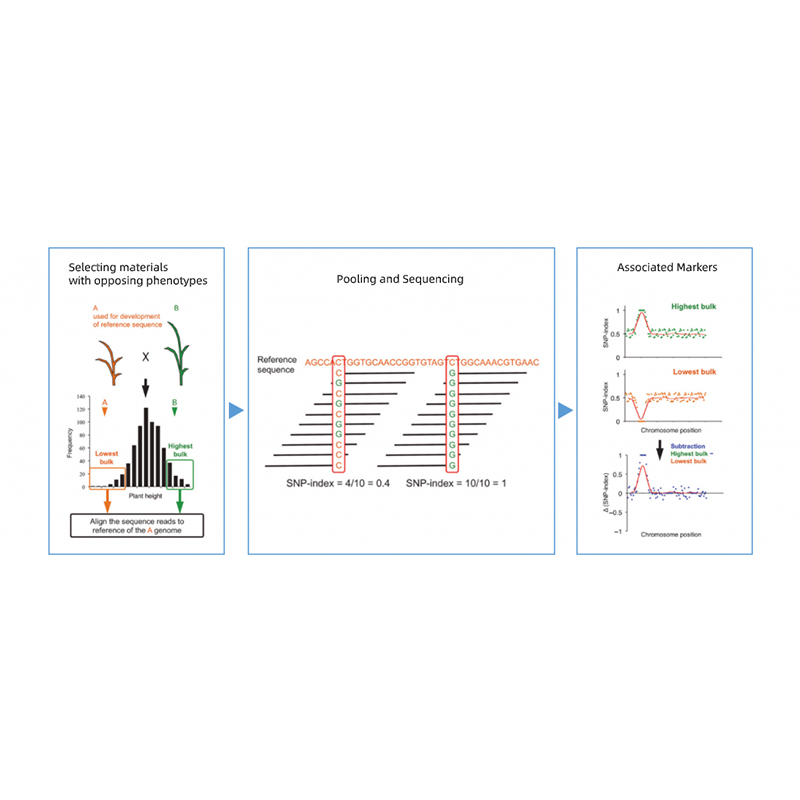
Takagi et al., De plantjoernaal, 2013
● Ekskurge lokalisaasje: it mingjen fan it plekken mei 30 + 30 oant 200 + 200 yndividuen om eftergrûngeluid te minimalisearjen; Net-Synonymme Mutatants-basearre kandidaat-regio-prognology.
● Untfange analyze: yn-djipte kandidaat Gene Funksje-funksje annotaasje, ynklusyf Nr, Swisssprot, Gean, gean, Kegg, kog, ensf.,
● Faster omslach tiid: Rapid Gene Localization binnen 45 wurkdagen.
● Wiidweidige ûnderfining: BMK hat bydroegen yn tûzenen trekken fan 'e pleatslike soarten, dieden fan ferskate soarten lykas gewearen, akwatyske produkten, bosk, blommen, fruit, ensfh.
Tsjinstspesifikaasjes
Befolking:
Segeare nammentlike fan âlders mei tsjinoerstelde fenotypes.
bgl. F2-nagersen, backcrossing (f.Kr.), RECOMBINANT INBRED LINE (RIL)
Mingje pool
Foar kwalitative trekken: 30 oant 50 yndividuen (minimaal 20) / bulk
Foar kwantitative tratis: Top 5% oant 10% yndividuen mei ekstreme fenotypes yn 'e heule befolking (minimaal 30 + 30).
Oanrikkemandearre sequencing djipte
Teminsten 20x / âlder en 1x / neiteam yndividu (bgl. Foar neigeslacht mingd pool fan 30 + 30 yndividu, sequencing djipte sil 30x per bulk wêze)
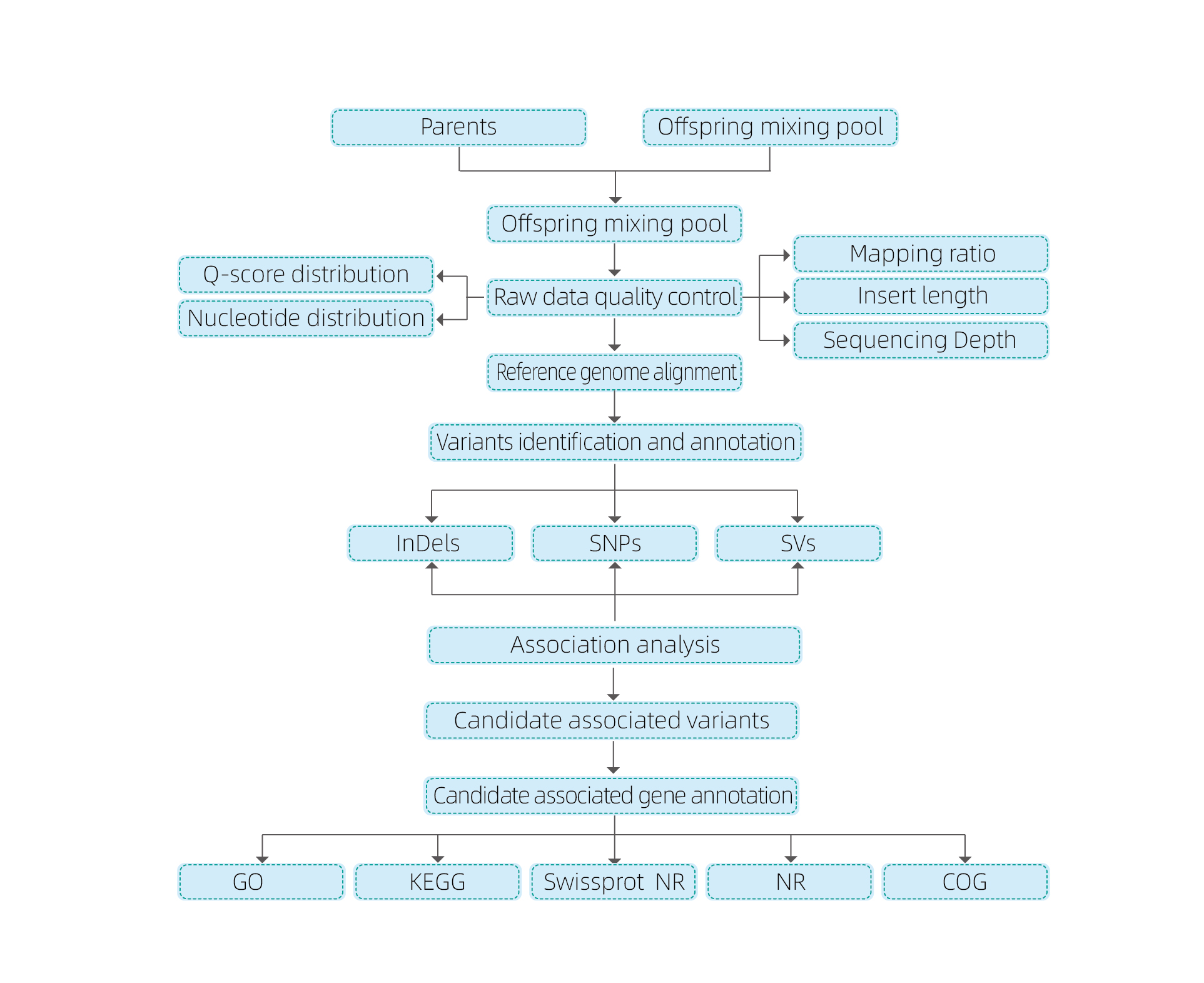
Bioinformatika analysen
● heule genume ringen
● Gegevensferwurking
● SNP / Indel skilje
● Kâncandairegio-screening
● annotaasje fan janidaat Gene Funksje

Sample-easken en levering
Foarbyldkeasken:
Nucleotiko's:
| GDna Sample | Tissue sample |
| Konsintraasje: ≥30 ng / μl | Planten: 1-2 G |
| Bedrach: ≥2 μg (volumn ≥15 μL) | Dieren: 0,5-1 g |
| Purity: OD260 / 280 = 1,6-2.5 | Heule bloed: 1,5 ml |
Tsjinstwurkstream

Eksperimintûntwerp

Sample levering

RNA-ekstraksje

Biblioteek konstruksje

Sequencing

Gegevensanalyse

Tsjinsten foar neisoarch
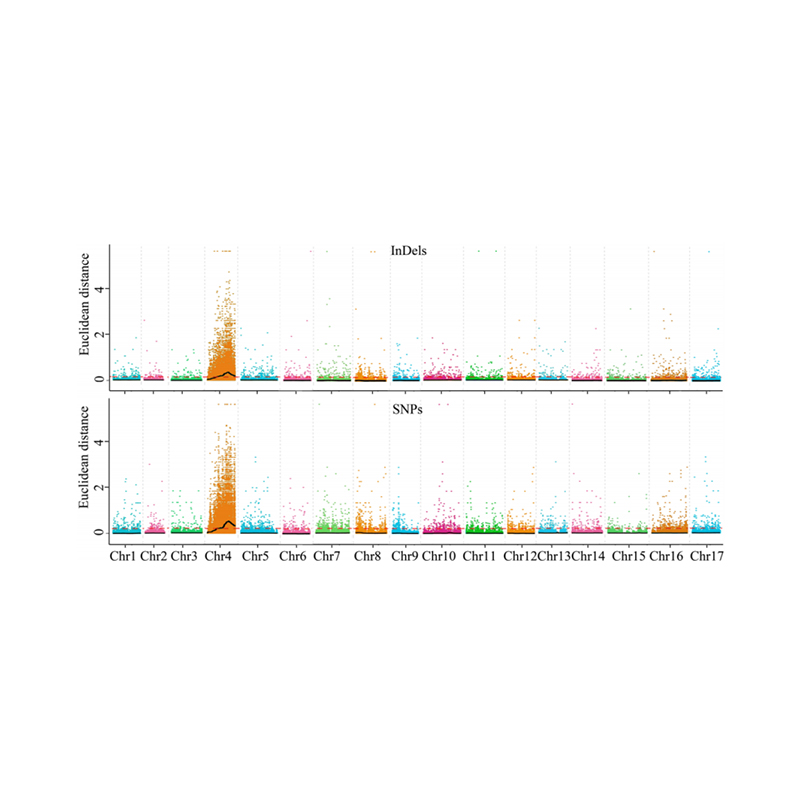
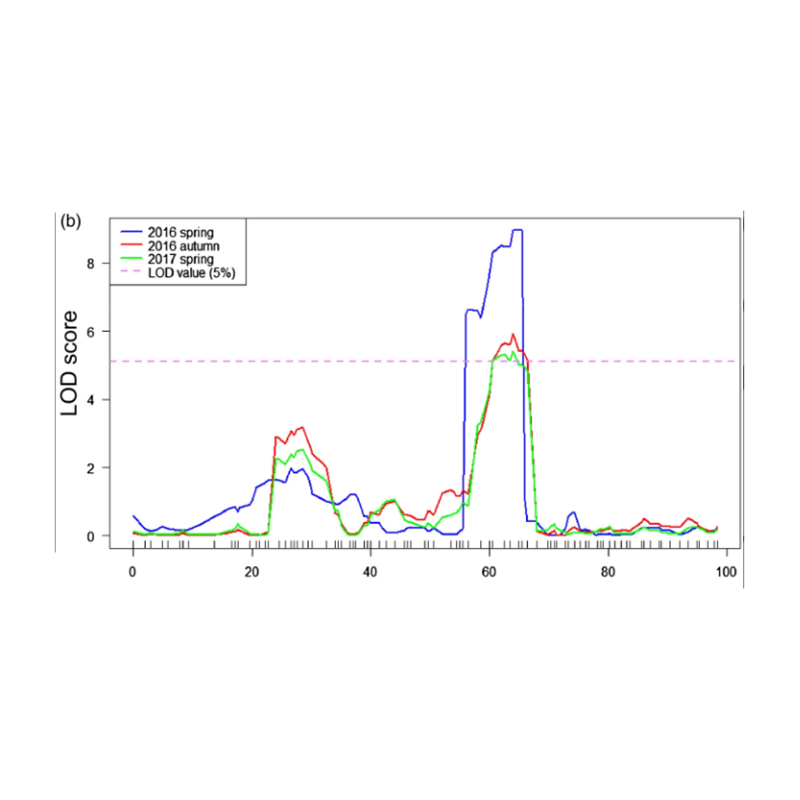
1.Smizing Analysay Base op Euclidean Ofstân (Ed) om kandidaatregio te identifisearjen. Yn 'e folgjende figuer
X-AXIS: Chromosome getal; Elke stip fertsjintwurdiget in edwearde fan in SNP. De swarte line oerienkomt mei ynrjochte edwearde. In hegere ED-wearde tsjut op in mear wichtige feriening tusken de side en it fenotype. Red Dash-line fertsjintwurdiget drompel fan wichtige feriening.

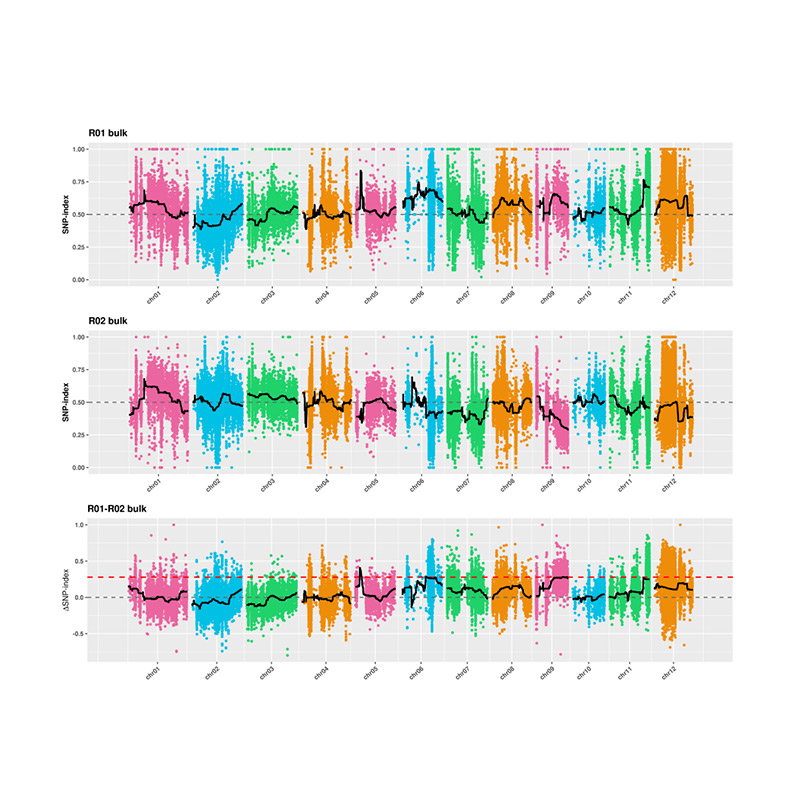
2.Sassociation Analyse basearre gjin SNP-yndeks
X-AXIS: Chromosome getal; Elke diem fertsjintwurdiget SNP-yndekswearde. De swarte line stiet foar ynrjochte SNP-yndekswearde. De gruttere de wearde is, hoe mear wichtige de feriening is.

BMK Saak
It haadritative trait-proto-effektive FNL7.1 Encodeart in lette embryogenesis oerfloedige proteïne assosjeare mei frjemde hals yn komkommer
Publisearre: Plant Biotechnology Journal, 2020
SEQUENCING STREATION:
Âlden (jin5-508, yn): heule gesichte resequencing foar 34 × en 20 ×.
DNA swimbaden (50 Long-halsed en 50 koarte-hals): RJOCHTEN FOAR 61 × en 52 ×
Kiesresultaten
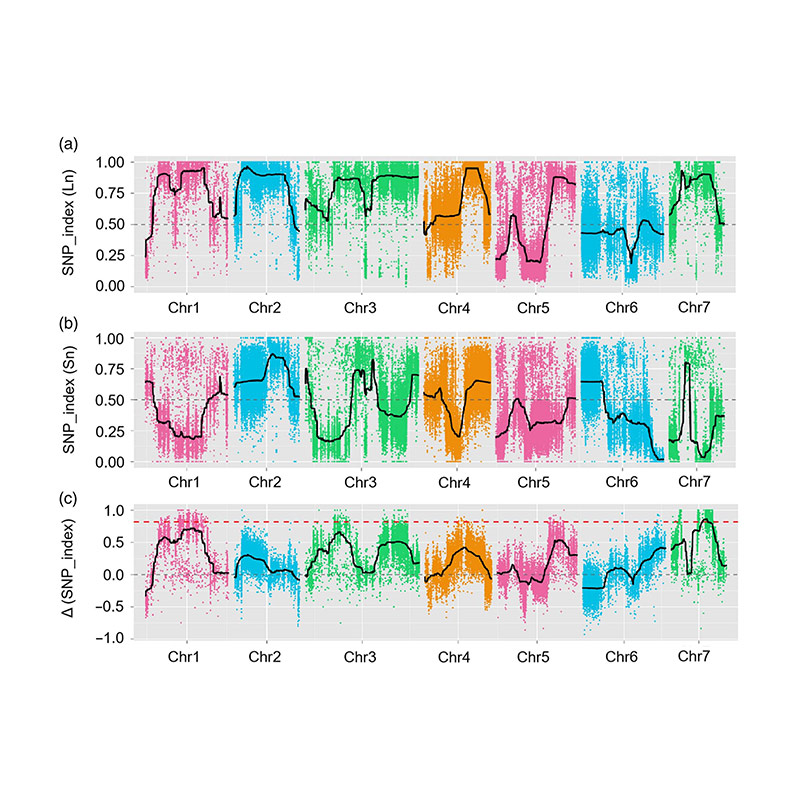
Yn dizze stúdzje waard populaasje (F2 en F2: 3 segreare (F2 en F2: 3) generearre troch oerstekke KUCUMPLIKE JIN5-508 en koarte nring yn. Twa DNA-swimbaden waarden konstruearre troch 50 ekstreme lange-hals yndividuïnten en 50 ekstreme koarte-hals yndividuen. MAJOR-EFFECT QTL IS IDENTIFIED OP CHR07 BY BSA ANALYSE EN BURDISJONEAL EANSTAL SUPL MAPP. De kandidaatregio waard fierder smelte troch FINE-MAPPEN, GENE-útdrukking kwantifikaasje en transgenyske eksperiminten, dy't toetsegen iepenbiere hawwe yn kontrolearjende nekke-lingte, CSFNL7.1. Derneist waard polymorphisme yn CSFNL7.1 Promoter-regio fûn te assosjearjen mei korrespondearjende útdrukking. Fierdere Phylogenetyske analyze suggereare dat fnl7.1 locus tige wierskynlik is ûntstien út Yndia.
 QTL-Mapping yn BSA-analyse om kandidaatregio te identifisearjen ferbûn mei lingte fan komkommer hal |  LOD-profilen fan komkommer hals-lingte QTL identifisearre op chr07 |
Xu, X., et al. "It MAID-EFTIPITATIVE TREEPITATIVE PREITUS FNL7.1 ENCODEN A LATE Embryogene Eignt Affect Protein Associated met fruit nekke Lengte yn komkommer." Plant Biotechnology Journal 18.7 (2020).