-

Séquençage métagénomique -NGS
Un métagénome est une collection du matériel génétique total d’une communauté mixte d’organismes, tels que les métagénomes environnementaux et humains. Il contient des génomes de micro-organismes cultivables et incultivables. Le séquençage métagénomique Shotgun avec NGS permet l’étude de ces paysages génomiques complexes intégrés dans des échantillons environnementaux en fournissant plus qu’un profil taxonomique, donnant également des informations granulaires sur la diversité des espèces, la dynamique de l’abondance et les structures complexes des populations. Au-delà des études taxonomiques, la métagénomique de chasse offre également une perspective de génomique fonctionnelle, permettant l'exploration des gènes codés et de leurs rôles putatifs dans les processus écologiques. Enfin, l'établissement de réseaux de corrélation entre les éléments génétiques et les facteurs environnementaux contribue à une compréhension holistique de l'interaction complexe entre les communautés microbiennes et leur contexte écologique. En conclusion, le séquençage métagénomique constitue un instrument essentiel pour démêler les subtilités génomiques de diverses communautés microbiennes, éclairant les relations multiformes entre la génétique et l’écologie au sein de ces écosystèmes complexes.
Plateformes : Illumina NovaSeq et DNBSEQ-T7
-

Séquençage métagénomique-TGS
Un métagénome est une collection de matériel génétique d'une communauté mixte d'organismes, tels que les métagénomes environnementaux et humains. Il contient des génomes de micro-organismes cultivables et incultivables. Le séquençage métagénomique permet l’étude de ces paysages génomiques complexes intégrés dans des échantillons écologiques en fournissant plus qu’un simple profilage taxonomique. Il offre également une perspective de génomique fonctionnelle en explorant les gènes codés et leurs rôles putatifs dans les processus environnementaux. Alors que les approches traditionnelles au fusil de chasse avec séquençage Illumina ont été largement utilisées dans les études métagénomiques, l’avènement du séquençage à lecture longue Nanopore et PacBio a changé le domaine. Les technologies Nanopore et PacBio améliorent les analyses bioinformatiques en aval, notamment l'assemblage du métagénome, garantissant des assemblages plus continus. Les rapports indiquent que la métagénomique basée sur Nanopore et PacBio a généré avec succès des génomes bactériens complets et fermés à partir de microbiomes complexes (Moss, EL, et al., Nature Biotech, 2020). L'intégration des lectures Nanopore avec les lectures Illumina fournit une approche stratégique pour la correction des erreurs, atténuant la faible précision inhérente de Nanopore. Cette combinaison synergique exploite les atouts de chaque plateforme de séquençage, offrant une solution robuste pour surmonter les limitations potentielles et faire progresser la précision et la fiabilité des analyses métagénomiques.
Plateforme : Nanopore PromethION 48, Illumia et PacBio Revio
-

Séquençage bisulfite du génome entier (WGBS)
Le séquençage au bisulfite du génome entier (WGBS) constitue la méthodologie de référence pour l'exploration approfondie de la méthylation de l'ADN, en particulier la cinquième position dans la cytosine (5-mC), un régulateur essentiel de l'expression des gènes et de l'activité cellulaire. Le principe du WGBS implique un traitement au bisulfite, induisant la conversion des cytosines non méthylées en uracile (C en U), tout en laissant les cytosines méthylées inchangées. Cette technique offre une résolution à base unique, permettant aux chercheurs d'étudier de manière approfondie le méthylome et de découvrir des modèles de méthylation anormaux associés à diverses affections, notamment le cancer. En utilisant le WGBS, les scientifiques peuvent acquérir des informations sans précédent sur les paysages de méthylation à l’échelle du génome, offrant ainsi une compréhension nuancée des mécanismes épigénétiques qui sont à la base de divers processus biologiques et maladies.
-

Test de chromatine accessible à la transposase avec séquençage à haut débit (ATAC-seq)
ATAC-seq est une technique de séquençage à haut débit utilisée pour l'analyse de l'accessibilité de la chromatine à l'échelle du génome. Son utilisation permet de mieux comprendre les mécanismes complexes du contrôle épigénétique global sur l’expression des gènes. La méthode utilise une transposase Tn5 hyperactive pour fragmenter et marquer simultanément les régions ouvertes de la chromatine en insérant des adaptateurs de séquençage. L'amplification PCR ultérieure aboutit à la création d'une bibliothèque de séquençage, qui permet l'identification complète des régions ouvertes de la chromatine dans des conditions spatio-temporelles spécifiques. ATAC-seq fournit une vue holistique des paysages chromatiniens accessibles, contrairement aux méthodes qui se concentrent uniquement sur les sites de liaison aux facteurs de transcription ou sur des régions spécifiques modifiées par des histones. En séquençant ces régions ouvertes de chromatine, ATAC-seq révèle des régions plus susceptibles d'activer des séquences régulatrices et des sites de liaison potentiels aux facteurs de transcription, offrant ainsi des informations précieuses sur la modulation dynamique de l'expression des gènes à travers le génome.
-

Séquençage d'amplicons 16S/18S/ITS-PacBio
Les gènes d'ARNr 16S et 18S, ainsi que la région de l'espaceur transcrit interne (ITS), servent de marqueurs d'empreintes moléculaires essentiels en raison de leur combinaison de régions hautement conservées et hyper-variables, ce qui en fait des outils précieux pour caractériser les organismes procaryotes et eucaryotes. L'amplification et le séquençage de ces régions offrent une approche sans isolement pour étudier la composition et la diversité microbiennes dans divers écosystèmes. Alors que le séquençage d'Illumina cible généralement de courtes régions hypervariables telles que V3-V4 de 16S et ITS1, il a été démontré qu'une annotation taxonomique supérieure est réalisable en séquençant la longueur totale de 16S, 18S et ITS. Cette approche globale aboutit à des pourcentages plus élevés de séquences classées avec précision, atteignant un niveau de résolution qui s'étend à l'identification des espèces. La plate-forme de séquençage en temps réel à molécule unique (SMRT) de PacBio se distingue en fournissant des lectures longues (HiFi) très précises qui couvrent les amplicons complets, rivalisant avec la précision du séquençage Illumina. Cette capacité permet aux chercheurs d’obtenir un avantage inégalé : une vue panoramique du paysage génétique. La couverture étendue augmente considérablement la résolution de l'annotation des espèces, en particulier au sein des communautés bactériennes ou fongiques, permettant une compréhension plus approfondie des subtilités des populations microbiennes.
-

Séquençage d'amplicons 16S/18S/ITS-NGS
Le séquençage d'amplicons avec la technologie Illumina, ciblant spécifiquement les marqueurs génétiques 16S, 18S et ITS, est une méthode puissante pour découvrir la phylogénie, la taxonomie et l'abondance des espèces au sein des communautés microbiennes. Cette approche implique le séquençage des régions hypervariables des marqueurs génétiques domestiques. Initialement introduit comme une empreinte moléculaire parMalheurs et alen 1977, cette technique a révolutionné le profilage du microbiome en permettant des analyses sans isolement. Grâce au séquençage du 16S (bactéries), du 18S (champignons) et de l'espaceur transcrit interne (ITS, champignons), les chercheurs peuvent identifier non seulement des espèces abondantes, mais également des espèces rares et non identifiées. Largement adopté comme outil essentiel, le séquençage des amplicons est devenu déterminant pour discerner les compositions microbiennes différentielles dans divers environnements, notamment la bouche humaine, les intestins, les selles et au-delà.
-

Re-séquençage du génome entier bactérien et fongique
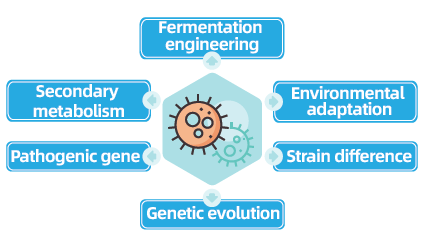
Les projets de re-séquençage du génome entier bactérien et fongique sont essentiels pour faire progresser la génomique microbienne en permettant la complétion et la comparaison des génomes microbiens. Cela facilite l’ingénierie de la fermentation, l’optimisation des processus industriels et l’exploration des voies métaboliques secondaires. En outre, le re-séquençage fongique et bactérien est crucial pour comprendre l’adaptation environnementale, optimiser les souches et révéler la dynamique de l’évolution génétique, avec de vastes implications en médecine, en agriculture et en sciences de l’environnement.
-

Séquençage d'amplicons 16S/18S/ITS pleine longueur PacBio
La plateforme Amplicon (16S/18S/ITS) est développée avec des années d'expérience dans l'analyse de projets de diversité microbienne, qui contient une analyse de base standardisée et une analyse personnalisée : l'analyse de base couvre le contenu d'analyse principal de la recherche microbienne actuelle, le contenu de l'analyse est riche et complet, et les résultats de l'analyse sont présentés sous forme de rapports de projet ; Le contenu de l’analyse personnalisée est diversifié. Les échantillons peuvent être sélectionnés et les paramètres peuvent être définis de manière flexible en fonction du rapport d'analyse de base et de l'objectif de recherche, pour répondre à des exigences personnalisées. Système d'exploitation Windows, simple et rapide.
-

PacBio-Transcriptome complet (non-référence)
En prenant comme entrée les données de séquençage des isoformes de Pacific Biosciences (PacBio), cette application est capable d'identifier des séquences de transcription complètes (sans assemblage). En cartographiant des séquences complètes par rapport au génome de référence, les transcrits peuvent être optimisés par des gènes, des transcrits, des régions codantes connus, etc. Dans ce cas, une identification plus précise des structures d'ARNm, telles qu'un épissage alternatif, etc., peut être obtenue. L'analyse conjointe avec les données de séquençage du transcriptome NGS permet une annotation plus complète et une quantification plus précise de l'expression au niveau de la transcription, ce qui profite largement à l'expression différentielle et à l'analyse fonctionnelle en aval.
-

Séquençage du bisulfite à représentation réduite (RRBS)
Le séquençage au bisulfite à représentation réduite (RRBS) est apparu comme une alternative rentable et efficace au séquençage au bisulfite du génome entier (WGBS) dans la recherche sur la méthylation de l'ADN. Bien que le WGBS fournisse des informations complètes en examinant l’intégralité du génome avec une résolution de base unique, son coût élevé peut être un facteur limitant. RRBS atténue stratégiquement ce défi en analysant sélectivement une partie représentative du génome. Cette méthodologie repose sur l'enrichissement des régions riches en îlots CpG par clivage MspI suivi d'une sélection de taille de fragments de 200 à 500/600 pb. Par conséquent, seules les régions proches des îlots CpG sont séquencées, tandis que celles comportant des îlots CpG éloignés sont exclues de l’analyse. Ce processus, combiné au séquençage au bisulfite, permet une détection haute résolution de la méthylation de l'ADN, et l'approche de séquençage PE150 se concentre spécifiquement sur les extrémités des inserts plutôt que sur le milieu, augmentant ainsi l'efficacité du profilage de la méthylation. Le RRBS est un outil précieux qui permet une recherche rentable sur la méthylation de l’ADN et fait progresser la connaissance des mécanismes épigénétiques.
-

Séquençage de l'ARN procaryote
Le séquençage de l’ARN permet le profilage complet de tous les transcrits d’ARN dans les cellules dans des conditions spécifiques. Cette technologie de pointe constitue un outil puissant, dévoilant des profils complexes d’expression génique, des structures génétiques et des mécanismes moléculaires associés à divers processus biologiques. Largement adopté dans la recherche fondamentale, les diagnostics cliniques et le développement de médicaments, le séquençage de l’ARN offre un aperçu des subtilités de la dynamique cellulaire et de la régulation génétique. Notre traitement des échantillons d’ARN procaryotes est adapté aux transcriptomes procaryotes, impliquant une déplétion de l’ARNr et une préparation directionnelle de bibliothèques.
Plateforme : Illumina NovaSeq
-

Séquençage du métatranscriptome
Tirant parti de la technologie de séquençage d'Illumina, le service de séquençage du métatranscriptome de BMKGENE dévoile l'expression dynamique des gènes d'un large éventail de microbes, allant des eucaryotes aux procaryotes et aux virus, dans des environnements naturels tels que le sol, l'eau, la mer, les selles et l'intestin. Notre service complet permet aux chercheurs d’approfondir les profils complets d’expression génique des communautés microbiennes complexes. Au-delà de l’analyse taxonomique, notre service de séquençage du métatranscriptome facilite l’exploration de l’enrichissement fonctionnel, mettant en lumière les gènes différentiellement exprimés et leurs rôles. Découvrez une richesse d'informations biologiques en parcourant les paysages complexes de l'expression des gènes, de la diversité taxonomique et de la dynamique fonctionnelle au sein de ces diverses niches environnementales.
-

Assemblage du génome fongique de novo
BMKGENE propose des solutions polyvalentes pour les génomes fongiques, répondant à divers besoins de recherche et à l'exhaustivité souhaitée du génome. L’utilisation du séquençage Illumina à lecture courte permet à elle seule de générer un projet de génome. Le séquençage à lecture courte et à lecture longue à l'aide de Nanopore ou Pacbio est combiné pour un génome fongique plus raffiné avec des contigs plus longs. De plus, l’intégration du séquençage Hi-C améliore encore les capacités, permettant d’obtenir un génome complet au niveau des chromosomes.