Séquençage d'ARNm non références-NGS
Caractéristiques
● Capture d'ARNm poly avant la préparation de la bibliothèque
● Indépendamment de tout génome de référence: basé sur l'assemblage de transcriptions de novo, générant une liste d'unigènes qui sont annotés avec plusieurs bases de données (NR, Swiss-Prot, Cog, Kog, Eggnog, PFAM, GO, KEGG)
● Analyse bioinformatique complète de l'expression des gènes et de la structure des transcrits
Avantages de service
●Expertise approfondie: Avec un historique de traitement de plus de 600 000 échantillons chez BMKGene, couvrant divers types d'échantillons tels que les cultures cellulaires, les tissus et les fluides corporels, notre équipe apporte une richesse d'expérience à chaque projet. Nous avons réussi à clôturer plus de 100 000 projets d'ARNm-Seq dans divers domaines de recherche.
●Contrôle de la qualité rigoureux: Nous mettons en œuvre des points de contrôle principaux à toutes les étapes, de la préparation des échantillons et de la bibliothèque au séquençage et à la bioinformatique. Cette surveillance méticuleuse garantit la livraison de résultats de haute qualité toujours de haute qualité.
● Annotation complète: Nous utilisons plusieurs bases de données pour annoter fonctionnellement les gènes exprimés différentiellement (DEG) et effectuer l'analyse d'enrichissement correspondante, fournissant des informations sur les processus cellulaires et moléculaires sous-jacents à la réponse du transcriptome.
●Support post-vente: Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant ce temps, nous offrons un suivi du projet, une assistance de dépannage et des séances de questions-réponses pour répondre à toutes les requêtes liées aux résultats.
Exemples d'exigences et de livraison
| Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| Poly A enrichi | Illumina PE150 DNBSEQ-T7 | 6-10 Go | Q30 ≥ 85% |
Exemples d'exigences:
Nucléotides:
| Conc. (Ng / μl) | Quantité (μg) | Pureté | Intégrité |
| ≥ 10 | ≥ 0,2 | OD260 / 280 = 1,7-2,5 OD260 / 230 = 0,5-2,5 Contamination limitée ou non protéique ou ADN montré sur le gel. | Pour les plantes: RIN ≥ 4,0; Pour les animaux: rin ≥ 4,5; 5.0≥28s / 18S ≥ 1,0; Élévation limitée ou non de référence |
● Plantes:
Racine, tige ou pétale: 450 mg
Feuille ou graine: 300 mg
Fruit: 1,2 g
● Animal:
Cœur ou intestin: 300 mg
Viscères ou cerveau: 240 mg
Muscle: 450 mg
Os, cheveux ou peau: 1g
● Arthropodes:
Insectes: 6G
Crustacea: 300 mg
● sang total: 1 tube
● Cellules: 106 cellules
Livraison d'échantillon recommandée
Récipient: 2 ml de tube à centrifugeuse (la papier d'étain n'est pas recommandée)
Exemple d'étiquetage: groupe + réplique par exemple A1, A2, A3; B1, B2, B3.
Expédition:
1. Face sèche: les échantillons doivent être emballés dans des sacs et enterrés dans la glace sèche.
2. Tubes raastables: les échantillons d'ARN peuvent être séchés dans un tube de stabilisation de l'ARN (par exemple RNastable®) et expédiés à température ambiante.
Flux de travail de service

Conception d'expérience

Livraison d'échantillons

Extraction de l'ARN

Construction de la bibliothèque

Séquençage

Analyse des données

Services après-vente
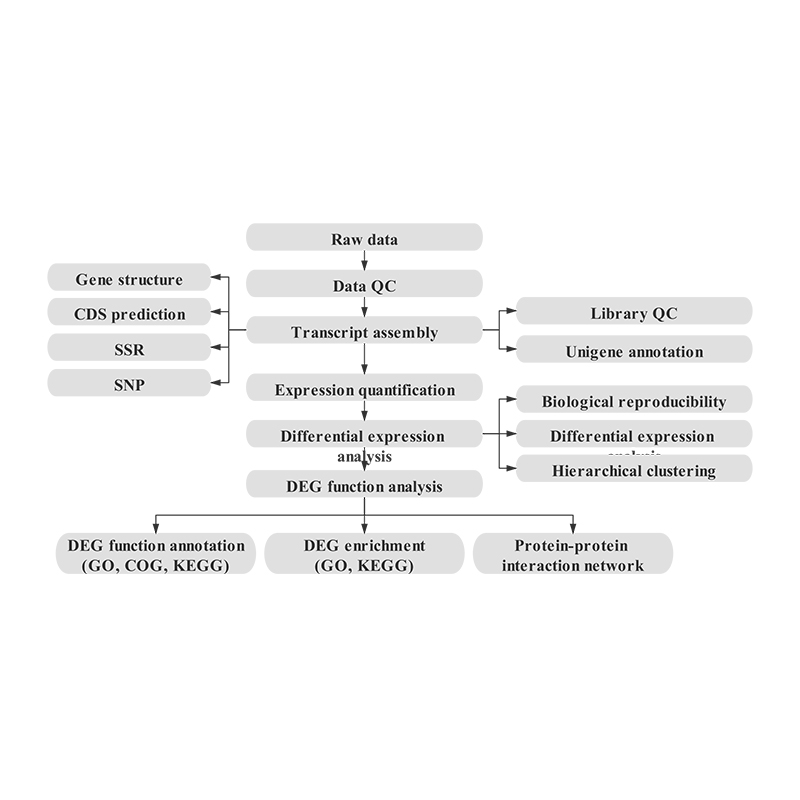
Bioinformatique
Assemblage du transcriptome et sélection unigène
Annotation unigène
Corrélation d'échantillons et évaluation des répliques biologiques
Gènes exprimés différentiellement (DEG)
Annotation fonctionnelle des DEG
Enrichissement fonctionnel des DEG
Explorez les progrès facilités par les services de séquençage de l'ARNm eucaryotique de BMKGene NGS à travers une collection organisée de publications.
Shen, F. et al. (2020) «De novo Transcriptome Assembly and Sexed Gene Expression in the Gonads of Amur Catfish (Silurus asotus)», Genomics, 112 (3), pp. 2603-2614. doi: 10.1016 / j.ygeno.2020.01.026.
Zhang, C. et al. (2016) «Analyse du transcriptome du métabolisme du saccharose pendant le gonflement et le développement des ampoules dans l'oignon (Allium cepa L.)», Frontiers in Plant Science, 7 (septembre), p. 212763. Doi: 10.3389 / fpl.2016.01425 / bibtex.
Zhu, C. et al. (2017) «De novo Assembly, caractérisation et annotation pour le transcriptome de Sarcocheilichthys sinensis», PLoS ONE, 12 (2). doi: 10.1371 / journal.pone.0171966.
Zou, L. et al. (2021) «L'analyse du transcriptome de novo fournit un aperçu de la tolérance au sel de Podocarpus macrophyllus sous le stress de salinité», BMC Plant Biology, 21 (1), pp. 1–17. doi: 10.1186 / s12870-021-03274-1 / Figures / 9.