Génomique humaine
Génétique de la nature
Le séquençage à longue lecture identifie les extensions de répétition du GGC dans Notch2NLC associée à une maladie d'inclusion intranucléaire neuronale
Ressencement ONT | Illumina | Séquençage de l'exome entier | CRISPR-CAS9 ONT CIBLEED SQUENCING | RNA-seq | ONT 5MC Methylation Calling
Points forts
1. Par analyse de liaison sur une grande famille NIID, deux régions liées ont été identifiées.
2. Le séquençage à lecture longue basée sur et et le séquençage ONT médié par Cas-9 ont découvert une cause génétique potentielle de NIID, les extensions de répétition GGC dans 5 'UTR de Notch2NLC. Cette étude a signalé des extensions répétées dans les gènes spécifiques de l'homme pour la première fois qui ont évolué par des duplications segmentaires.
3. Le séquençage de l'ARNA a révélé des transcrits antisens anormaux au début ou à l'intérieur des régions d'expansion de répétition GGC dans Notch2NLC.
Arrière-plan
NLa maladie de l'inclusion intranucléaire euronale (NIID) est une maladie neurodégénérative progressive et mortelle, qui se caractérise par la présence d'inclusions intranucléaires hyalines éosinophiles dans les systèmes nerveux central et périphérique. Ses manifestations cliniques très variables augmentent de grandes difficultés de diagnostic jusqu'à l'introduction de la biopsie cutanée. Cependant, les méthodes basées sur l'histopathologie souffrent toujours d'un diagnostic erroné, ce qui appelle à une compréhension génétique de la NIID.
Réalisations
Analyse de liaison
SLe séquençage du génome entier (WGS) basé sur le séquençage à lecture HORT (WGS) et le séquençage entièrement exome (WES) ont été réalisés sur une grande famille NIID (13 membres affectés et 7 membres non affectés). L'analyse de liaison sur les SNP extraits de ces données n'a révélé que deux régions liées: une région de 3,5 Mo à 1p36.31-p36.22 (MAXIMUM LOD = 2,32) et une région de 58,1 Mb à 1p22.1-Q21.3 (LOD maximum: 4,21 ). Cependant, aucun SNP pathogène ou CNVS n'a été identifié dans ces régions liées.
Extensions de répétition GGC dans Notch2NLC
NLe séquençage basé sur Anopore a été traité sur 13 membres affectés et 4 membres non affectés de 8 familles (un autre membre affecté a été séquencé par la plate-forme de séquençage à longue lecture de PacBio.). Les données à lutte à lutte ont révélé des extensions de répétition GGC associées à la maladie dans le 5 'UTR de la cartographie du gène Notch2NLC à une région liée à 58,1 Mo (figure 1). Ces extensions de répétition ont également été identifiées dans les 40 cas de NIID sporadiques testés par RP-PCR.
CLe séquençage cible médié par AS-9 sur la plate-forme Nanopore a été utilisé pour obtenir une couverture en lecture plus élevée sur Notch2NLC Repeat (100 x-1 795 x). Ces séquences de consensus se sont bien convenues avec les résultats précédents sur les extensions de répétition GGC. De plus, les répétitions {(gga) n (ggc) n} n ont été identifiées comme un marqueur génétique potentiel pour le phénotype à faiblesse dominante (figure 2).
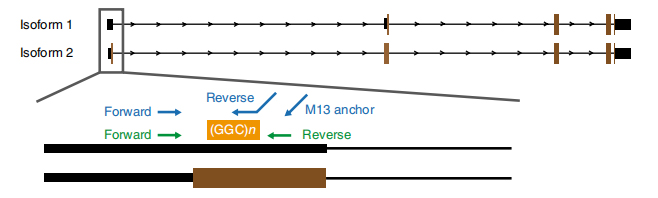
Figure 1. Expansion de répétition associée à la maladie identifiée sur l'exon 1 des isoformes Notch2NLC.
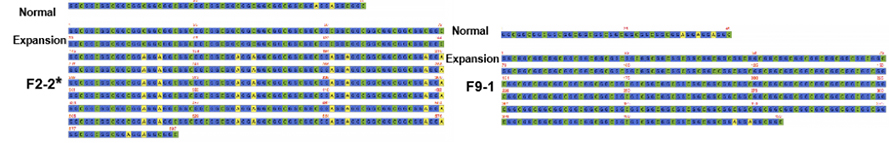
Figure 2. Séquences consensuelles de NPTCH2NLC Répétez chez les patients NIID avec (*) ou sans phénotype à dominante faiblesse
NLes gènes OPTH2NL sont des gènes spécifiques à l'homme, qui sont censés jouer un rôle vital dans l'évolution du cerveau humain et les maladies neurologiques. Cependant, trois gènes liés à Notch2 (Notch2NLA, Notch2NLB et Notch2NLC) avec> 99,1% d'identité de séquence n'ont été résolus que le dernier assemblage du génome humain. Le séquençage sans synthèse et longue lecture sur la plate-forme Nanopore a montré des avantages notables dans la résolution des régions de haute similitude et (GGC) N répète avec 100% de RICH GC.
Extensions de répétition GGC dans Notch2NLC
TLe séquençage du ranscriptome a été traité sur 2 membres affectés et 2 membres non affectés. La profondeur de lecture normalisée a été calculée sur les brins sens et antisens en amont des premiers exons de paralogues Notch2NL. Des transcrits anormaux anti-sens ont été trouvés uniquement dans les cas affectés, qui se trouvent au début ou à l'intérieur de la région d'expansion de répétition (pics violets en F1-14 et F1-16 sur la figure 3.). De plus, 54 degs ont été identifiés et tous ont été enrichis en termes GO et MPO liés aux fonctions neuronales.
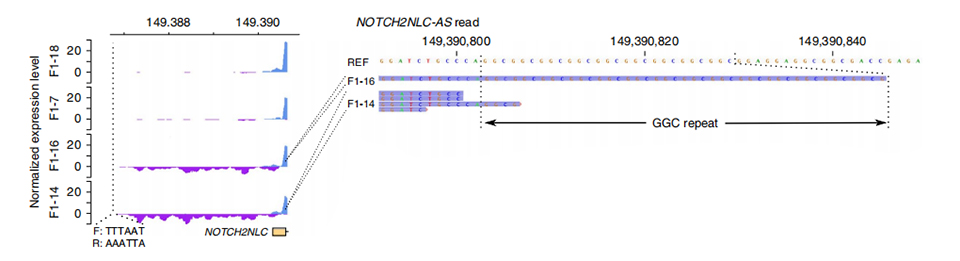
Figure 3. Profondeur de lecture normalisée en amont du premier exon de Notch2NLC dans les cas non affectés (ci-dessus) et affectés (ci-dessous).
Technologie
Oxford Nanopore Teghnologies (ONT)
NLe séquençage d'Anopore se distingue des autres plates-formes de séquençage, en ce que les nucléotides sont lus directement sans processus de synthèse d'ADN. Au fur et à mesure qu'un ADN à brin unique passe par un pore de protéines de taille nano-taille (nanopore), différents nucléotides génèrent différents courants ioniques, qui peuvent être capturés et transférés dans une séquence de bases. La plate-forme de séquençage ONT elle-même ne montre pas de limite technique apparente sur la longueur de la lecture de l'ADN. Par conséquent, des lectures ultra-longues (ULR) sont disponibles pour l'assemblage du génome de haute qualité. De plus, ces lectures extrêmement longues, qui sont suffisamment longues pour traverser les caractéristiques de séquence complexes ou la variation structurelle, aident à surmonter les limites du séquençage à lecture courte ici.

Séquençage de nanopore
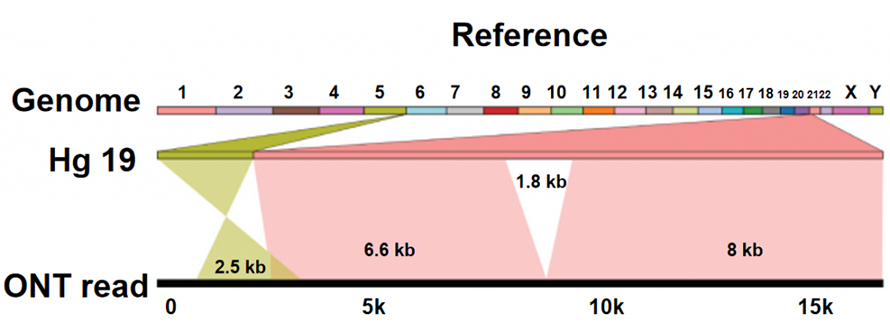
Identification de la variation de la structure (SV)
SInformations sur la méthylation de l'ADN sans préservation de la ynthesis sur le modèle. Methylé A, T, C et G génèrent des courants ioniques distincts de ceux non méthylés non méthylés, qui peuvent être lus directement par la plate-forme. Le séquençage des nanopores permet un profilage du génome entier de 5mc et 6MA à une résolution un seul nucléotide.
Référence
Jun sone, et. al. Le séquençage à longue lecture identifie les extensions de répétition du GGC dans Notch2NLC associée à une maladie d'inclusion intranucléaire neuronale. Nature Genetics (2019)
Tech et faits saillants vise à partager une application réussie la plus récente de différentes technologies de séquençage à haut débit dans divers arènes de redéachs ainsi que des idées brillantes dans la conception expérimentale et l'exploration de données.
Heure du poste: Jan-06-2022


