Séquençage de l’exome humain entier
Caractéristiques des services
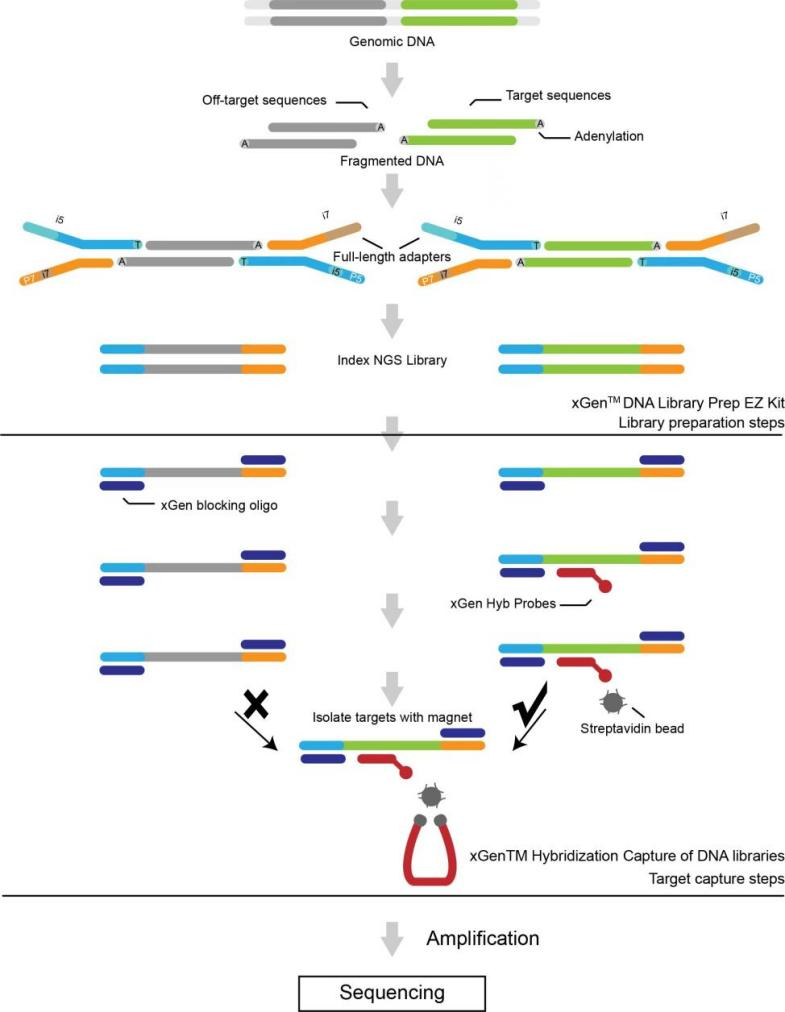
● Deux panels d'exome disponibles basés sur l'enrichissement de cibles avec des sondes : Sure Select Human All Exon v6 (Agilent) et xGen Exome Hybridization Panel v2 (IDT).
● Séquençage sur Illumina NovaSeq.
● Pipeline bioinformatique orienté vers l'analyse de maladies ou l'analyse de tumeurs.
Avantages des services
●Cible la région codante pour les protéines: En capturant et en séquençant les régions codantes pour les protéines, hWES est utilisé pour révéler des variantes liées à la structure des protéines.
●Rentable :Le hWES produit environ 85 % des mutations associées aux maladies humaines sur 1 % du génome humain.
●Haute précision: Avec une profondeur de séquençage élevée, hWES facilite la détection à la fois des variantes courantes et des variantes rares avec des fréquences inférieures à 1 %.
●Contrôle qualité rigoureux: Nous mettons en œuvre cinq points de contrôle principaux à toutes les étapes, de la préparation des échantillons et des bibliothèques au séquençage et à la bioinformatique. Ce suivi méticuleux garantit la fourniture de résultats constants de haute qualité.
●Analyse bioinformatique complète: notre pipeline va au-delà de l'identification des variations du génome de référence, car il intègre des analyses avancées conçues pour répondre spécifiquement aux questions de recherche liées aux aspects génétiques des maladies ou à l'analyse des tumeurs.
●Assistance après-vente :Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant cette période, nous proposons un suivi de projet, une assistance au dépannage et des séances de questions-réponses pour répondre à toute question liée aux résultats.
Exemples de spécifications
| Stratégie de capture d'exons | Stratégie de séquençage | Sortie de données recommandée |
| Bien sûr, sélectionnez Human All Exon v6 (Agilent) ou Panel d'hybridation xGen Exome v2 (IDT)
| Illumina NovaSeq PE150 | 5 à 10 Go Pour les troubles mendéliens/maladies rares : > 50x Pour les échantillons de tumeurs : ≥ 100x |
Exemples d'exigences
| Type d'échantillon
| Montant(Qubit®)
| Concentration | Volume
| Pureté (NanoDrop™) |
|
ADN génomique
| ≥ 50ng | ≥ 6 ng/μL | ≥ 15 µL | OD260/280=1,8-2,0 aucune dégradation, aucune contamination
|
Bioinformatique

L'analyse bioinformatique des échantillons de maladie hWES comprend :
● CQ des données de séquençage
● Alignement du génome de référence
● Identification des SNP et des InDels
● Annotation fonctionnelle des SNP et InDels

L'analyse bioinformatique des échantillons de tumeurs comprend :
● CQ des données de séquençage
● Alignement du génome de référence
● Identification des SNP, InDels et variations somatiques
● Identification des variantes germinales
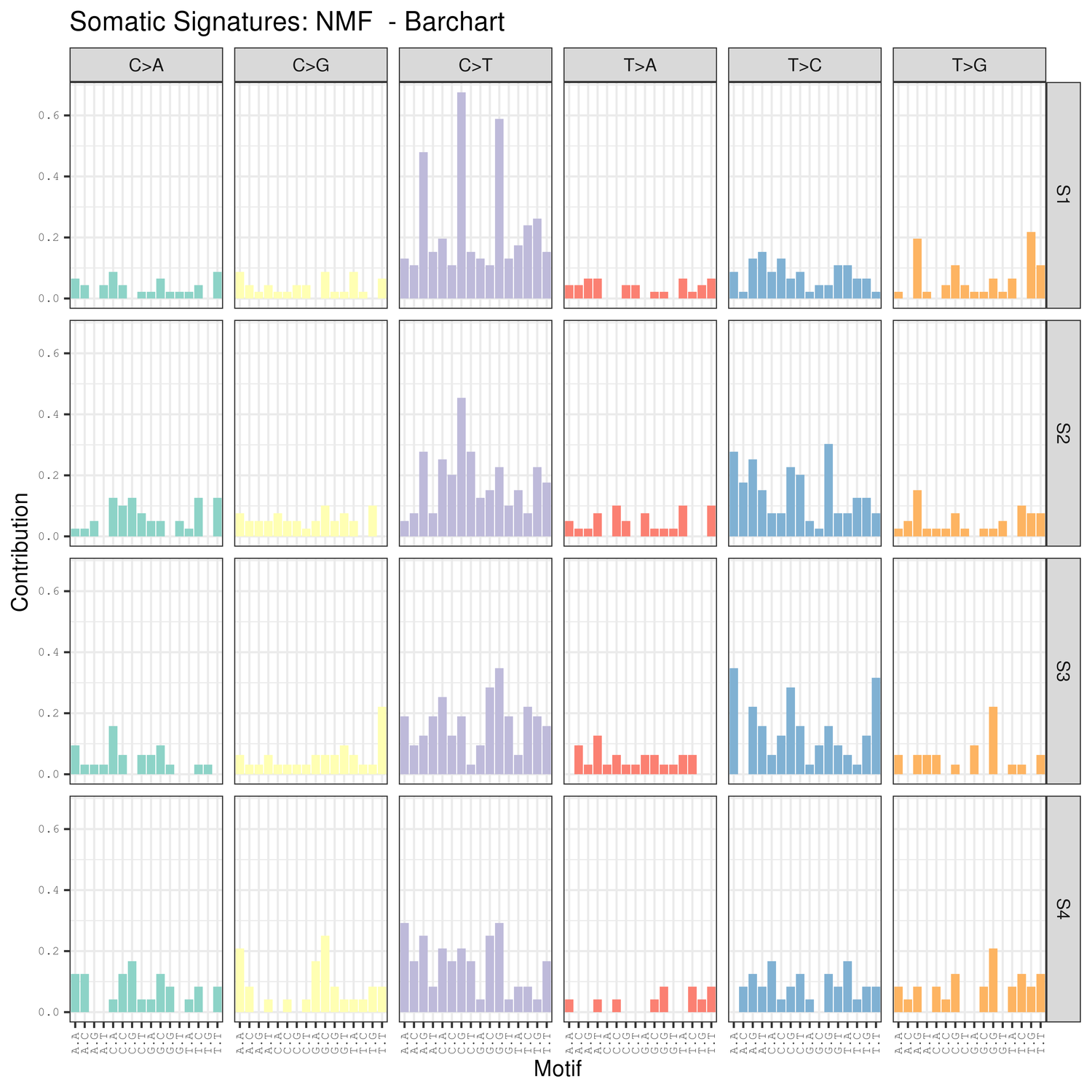
● Analyse des signatures de mutation
● Identification de gènes d'entraînement basée sur des mutations à gain de fonction
● Annotation des mutations au niveau de la sensibilité aux médicaments
● Analyse d'hétérogénéité – calcul de pureté et de ploïdie
Flux de travail des services

Livraison d'échantillon

Extraction d'ADN

Construction d'une bibliothèque

Séquençage

Analyse des données

Livraison des données

Services après-vente
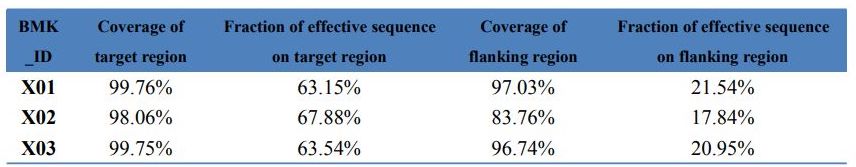
Data QC – Statistiques de capture Exome
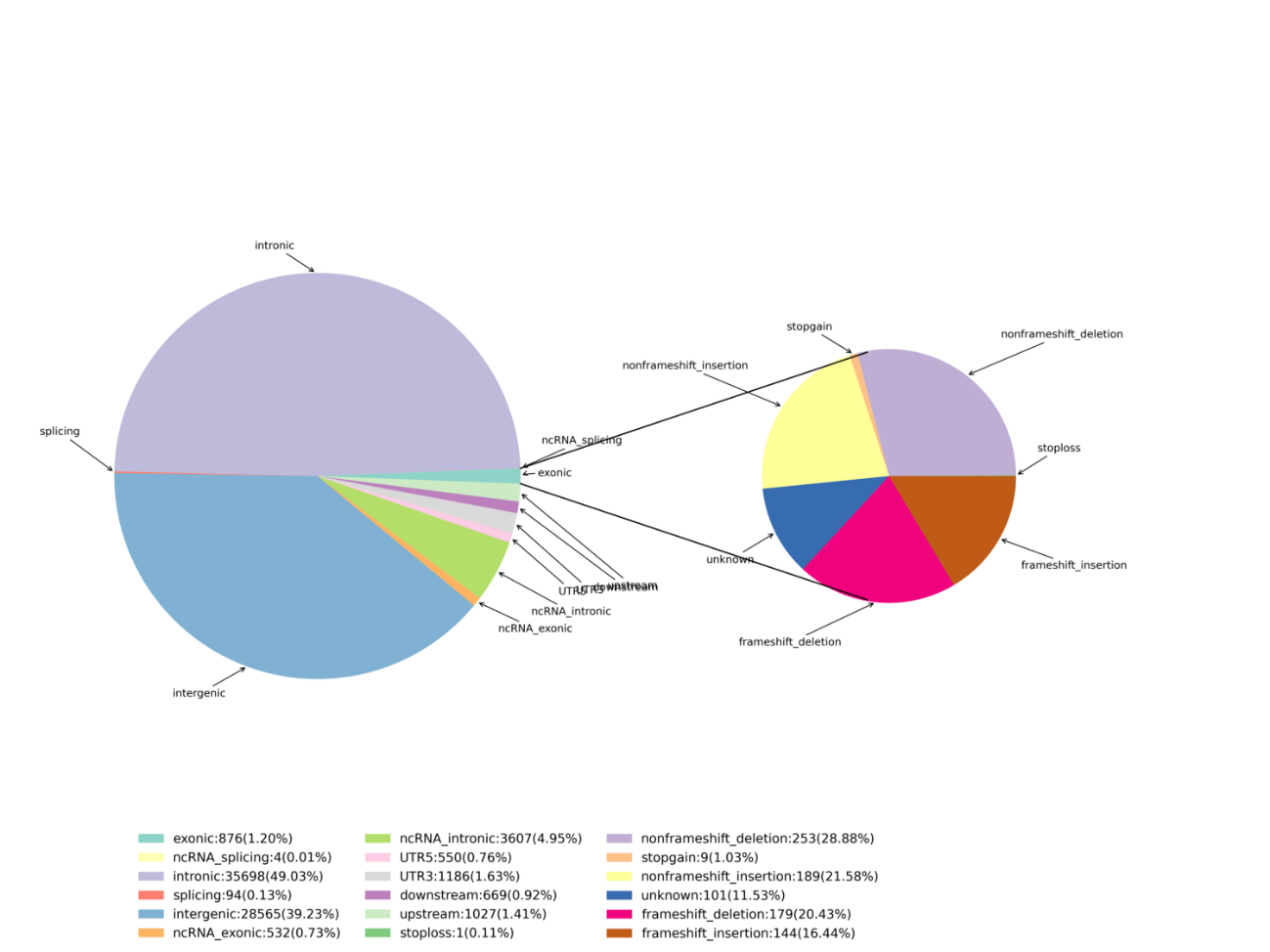
Identification des variantes – InDels
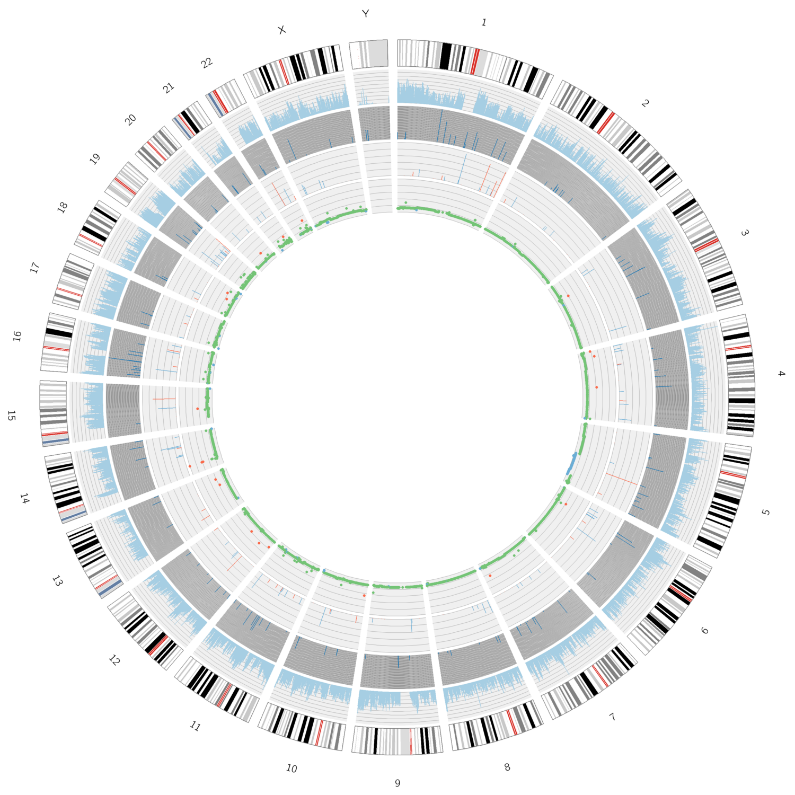
Analyse avancée : identification et distribution des SNP/InDels délétères – Tracé Circos
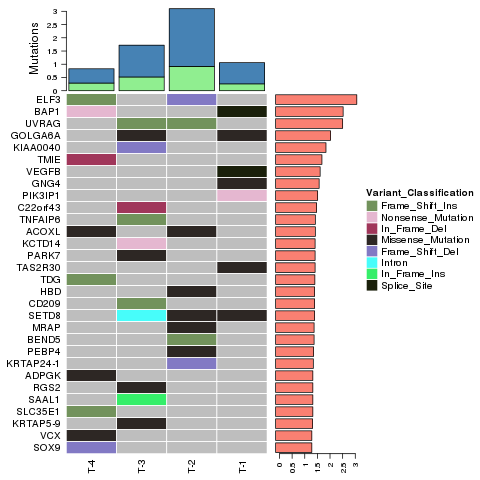
Analyse tumorale : identification et répartition des mutations somatiques – Tracé Circos
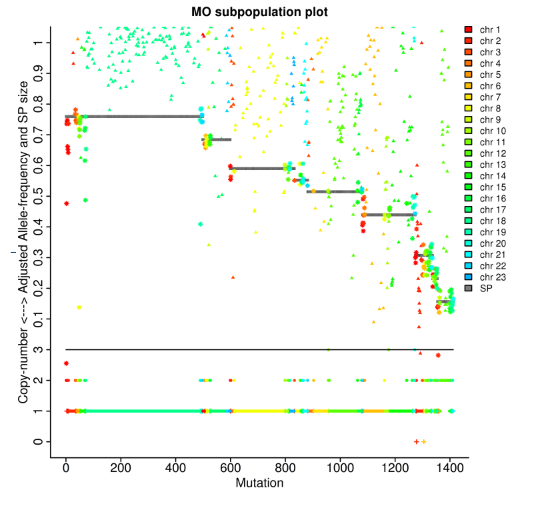
Analyse tumorale : lignées clonales