Assemblage du génome basé sur Hi-C
Fonctionnalités de service
● Séquençage sur Illumina Novaseq avec PE150.
● Le service nécessite des échantillons de tissus, au lieu des acides nucléiques extraits, pour rétiser le formaldéhyde et conserver les interactions ADN-protéine.
● L'expérience HI-C implique la restriction et la réparation finale des extrémités collantes par la biotine, suivie d'une circularisation des extrémités émoussées résultantes tout en préservant les interactions. L'ADN est ensuite abaissé avec des billes de streptavidine et purifié pour la préparation des bibliothèques ultérieures.
Avantages de service

Aperçu de Hi-C
(Lieberman-Aiden E et al.,Science, 2009)
●Éliminer le besoin de données de population génétique:Hi-C remplace les informations essentielles requises pour l'ancrage contig.
●Densité de marqueurs élevée:conduisant à un taux d'ancrage continu élevé supérieur à 90%.
●Expertise approfondie et dossiers de publication:BMKGene possède une vaste expérience avec plus de 2000 cas d'assemblage du génome Hi-C de 1000 espèces différentes et divers brevets. Plus de 200 cas publiés ont un facteur d'impact accumulatif de plus de 2000.
●Équipe de bioinformatique hautement qualifiée:Avec les brevets internes et les droits d'auteur de logiciels pour les expériences HI-C et l'analyse des données, le logiciel de données de visualisation auto-développé permet le déplacement, l'inversion, la révocation et la refonte manuels.
●Support post-vente:Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant ce temps, nous offrons un suivi du projet, une assistance de dépannage et des séances de questions-réponses pour répondre à toutes les requêtes liées aux résultats.
●Annotation complète: Nous utilisons plusieurs bases de données pour annoter fonctionnellement les gènes avec des variations identifiées et effectuer l'analyse d'enrichissement correspondante, fournissant des informations sur plusieurs projets de recherche.
Spécifications de service
| Préparation de bibliothèque | Stratégie de séquençage | Sortie de données recommandée | Contrôle de qualité |
| Bibliothèque Hi-C | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Exigences d'échantillonnage
| Tissu | Montant requis |
| Viscères animaux | ≥ 2 g |
| Muscle animal | |
| Sang de mammifère | ≥ 2 ml |
| Poultry / Fish Blood | |
| Plant- feuille fraîche | ≥ 3 g |
| Cellules cultivées | ≥ 1x107 |
| Insecte | ≥ 2 g |
Flux de travail de service

Conception d'expérience

Livraison d'échantillons

Construction de la bibliothèque

Séquençage

Analyse des données

Services après-vente
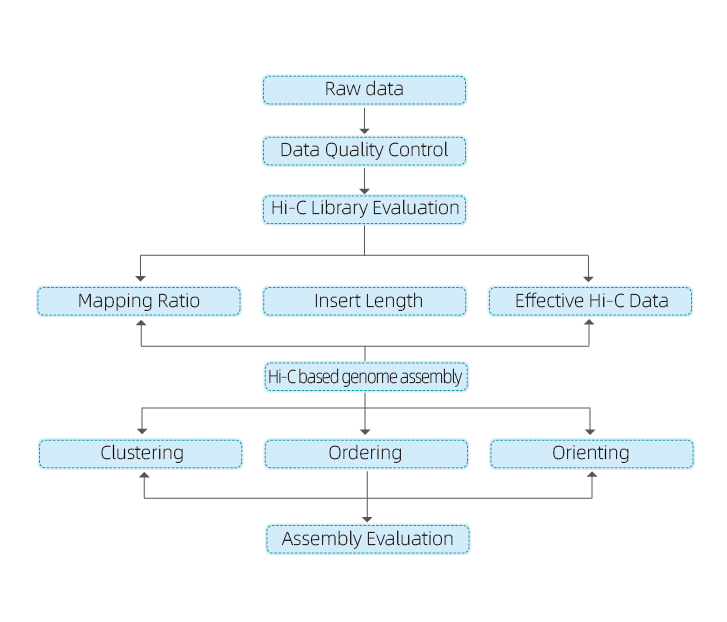
1) Données brutes QC
2) Hi-C Library QC: Estimation des interactions HI-C valides
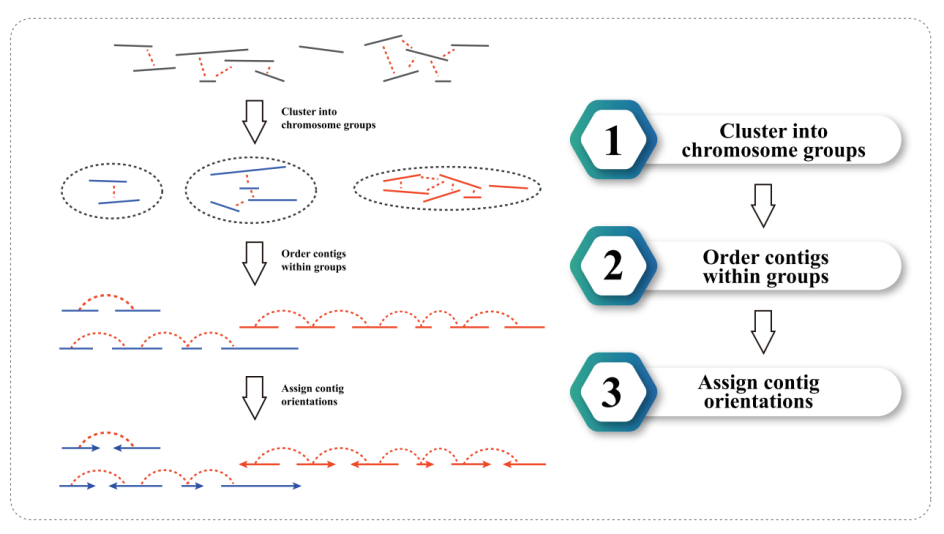
3) Assemblage Hi-C: regroupement des contigs en groupes, suivi de l'ordre contig dans chaque groupe et attribuant l'orientation contig
4) Évaluation Hi-C
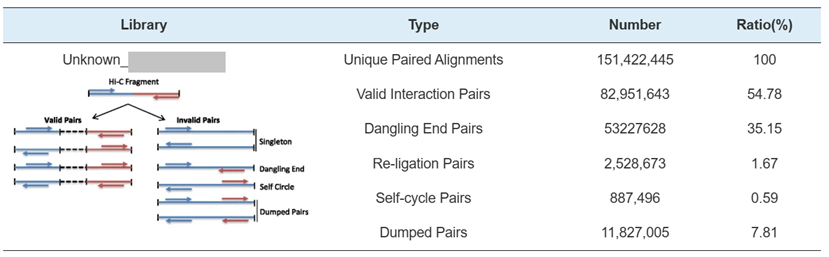
Hi-C Library QC - Estimation des paires d'interaction valides Hi-C
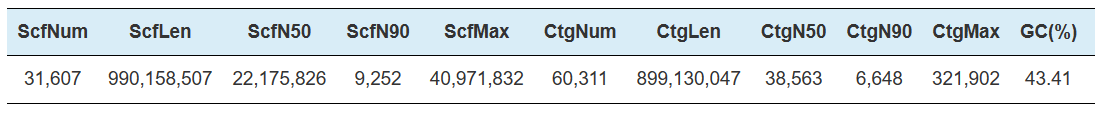
Assemblée HI-C - Statistiques
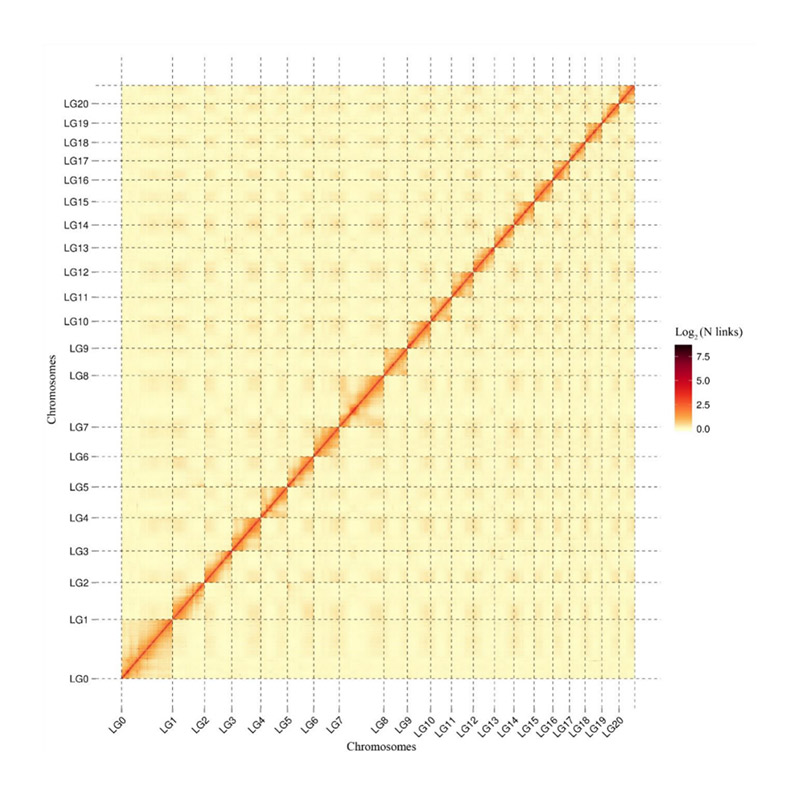
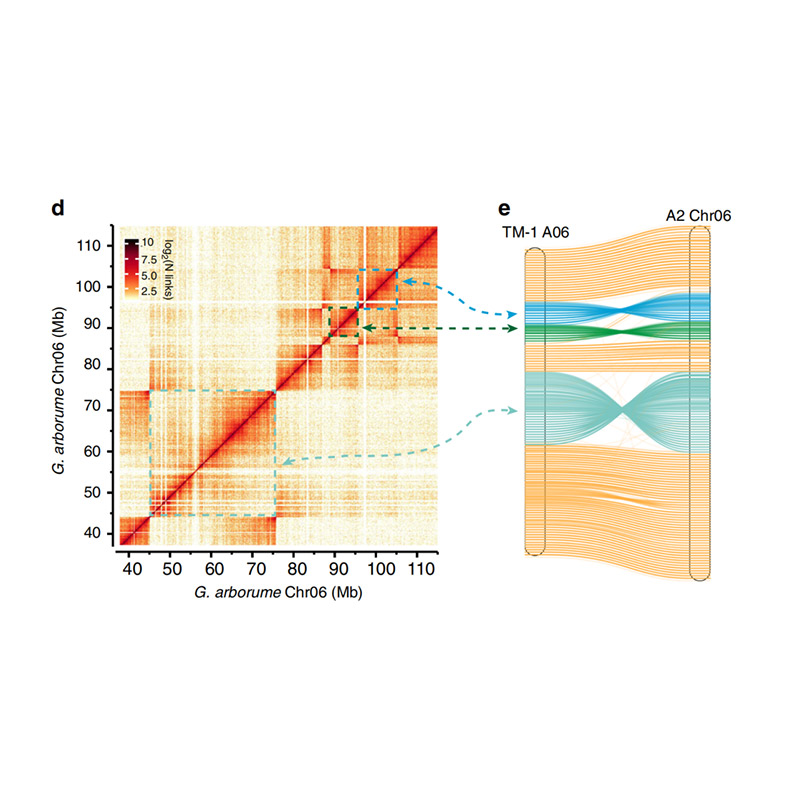
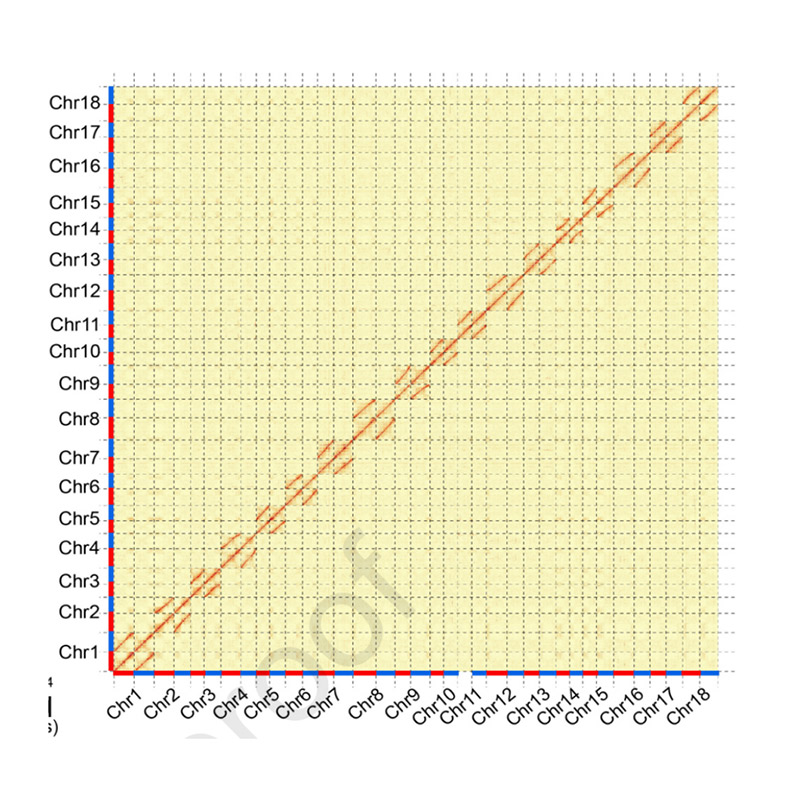
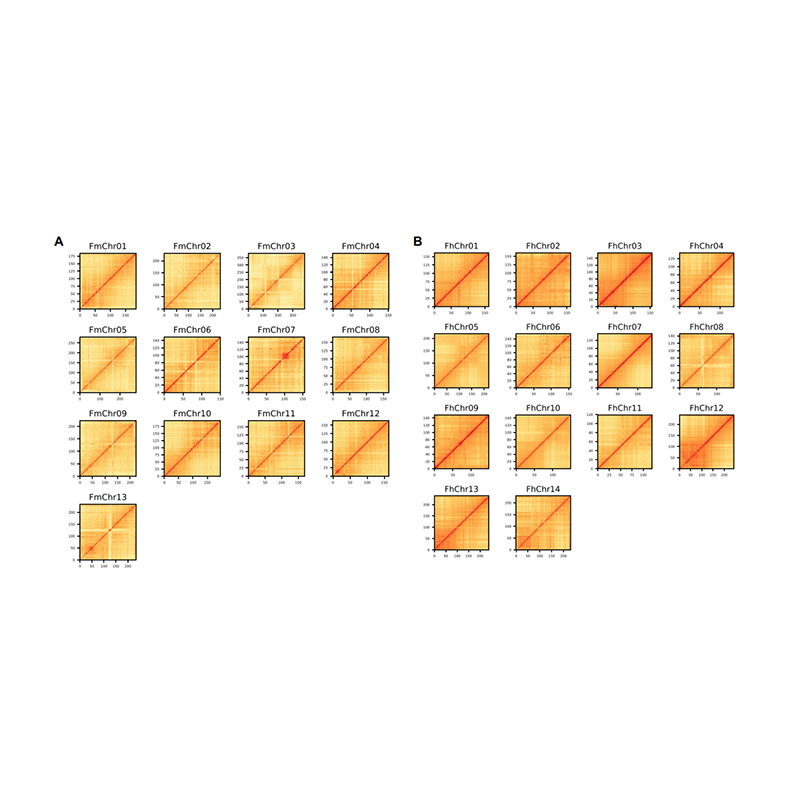
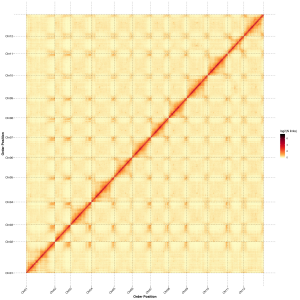
Évaluation post-assemblage - Carte thermique de l'intensité du signal entre les bacs
Explorez les progrès facilités par les services d'assemblage Hi-C de BMKGene grâce à une collection organisée de publications.
Tian, T. et al. (2023) «Assemblage du génome et dissection génétique d'un maïs à la sécheresse proéminent», Nature Genetics 2023 55: 3, 55 (3), pp. 496–506. doi: 10.1038 / s41588-023-01297-y.
Wang, ZL et al. (2020) «Un assemblage à l'échelle des chromosomes du génome des API de miel asiatique Cerana», Frontiers in Genetics, 11, p. 524140. Doi: 10.3389 / fgene.2020.00279 / bibtex.
Zhang, F. et al. (2023) «Révolution de l'évolution de la biosynthèse des alcaloïdes tropane en analysant deux génomes dans la famille Solanaceae», Nature Communications 2023 14: 1, 14 (1), pp. 1–18. doi: 10.1038 / s41467-023-37133-4.
Zhang, X. et al. (2020) «Les génomes de l'arbre banyan et de la guêpe des pollinisateurs fournissent un aperçu de la coévolution des figues», Cell, 183 (4), pp. 875-889.e17. doi: 10.1016 / j.cell.2020.09.043