Analyse d'association à l'échelle du génome
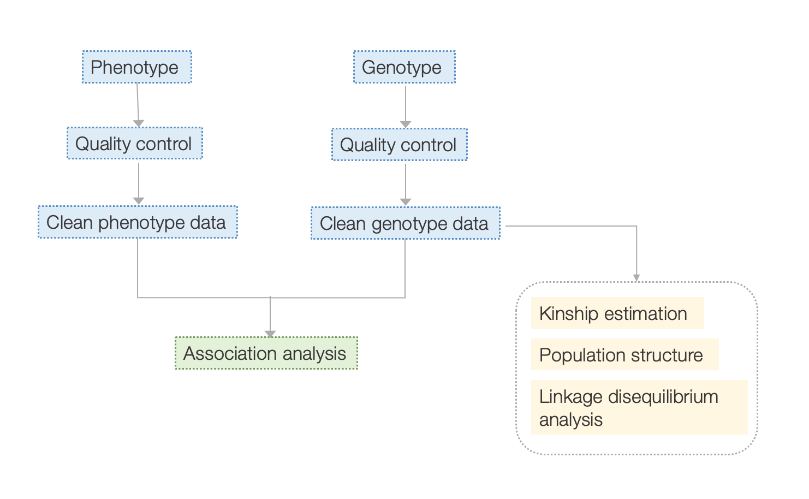
Flux de travail

Avantages des services
●Expertise approfondie et dossiers de publications: Fort de son expérience accumulée dans GWAS, BMKGene a réalisé des centaines de projets d'espèces dans la recherche sur les populations GWAS, aidé les chercheurs à publier plus de 100 articles et le facteur d'impact cumulé a atteint 500.
● Analyse bioinformatique complète: le flux de travail comprend l'analyse d'association de traits SNP, fournissant un ensemble de gènes candidats et leur annotation fonctionnelle correspondante.
●Équipe bioinformatique hautement qualifiée et cycle d’analyse court: forte d'une grande expérience en analyse génomique avancée, l'équipe de BMKGene fournit des analyses complètes dans des délais rapides.
●Assistance après-vente :Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant cette période, nous proposons un suivi de projet, une assistance au dépannage et des séances de questions-réponses pour répondre à toute question liée aux résultats.
Spécifications et exigences du service
| Type de séquençage | Échelle de population recommandée | Stratégie de séquençage | Besoins en nucléotides |
| Séquençage du génome entier | 200 échantillons | 10x | Concentration : ≥ 1 ng/μL Montant total≥ 30ng Dégradation ou contamination limitée ou nulle |
| Fragment amplifié à locus spécifique (SLAF) | Profondeur de la balise : 10x Nombre de balises : < 400 Mo : WGS est recommandé < 1 Go : 100 000 balises 1 Go > 2 Go : 300 000 balises Max 500 000 balises | Concentration ≥ 5 ng/µL Quantité totale ≥ 80 ng Nanogoutte OD260/280=1,6-2,5 Gel d'agarose : pas ou peu de dégradation ou de contamination
|
Sélection des matériaux



Différentes variétés, sous-espèces, races locales/banques de gènes/familles mixtes/ressources sauvages
Différentes variétés, sous-espèces, races locales
Famille demi-frère/famille pleine-frère/ressources sauvages
Flux de travail des services

Conception d'expériences

Livraison d'échantillon

Extraction d'ARN

Construction d'une bibliothèque

Séquençage

Analyse des données

Services après-vente
Comprend l’analyse suivante :
- Analyse d'association à l'échelle du génome : modèles LM, LMM, EMMAX, FASTLMM
- Annotation fonctionnelle des gènes candidats
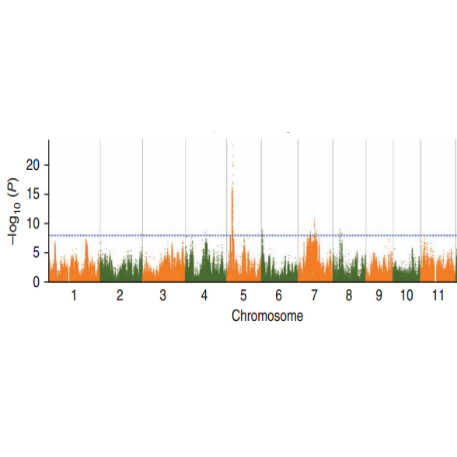
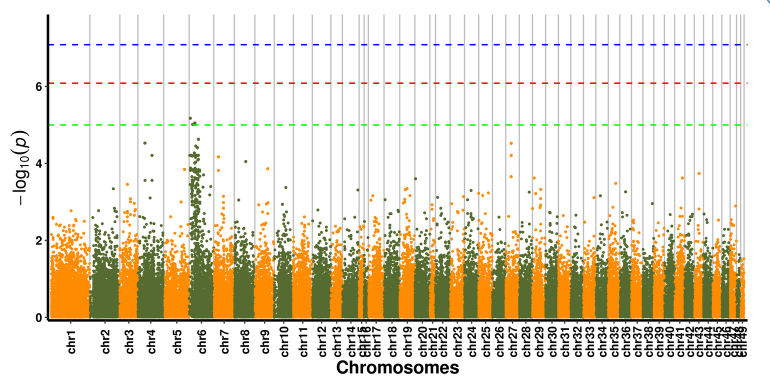
Analyse d'association SNP-trait – Graphique de Manhattan
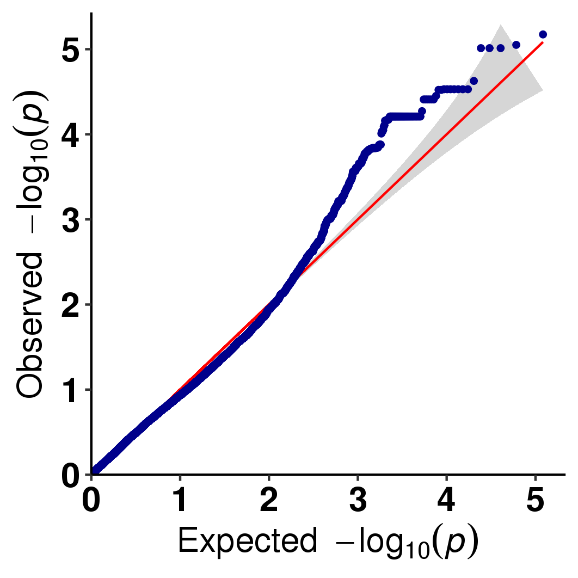
Analyse d'association SNP-trait – tracé QQ
Explorez les avancées facilitées par les services de GWAS de BMKGene à travers une collection de publications organisée :
Lv, L. et coll. (2023) « Aperçu des bases génétiques de la tolérance à l'ammoniac chez le couteau Sinonovacula constricta par une étude d'association à l'échelle du génome »,Aquaculture, 569, p. 739351. est ce que je : 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et coll. (2022) « Les analyses multi-omiques de 398 accessions de millet sétaire révèlent des régions génomiques associées à la domestication, aux traits métabolites et aux effets anti-inflammatoires »,Plante moléculaire, 15(8), pages 1367 à 1383. est ce que je : 10.1016/j.molp.2022.07.003.
Li, J. et coll. (2022) « Cartographie d'association à l'échelle du génome des phénotypes à peine nus dans un environnement de sécheresse »,Frontières de la science végétale, 13, p. 924892. est ce que je : 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) « GmST1, qui code pour une sulfotransférase, confère une résistance aux souches G2 et G3 du virus de la mosaïque du soja »,Plante, Cellule & Environnement, 44(8), pages 2777 à 2792. est ce que je: 10.1111/PCE.14066.