Assemblage de génome fongique de novo
Fonctionnalités de service
Avec trois options possibles à choisir en fonction du degré souhaité d'exhaustivité du génome:
● Projet d'option du génome: séquençage à lecture courte avec Illumina Novaseq PE150.
● Option de génome fin fongique:
Enquête sur le génome: Illumina Novaseq PE150.
Assemblage du génome: Revio PacBio (lecture HiFi) ou Nanopore Prométhion 48.
● Génome fongique au niveau du chromosome:
Enquête sur le génome: Illumina Novaseq PE150.
Assemblage du génome: Revio PacBio (lecture HiFi) ou Nanopore Prométhion 48.
Ancrage contig avec un assemblage Hi-C.
Avantages de service
●Plusieurs stratégies de séquençage disponibles: Pour différents objectifs de recherche et exigences de l'exhaustivité du génome
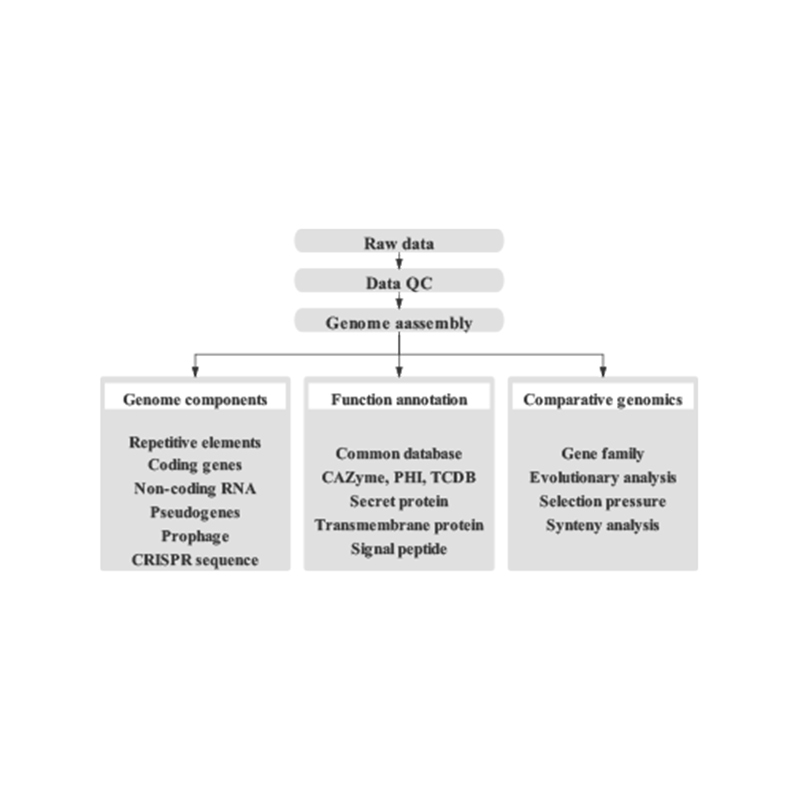
●Flux de travail bioinformatique complet:Cela comprend l'assemblage du génome et la prédiction de plusieurs éléments génomiques, l'annotation du gène fonctionnel et l'ancrage contig.
●Expertise approfondie: Avec plus de 12 000 génomes microbiens assemblés, nous apportons plus d'une décennie d'expérience, d'une équipe d'analyse hautement qualifiée, d'un contenu complet et d'un excellent support post-vente.
●Support post-vente:Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant ce temps, nous offrons un suivi du projet, une assistance de dépannage et des séances de questions-réponses pour répondre à toutes les requêtes liées aux résultats.
Spécifications de service
| Service | Stratégie de séquençage | Contrôle de qualité |
| Projet de génome | Illumina PE150 100X | Q30 ≥ 85% |
| Génome fin | Enquête sur le génome: Illumina PE150 50 X Assemblage: Pacbi Hifi 30x ou Nanopore 100X | contig n50 ≥1mb (pacbio unicellulaire) contig n50 ≥2 Mo (ONT unicellulaire) contig n50 ≥500kb (autres) |
| Génome au niveau du chromosome | Enquête sur le génome: Illumina PE150 50 X Assemblage: Pacbi Hifi 30x ou Nanopore 100X Assemblage Hi-C 100X | Ratio d'ancrage contig> 90%
|
Exigences de service
| Concentration (ng / µl) | Quantité totale (µg) | Volume (µl) | OD260 / 280 | OD260 / 230 | |
| Pacbio | ≥20 | ≥2 | ≥20 | 1.7-2.2 | ≥1,6 |
| Nanopore | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1.0-3.0 |
| Illumina | ≥1 | ≥0,06 | ≥20 | - | - |
Fongus unicellulaire: ≥3,5x1010 cellules
Macro Fungus: ≥ 10 g
Flux de travail de service

Livraison d'échantillons

Construction de la bibliothèque

Séquençage

Analyse des données

Services après-vente
Comprend l'analyse suivante:
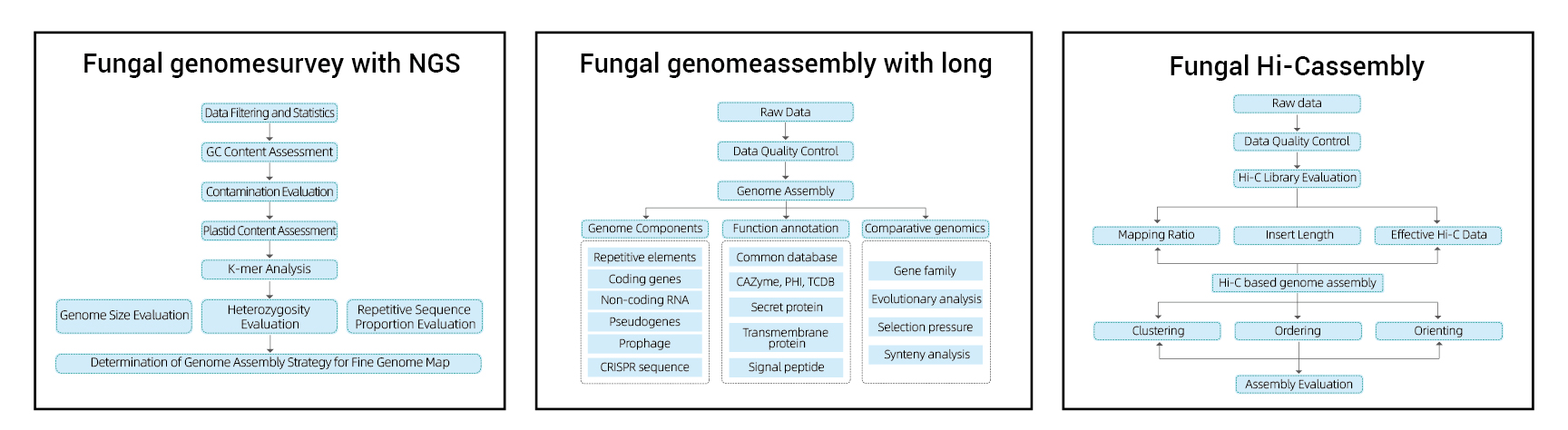
Survey du génome:
- Séquençage du contrôle de la qualité des données
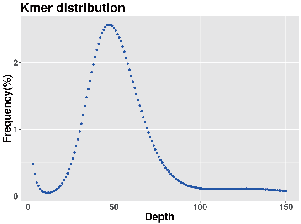
- Estimation du génome: taille, hétérozygotie, éléments répétitifs
Assemblage du génome fin:
- Séquençage du contrôle de la qualité des données
- De novoAssemblée
- Analyse des composants du génome: prédiction des CD et des éléments génomiques multiples
- Annotation fonctionnelle avec plusieurs bases de données générales (Go, KEGG, etc.) et des bases de données avancées (carte, VFDB, etc.)
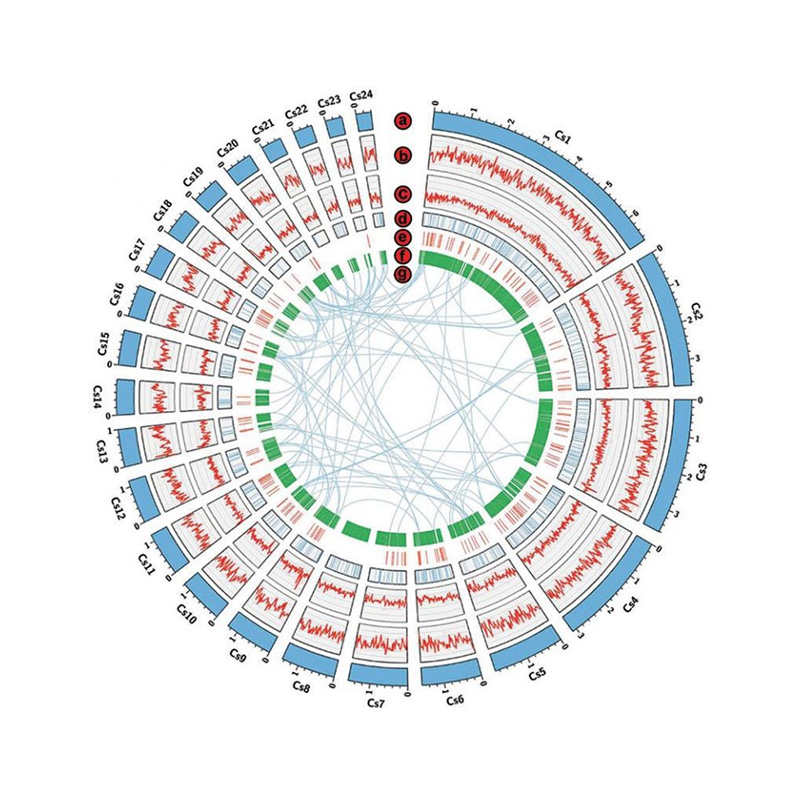
Assemblage Hi-C:
- Évaluation de la bibliothèque HI-C.
- Contigs ancrant le regroupement, l'ordre et l'orientation
- Évaluation de l'assemblage Hi-C: basé sur le génome de référence et la carte thermique
Enquête sur le génome: distribution K-Mer
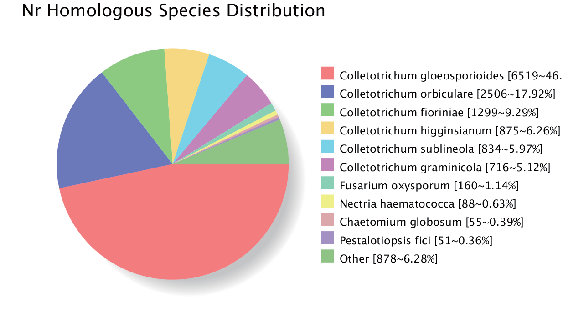
Assemblage du génome: annotation homologue gène (base de données NR)
Assemblage du génome: annotation du gène fonctionnel (GO)
Explorez les progrès facilités par les services d'assemblage fongique du génome fongique de BMKGene grâce à une collection organisée de publications.
Hao, J. et al. (2023) «Profil omic intégré du champignon médicinal Inonotus obliquus dans des conditions submergées»,BMC Genomics, 24 (1), pp. 1–12. doi: 10.1186 / S12864-023-09656-Z / Figures / 3.
Lu, L. et al. (2023) «Le séquençage du génome révèle l'évolution et les mécanismes pathogènes de l'agent pathogène du blé et de la rhizoctonia ceréalis»,Le journal des cultures, 11 (2), pp. 405–416. doi: 10.1016 / j.cj.2022.07.024.
Zhang, H. et al. (2023) «Ressources du génome pour quatre espèces de Clarireedia provoquant une tache en dollars sur divers gazons»,Maladie des plantes, 107 (3), pp. 929–934. doi: 10.1094 / PDIS-08-22-1921-A
Zhang, SS et al. (2023) «Preuve génétique et moléculaire d'un système d'accouplement tétrapolaire dans le champignon comestible Grifola frondosa»,Journal of Fungi, 9 (10), p. 959. Doi: 10.3390 / jof9100959 / s1.