Séquençage d'ARNm complet-nanopore
Caractéristiques
● Capture de l'ARNm poly-a suivi d'une synthèse d'ADNc et d'une préparation de bibliothèque
● Séquençage des transcriptions complètes
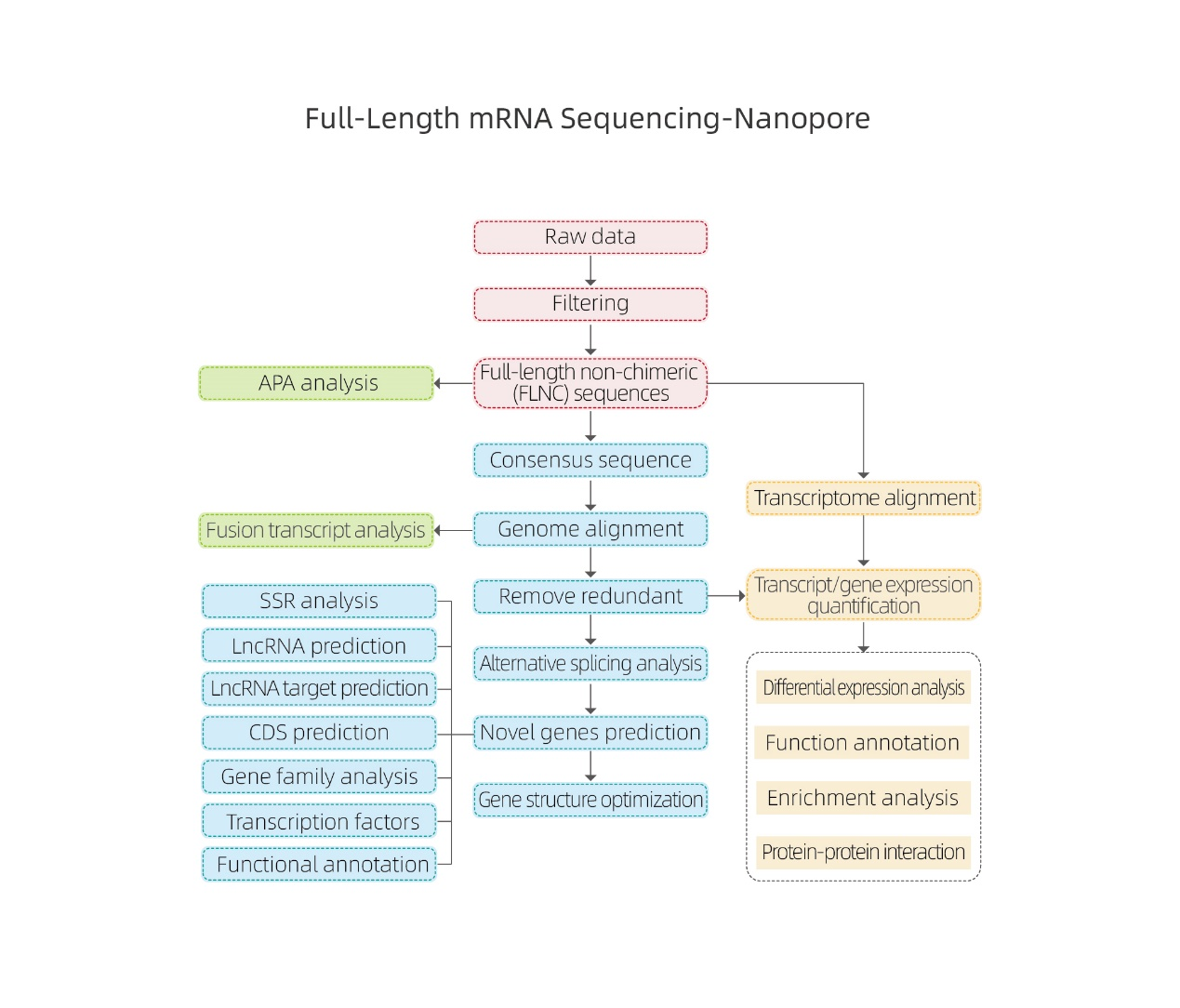
● Analyse bioinformatique basée sur l'alignement sur un génome de référence
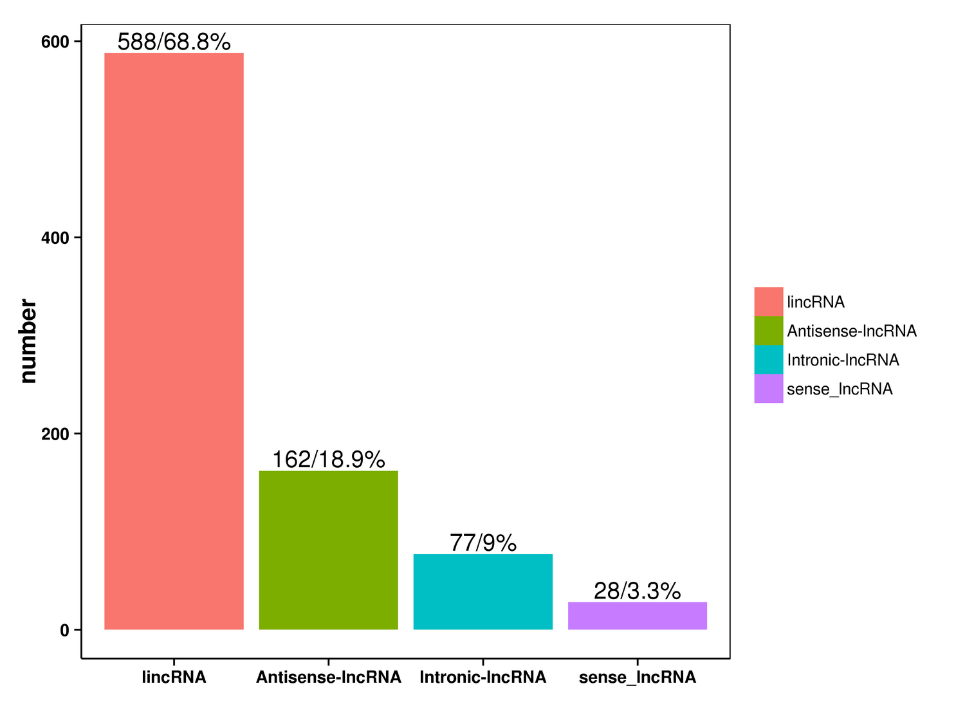
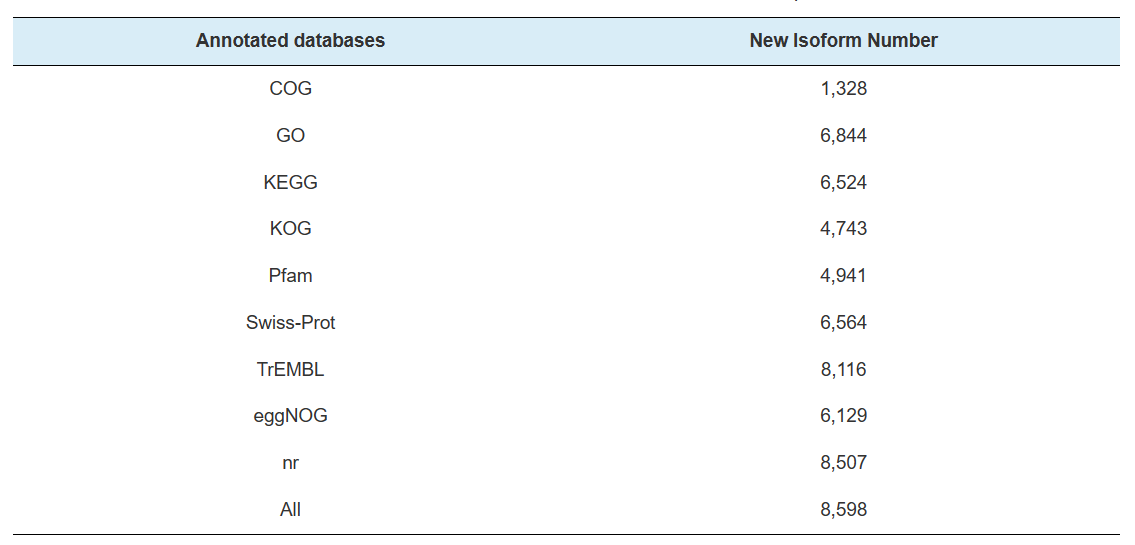
● L'analyse bioinformatique comprend non seulement l'expression au niveau du gène et du niveau des isoformes, mais aussi une analyse de LNCRNA, des fusions géniques, de la poly-adénylation et de la structure des gènes
Avantages de service
●Quantification de l'expression au niveau des isoformes: activer une analyse d'expression détaillée et précise, dévoiler un changement qui peut être masqué lors de l'analyse de l'expression du gène entier
●Réduction des demandes de données:Par rapport au séquençage de nouvelle génération (NGS), le séquençage des nanopores présente des exigences de données plus faibles, permettant des niveaux équivalents de saturation de quantification de l'expression des gènes avec des données plus petites.
●Précision plus élevée de la quantification de l'expression: à la fois au niveau du gène et de l'isoforme
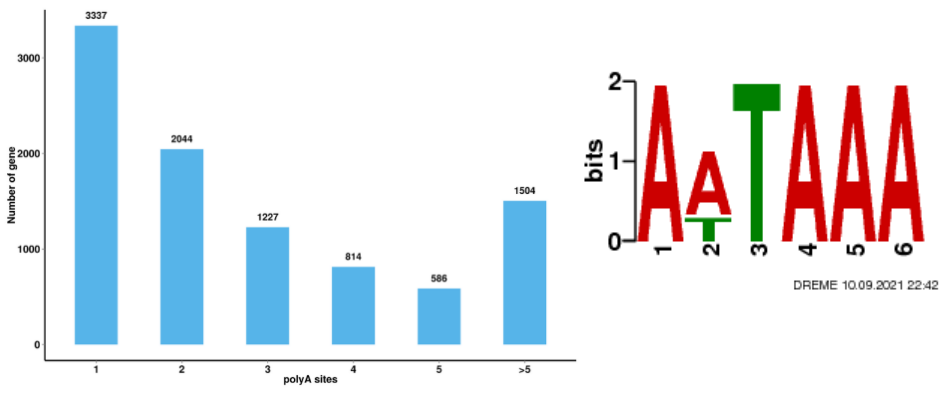
●Identification des informations transcriptomiques supplémentaires: polyadénylation alternative, gènes de fusion et LCNRNA et leurs gènes cibles
●Expertise approfondie: Notre équipe apporte une richesse d'expérience à chaque projet, ayant achevé plus de 850 projets de transcriptome complet de nanopore et traité plus de 8 000 échantillons.
●Support post-vente: Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant ce temps, nous offrons un suivi du projet, une assistance de dépannage et des séances de questions-réponses pour répondre à toutes les requêtes liées aux résultats.
Exemples d'exigences et de livraison
| Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| Poly A enrichi | Illumina PE150 | 6/12 Go | Score de qualité moyen: Q10 |
Exemples d'exigences:
Nucléotides:
| Conc. (Ng / μl) | Quantité (μg) | Pureté | Intégrité |
| ≥ 100 | ≥ 1,0 | OD260 / 280 = 1,7-2,5 OD260 / 230 = 0,5-2,5 Contamination limitée ou non protéique ou ADN montré sur le gel. | Pour les plantes: RIN ≥ 7,0; Pour les animaux: rin ≥ 7,5; 5.0≥28s / 18S ≥ 1,0; Élévation limitée ou non de référence |
● Plantes:
Racine, tige ou pétale: 450 mg
Feuille ou graine: 300 mg
Fruit: 1,2 g
● Animal:
Cœur ou intestin: 300 mg
Viscères ou cerveau: 240 mg
Muscle: 450 mg
Os, cheveux ou peau: 1g
● Arthropodes:
Insectes: 6G
Crustacea: 300 mg
● sang total: 1 tube
● cellules: 106 cellules
Livraison d'échantillon recommandée
Récipient: 2 ml de tube à centrifugeuse (la papier d'étain n'est pas recommandée)
Exemple d'étiquetage: groupe + réplique par exemple A1, A2, A3; B1, B2, B3.
Expédition:
1. Face sèche: les échantillons doivent être emballés dans des sacs et enterrés dans la glace sèche.
2. Tubes raastables: les échantillons d'ARN peuvent être séchés dans un tube de stabilisation de l'ARN (par exemple RNastable®) et expédiés à température ambiante.
Flux de travail de service
Nucléotides:

Livraison d'échantillons

Construction de la bibliothèque

Séquençage

Analyse des données

Services après-vente
Flux de travail de service
Tissu:

Conception d'expérience

Livraison d'échantillons

Extraction de l'ARN

Construction de la bibliothèque

Séquençage

Analyse des données

Services après-vente
● Traitement des données brutes
● Identification des transcrits
● Épissage alternatif
● Quantification de l'expression au niveau des gènes et au niveau des isoformes
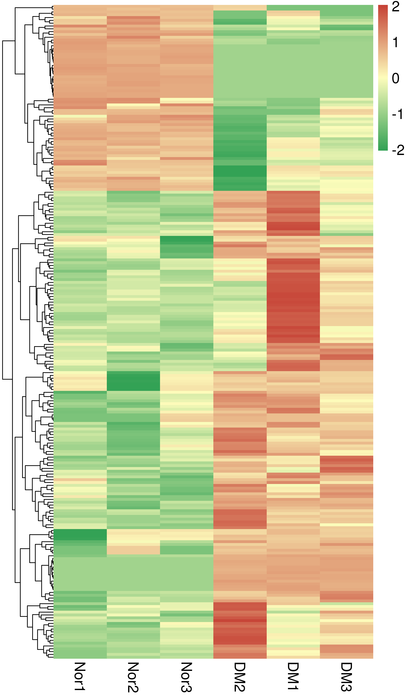
● Analyse de l'expression différentielle
● Annotation et enrichissement de la fonction (Degs et DÉTS)
Analyse d'épissage alternative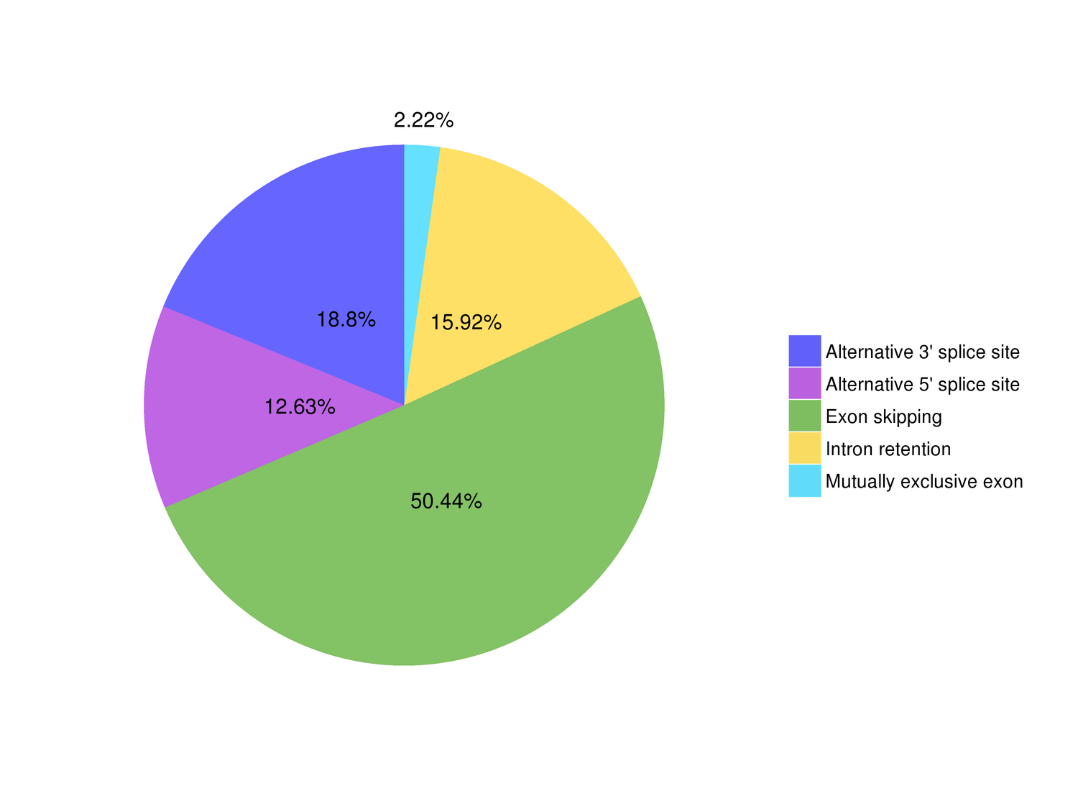 Analyse alternative en polyadénylation (APA)
Analyse alternative en polyadénylation (APA)
prédiction de l'ARNNm
Annotation de nouveaux gènes
Clustering de DÉTS
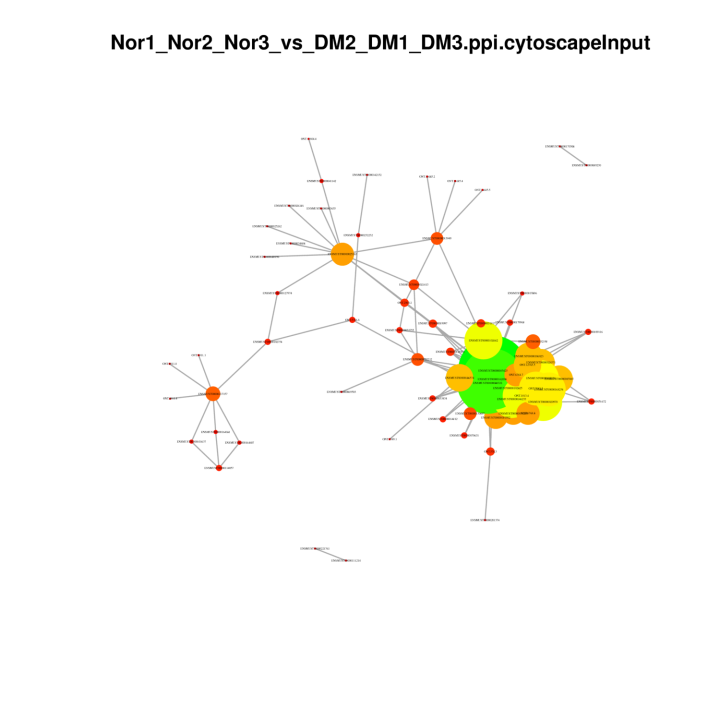
Réseaux protéiques-protéine en degs
Explorez les progrès facilités par les services de séquençage d'ARNm complet de Nanopore de BMKGene grâce à une collection de publications organisée.
Gong, B. et al. (2023) «Activation épigénétique et transcriptionnelle de la sécrétoire Kinase FAM20C en tant qu'oncogène dans le gliome», Journal of Genetics and Genomics, 50 (6), pp. 422–433. doi: 10.1016 / j.jgg.2023.01.008.
Lui, Z. et al. (2023) `` Le séquençage de transcriptome pleine longueur des lymphocytes répond à l'IFN-γ révèle une réponse immunitaire ciblée de Th1 chez Flounder (Paralichthys olivaceus) ', Fish & Shellfish Immunology, 134, p. 108636. Doi: 10.1016 / j.fsi.2023.108636.
Ma, Y. et al. (2023) «Analyse comparative des méthodes de séquençage de l'ARN PacBio et ONT pour l'identification du venin Nemopilema nomurai», Genomics, 115 (6), p. 110709. Doi: 10.1016 / j.ygeno.2023.110709.
Yu, D. et al. (2023) 'L'analyse Nano-Seq révèle une tendance fonctionnelle différente entre les exosomes et les microvésicules dérivées de HUMSC', Research and Therapy, 14 (1), pp. 1–13. doi: 10.1186 / s13287-023-03491-5 / tableaux / 6.