Séquençage de l'ARNm eucaryote-NGS
Caractéristiques
● Capture de poly ARNm avant la préparation de la bibliothèque
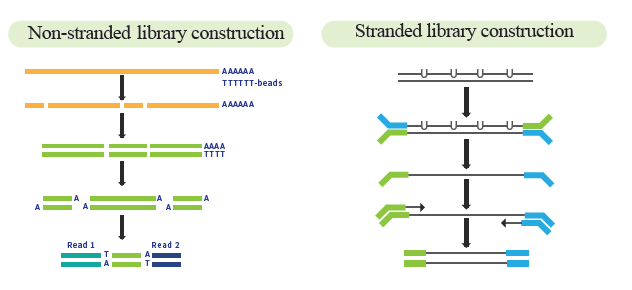
● Préparation de bibliothèque d'ARNm directionnelle en option pour permettre l'obtention de données de séquençage spécifiques à un brin
● Analyse bioinformatique de l'expression des gènes et de la structure des transcriptions
Avantages
●Une expertise étendue: Notre équipe a traité plus de 600 000 échantillons chez BMKgene, couvrant divers types d'échantillons tels que des cultures cellulaires, des tissus et des fluides corporels. Nous apportons une riche expérience à chaque projet et avons réalisé avec succès plus de 100 000 projets ARNm-Seq dans divers domaines de recherche.
●Contrôle qualité rigoureux: Nous mettons en œuvre des points de contrôle de base à toutes les étapes, de la préparation des échantillons et des bibliothèques au séquençage et à la bioinformatique. Ce suivi méticuleux garantit la fourniture de résultats constants de haute qualité, vous assurant que votre projet est entre de bonnes mains.
●Annotation complète: Nous utilisons plusieurs bases de données pour annoter fonctionnellement les gènes exprimés différentiellement (DEG) et effectuer les analyses d'enrichissement correspondantes. Cette approche globale fournit un aperçu des processus cellulaires et moléculaires sous-jacents à la réponse du transcriptome, vous garantissant ainsi d'être pleinement informé des résultats de votre projet.
●Assistance après-vente: Notre engagement s'étend au-delà de la réalisation du projet avec une période de service après-vente de 3 mois. Pendant cette période, nous proposons un suivi de projet, une assistance au dépannage et des séances de questions-réponses pour répondre à toute question liée aux résultats.
Exigences et livraison des échantillons
| Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| Poly A enrichi | Illumina PE150 DNBSEQ-T7 | 6 à 10 Go | Q30≥85 % |
Exigences de l'échantillon :
Nucléotides :
| Conc. (ng/μl) | Quantité (μg) | Pureté | Intégrité | |
| Bibliothèque standard | ≥ 10 | ≥ 0,2 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contamination limitée ou inexistante en protéines ou en ADN indiquée sur le gel. | Pour les plantes : RIN≥4,0 ; Pour les animaux : RIN≥4,5 ; 5,0≥28S/18S≥1,0 ; élévation de la ligne de base limitée ou inexistante |
| Bibliothèque directionnelle | ≥ 10 | ≥ 0,2 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contamination limitée ou inexistante en protéines ou en ADN indiquée sur le gel. | Pour les plantes : RIN≥4,0 ; Pour les animaux : RIN≥4,5 ; 5,0≥28S/18S≥1,0 ; élévation de la ligne de base limitée ou inexistante |
● Plantes :
Racine, tige ou pétale : 450 mg
Feuille ou graine : 300 mg
Fruits : 1,2 g
●Animal:
COEUR ou Intestin : 300 mg
Viscères ou Cerveau : 240 mg
Muscles : 450 mg
Os, cheveux ou peau : 1g
● Arthropodes :
Insectes : 6g
Crustacés : 300 mg
● Sang total: 1 tube
● Cellules: 106 cellules
Livraison d’échantillon recommandée
Récipient : tube à centrifuger de 2 ml (le papier d'aluminium n'est pas recommandé)
Étiquetage des échantillons : Groupe + répétition, par exemple A1, A2, A3 ; B1, B2, B3... ...
Expédition:
1. Glace sèche : Les échantillons doivent être emballés dans des sacs et enterrés dans de la neige carbonique.
2. Tubes de table d'ARN : les échantillons d'ARN peuvent être séchés dans un tube de stabilisation d'ARN (par exemple RNAstable®) et expédiés à température ambiante.
Flux de travail des services

Conception d'expériences

Livraison d'échantillon

Extraction d'ARN

Construction d'une bibliothèque

Séquençage

Analyse des données

Services après-vente
Bioinformatique
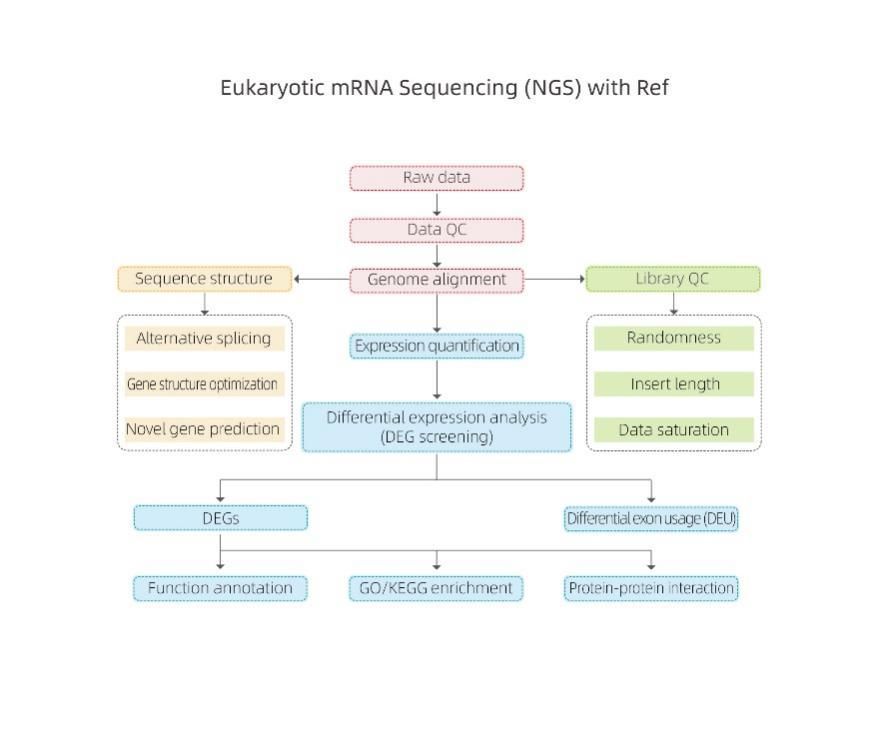
Eucaryote Flux de travail d'analyse de séquençage d'ARNm
Bioinformatique
ØContrôle qualité des données brutes
ØAlignement du génome de référence
ØAnalyse de la structure de la transcription
ØQuantification des expressions
ØAnalyse d'expression différentielle
ØAnnotation et enrichissement des fonctions
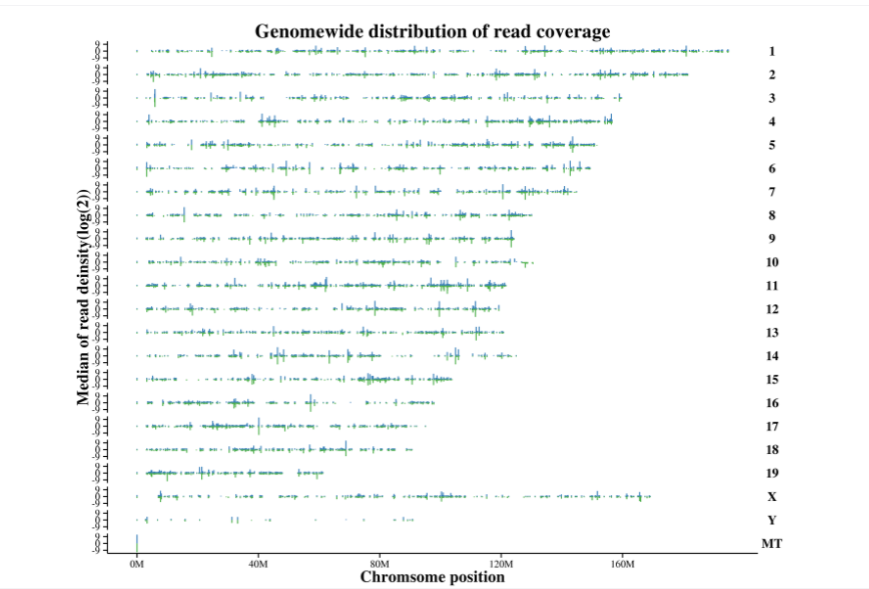
Alignement du génome de référence
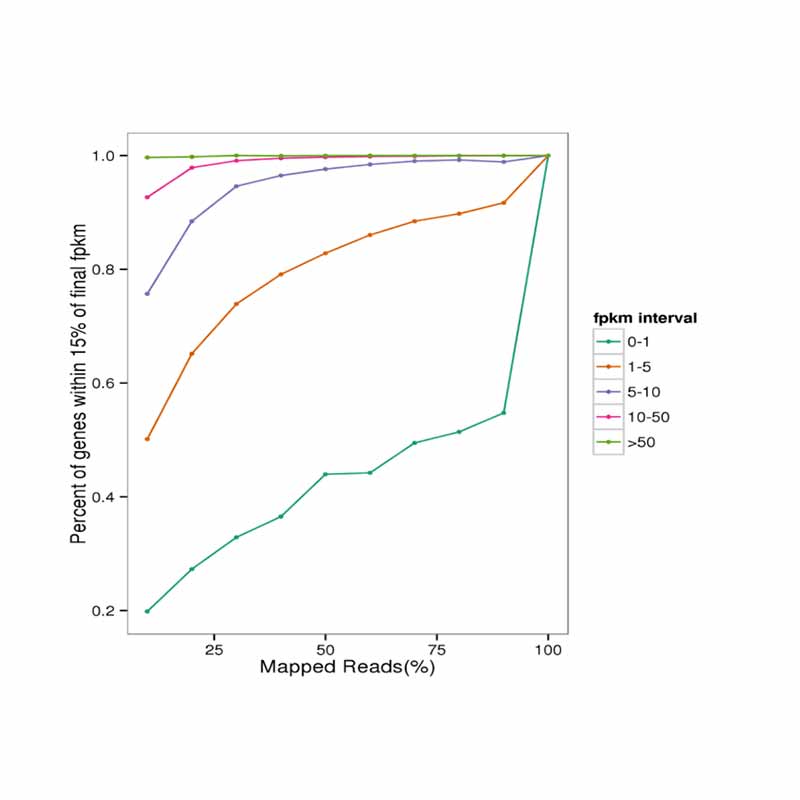
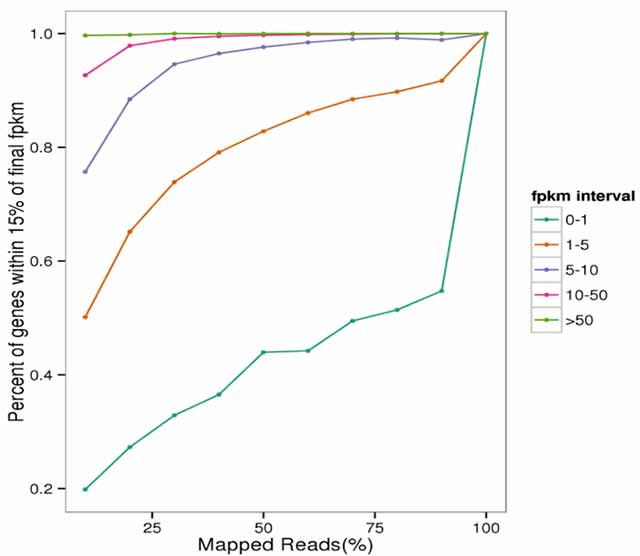
Saturation des données
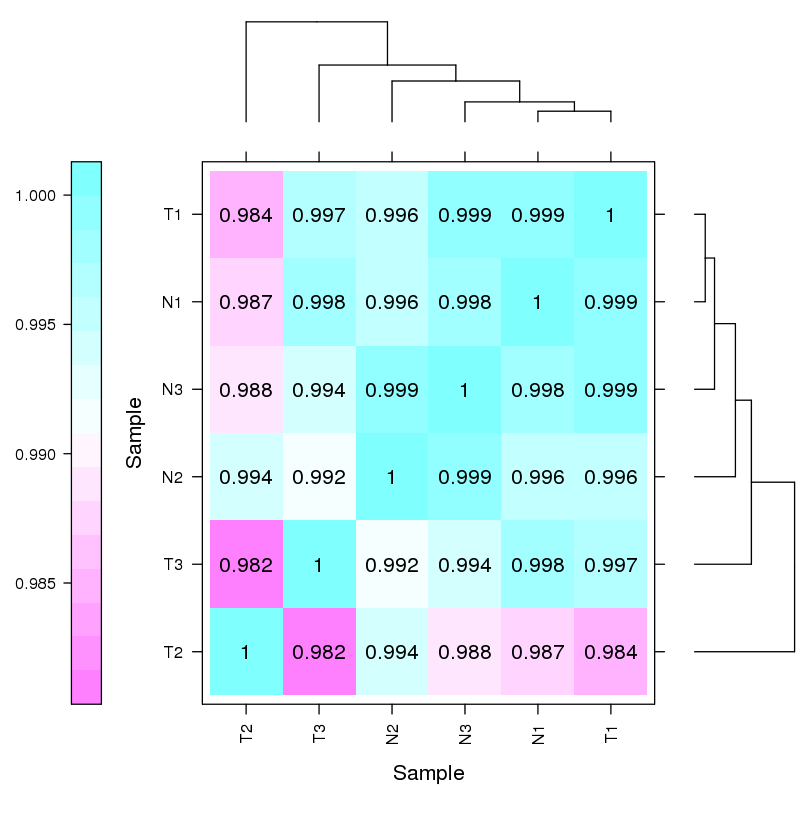
Corrélation d'échantillons et évaluation de répétitions biologiques
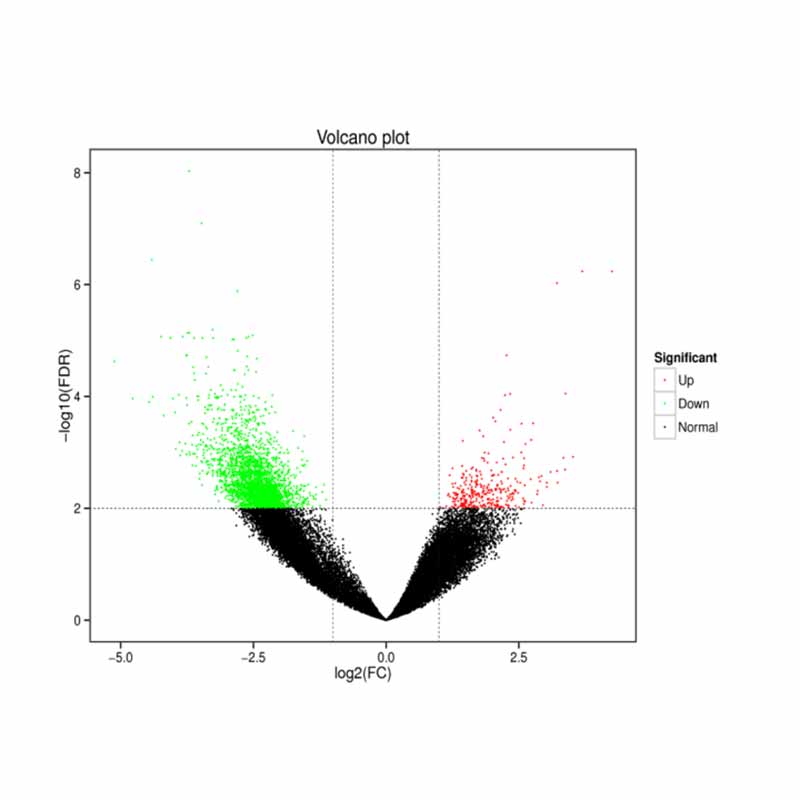
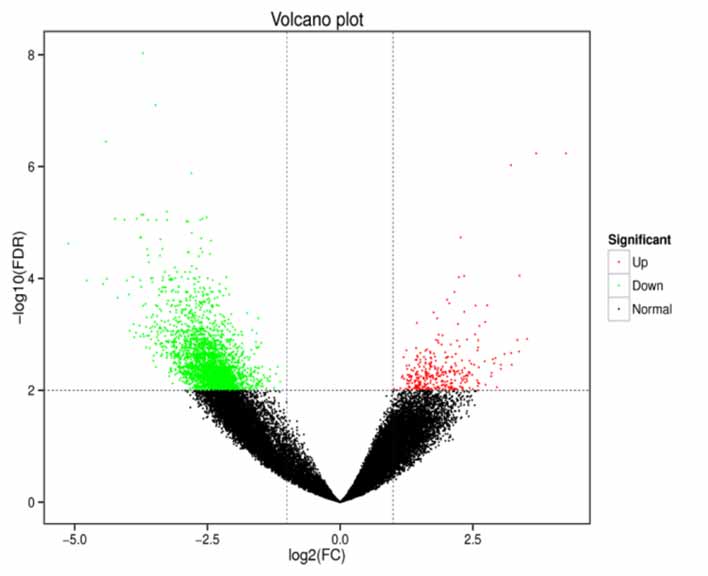
Gènes exprimés différentiellement (DEG)
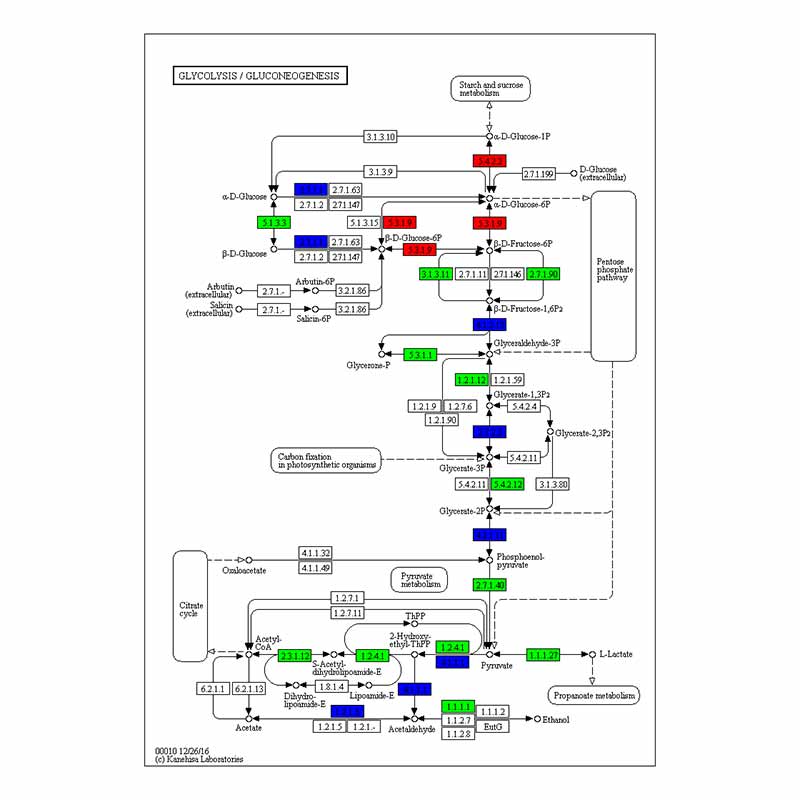
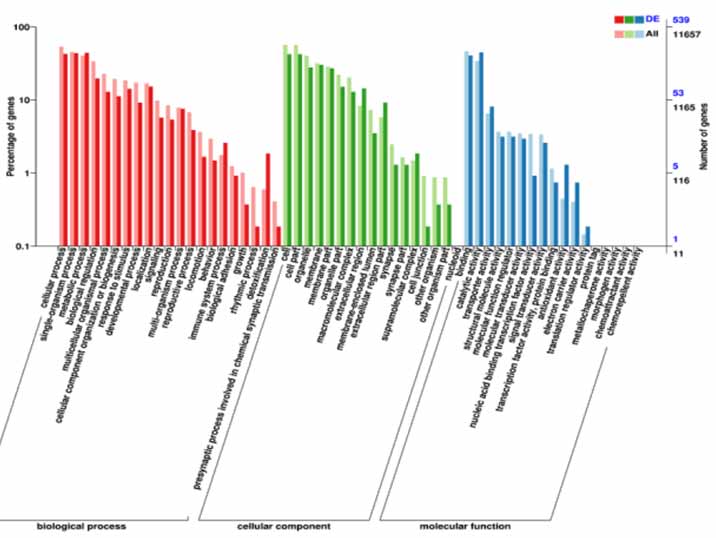
Annotation fonctionnelle des DEG
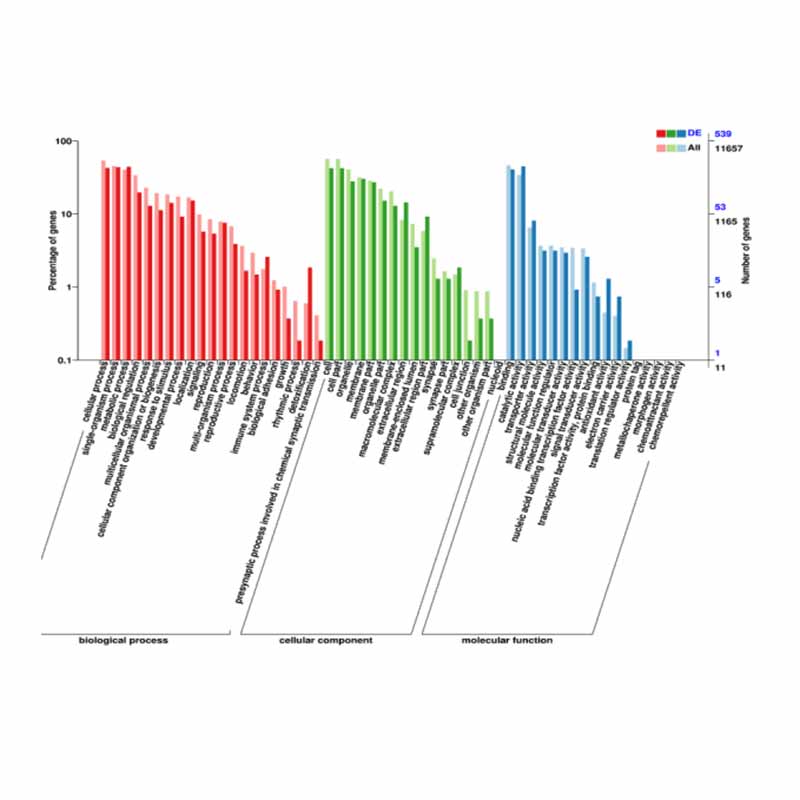
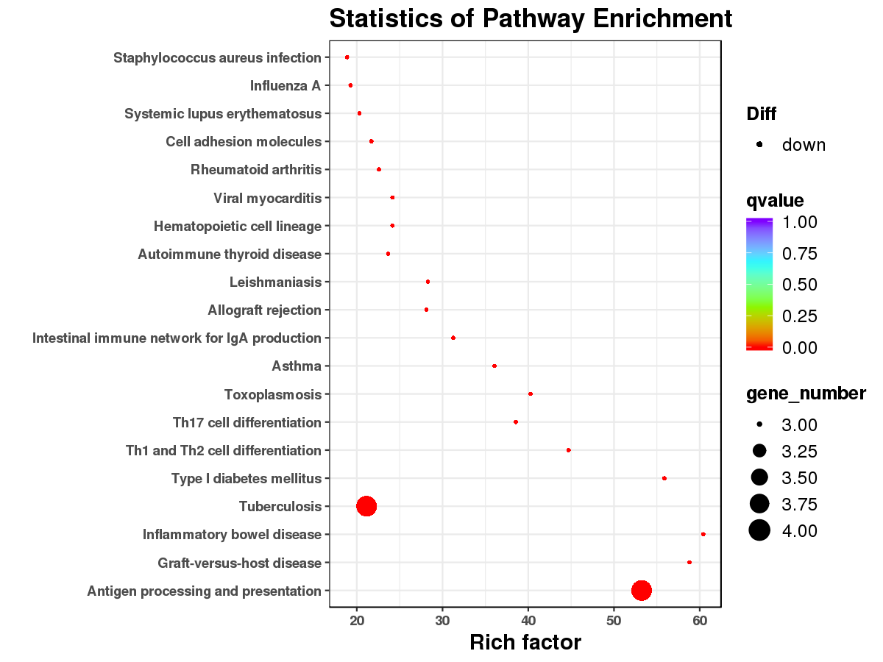
Enrichissement fonctionnel des DEG
Explorez les progrès facilités par les services de séquençage d’ARNm eucaryotes NGS de BMKGene à travers une collection organisée de publications.
Huang, L. et coll. (2023) « Le triclosan et le triclocarban affaiblissent la capacité olfactive des poissons rouges en limitant la reconnaissance des odeurs, en perturbant la transduction du signal olfactif et en perturbant le traitement de l'information olfactive », Water Research, 233, p. 119736. est ce que je : 10.1016/J.WATRES.2023.119736.
Jia, LJ et coll. (2023) « Aspergillus fumigatus détourne la p11 humaine pour rediriger les phagosomes contenant des champignons vers une voie non dégradante », Cell Host & Microbe, 31(3), pp. 373-388.e10. est ce que je: 10.1016/J.CHOM.2023.02.002.
Jin, K. et al. (2022) « Facteurs de transcription TCP impliqués dans le développement des pousses du bambou Ma (Dendrocalamus latiforus Munro) », Frontiers in Plant Science, 13, p. 884443. est ce que je : 10.3389/FPLS.2022.884443/BIBTEX.
Wen, X. et coll. (2022) « Le génome de Chrysanthemum lavandulifolium et le mécanisme moléculaire sous-jacent à divers types de capitules », Horticulture Research, 9. doi : 10.1093/HR/UHAB022.
Zhang, Yujie et coll. (2023) « Un nanoréacteur en cascade pour améliorer la thérapie sonodynamique sur le cancer colorectal via une augmentation synergique des ROS et un blocage de l'autophagie », Nano Today, 49, p. 101798. est ce que je : 10.1016/J.NANTOD.2023.101798.