Bibliothèques pré-faites DNBSEQ
Caractéristiques
●Plate-forme:MGI-DNBSEQ-T7
●Modes de séquençage:PE150
●Transfert de bibliothèques Illumina àMGI:permettant le séquençage de volumes de données élevés à faible coût.
●Contrôle de la qualité des bibliothèques avant le séquençage.
●Données de séquençage QC et livraison:La livraison du rapport QC et des données brutes au format FastQ après démultiplexage et filtrage des lectures de Q30.
Avantages
●Polyvalence des services de séquençage:Le client peut choisir de séquencer par voie ou quantité de données.
●Sortie de données élevée:1500 Go / Lane
●Livraison du séquençage du rapport QC:Avec des mesures de qualité, la précision des données et les performances globales du projet de séquençage.
●Processus de séquençage mature:Avec un court délai de retournement.
●Contrôle de la qualité rigoureux: Nous mettons en œuvre des exigences strictes QC pour garantir la livraison de résultats de haute qualité de haute qualité.
Exigences d'échantillonnage
| Montant des données (x) | Concentration (qPCR / nm) | Volume | |
| Voie partielle
| X ≤ 10 Go | ≥ 1 nm | ≥ 25 μl |
| 10 Go <x ≤ 50 Go | ≥ 2 nm | ≥ 25 μl | |
| 50 Go <x ≤ 100 Go | ≥ 3 nm | ≥ 25 μl | |
| X> 100 Go | ≥ 4 nm | ||
| Voie unique | Par voie | Pool ≥ 1,5 nm / bibliothèque | ≥ 25 μl / piscine de bibliothèque |
En plus de la concentration et de la quantité totale, un schéma de pic approprié est également requis.
Remarque: Le séquençage de voie des bibliothèques à faible diversité nécessite Phix Spike-in pour assurer une vocation de base robuste.
Nous vous recommandons de soumettre des bibliothèques préalables comme échantillons. Si vous avez besoin de BMKGene pour effectuer un pool de bibliothèques, veuillez vous référer à
Exigences de la bibliothèque pour le séquençage de voie partielle.
Taille de la bibliothèque (carte de pointe)
Le pic principal doit être à moins de 300 à 450 pb.
Les bibliothèques doivent avoir un seul pic principal, pas de contamination de l'adaptateur et pas de dimères d'amorce.
Flux de travail de service




Rapport de la bibliothèque QC
Un rapport sur la qualité de la bibliothèque est fourni avant le séquençage, évaluant le montant de la bibliothèque et fragmentation.
Rapport QC de séquençage
Tableau 1. Statistiques sur les données de séquençage.
| Échantillonnage | Bmkid | Lectures brutes | Données brutes (BP) | Clean Reads (%) | Q20 (%) | Q30 (%) | GC (%) |
| C_01 | BMK_01 | 22 870 1320 | 6 861 036 000 | 96.48 | 99.14 | 94.85 | 36.67 |
| C_02 | BMK_02 | 14,717 867 | 4 415 360,100 | 96.00 | 98,95 | 93.89 | 37.08 |
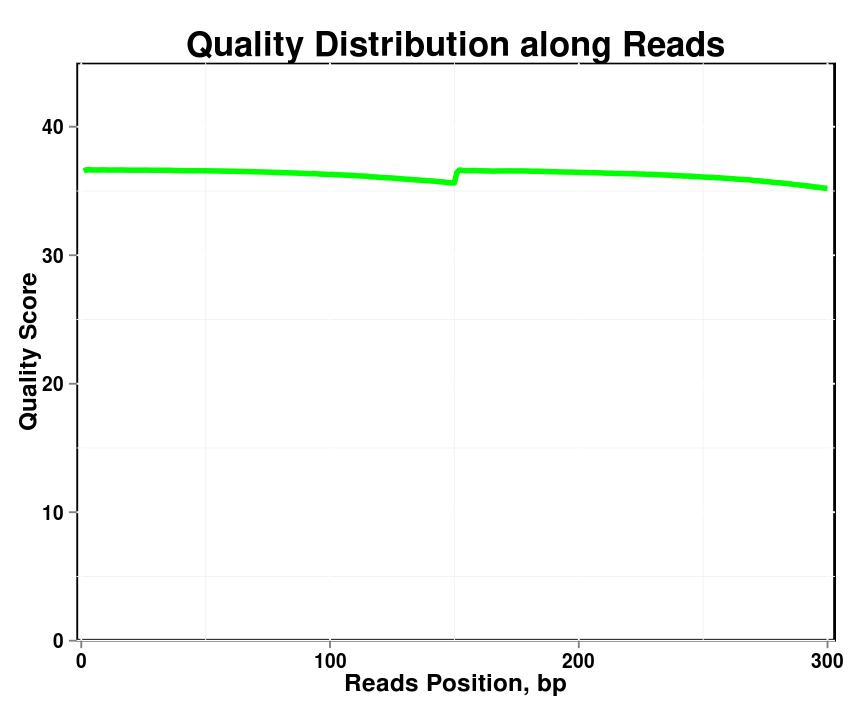
Figure 1. Distribution de qualité le long des lectures dans chaque échantillon
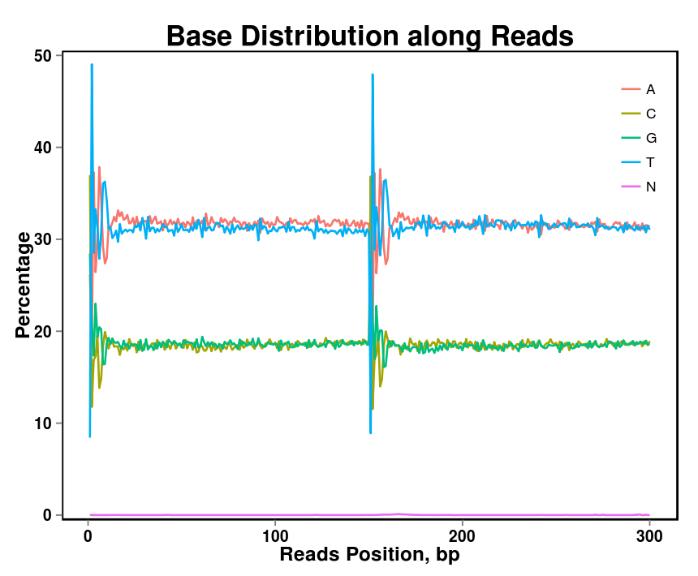
Figure 2. Distribution de contenu de base
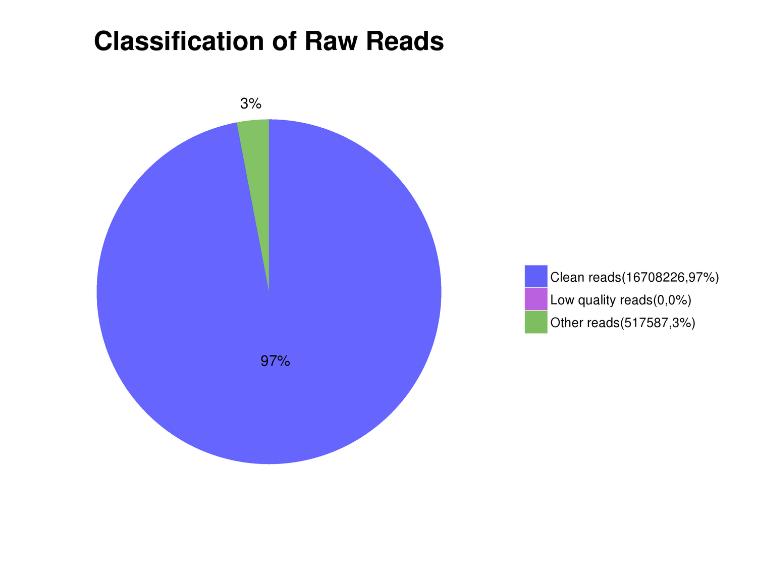
Figure 3. Distribution du contenu en lecture dans les données de séquençage