Assemblage du génome bactérien de novo
Caractéristiques des services
● Avec deux options possibles au choix selon le degré d'exhaustivité du génome souhaité.
● Option Draft Genome : séquençage en lecture courte avec Illumina NovaSeq PE150.
● Génome complet à 0 lacune.
● Séquençage en lecture longue sur Nanopore PromethION 48 ou PacBio Revio pour l'assemblage du génome.
● Séquençage en lecture courte sur Illumina NovaSeq pour la validation du génome et la correction d'erreurs (Nanopore) ou pour générer un projet de génome.
Avantages des services
●Génome Zero-Gap garanti: Cela est dû à l’intégration du séquençage Illumina avec le séquençage à lecture longue (Nanopore ou PacBio).
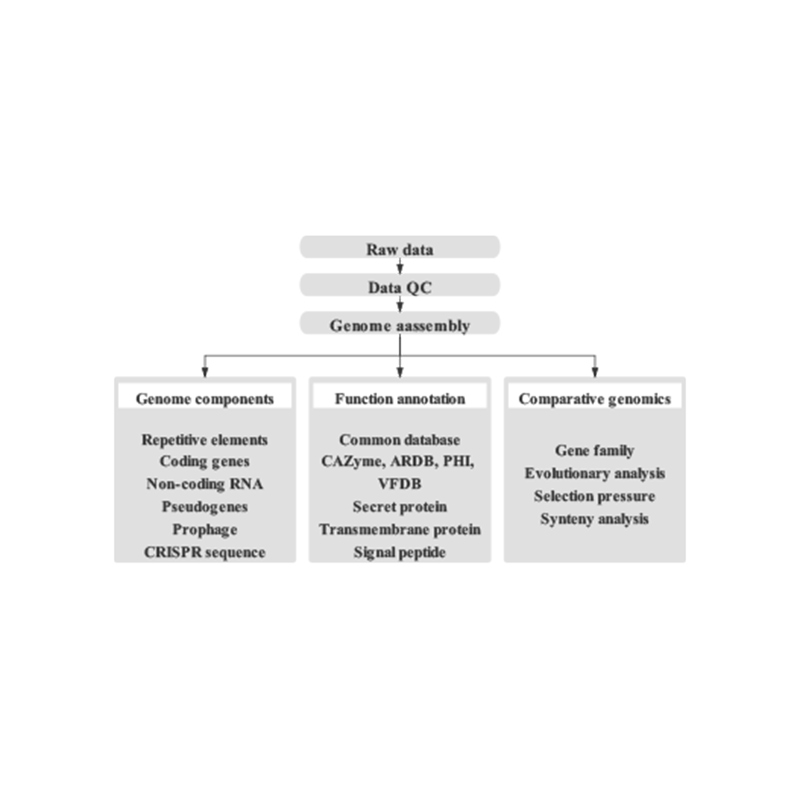
●Flux de travail bioinformatique complet :Cela inclut l'assemblage du génome et la prédiction de plusieurs éléments génomiques, l'annotation fonctionnelle des gènes et les visualisations du génome telles que le tracé Circos.
●Une expertise étendue: Avec plus de 20 000 génomes microbiens assemblés, BMKGENE apporte plus d'une décennie d'expérience, une équipe d'analyse hautement qualifiée, un contenu complet et un excellent support après-vente.
●Assistance après-vente :Notre engagement s'étend au-delà de l'achèvement du projet avec une période de service après-vente de 3 mois. Pendant cette période, nous proposons un suivi de projet, une assistance au dépannage et des séances de questions-réponses pour répondre à toute question liée aux résultats.
●Plusieurs stratégies de séquençage disponibles :Pour différents objectifs de recherche et exigences en matière d’exhaustivité du génome.
Spécifications des services
| Service | Stratégie de séquençage |
| Projet de génome | Illumina PE150 100x |
| Génome à 0 écart | Nanopore 100x + Illumina PE150 100x Or Pacbio HiFi 30x + Illumina PE150 100x (en option) |
Exigences de l'échantillon :
| Concentration (ng/μL) | Quantité totale (µg) | Volume (µL) | OD260/280 | OD260/230 | |
| PacBio | ≥20 | ≥1,2 | ≥20 | 1.7-2.2 | ≥1,0 |
| Nanopore | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1,0-3,0 |
| Illumine | ≥1 | ≥0,06 | ≥20 | - | - |
· Bactéries : ≥3,5x1010 cellules
Flux de travail des services

Livraison d'échantillon

Construction d'une bibliothèque

Séquençage

Analyse des données

Services après-vente
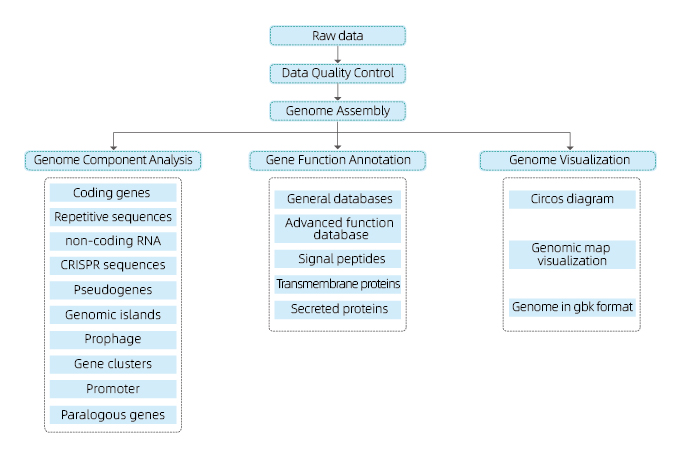
Comprend l'analyse suivante :
● Contrôle de la qualité des données de séquençage
● Assemblage du génome
● Analyse des composants du génome : prédiction du CDS et de plusieurs éléments génomiques
● Annotation fonctionnelle avec plusieurs bases de données générales (GO, KEGG, etc) et bases de données avancées (CARD, VFDB, etc)
● Visualisation du génome
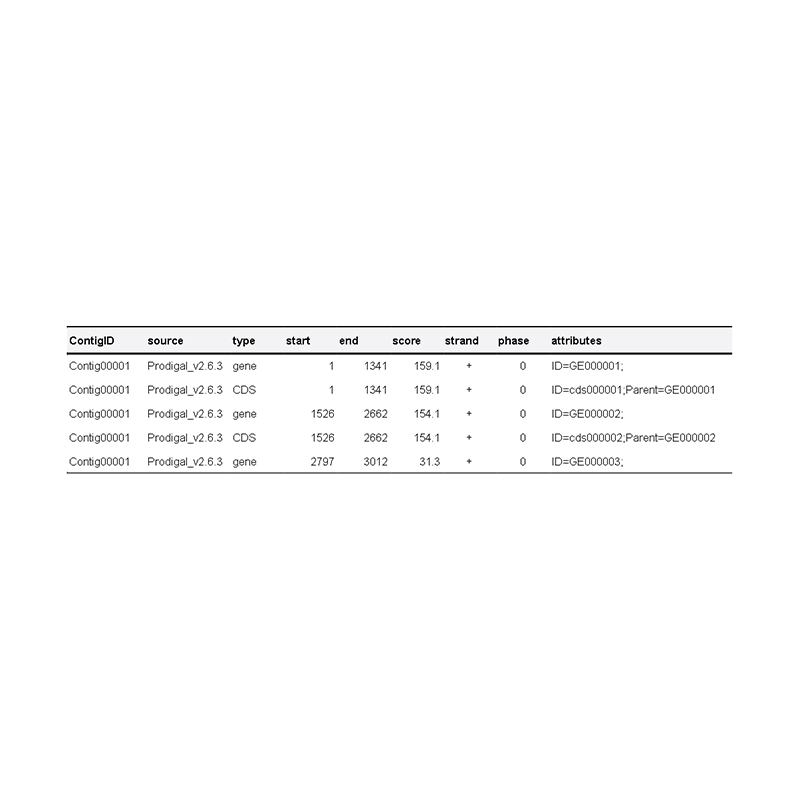
Nous fournissons le génome dans un format fasta facilement accessible et le fichier d'annotation du génome (gff).
Annotation des gènes – GO
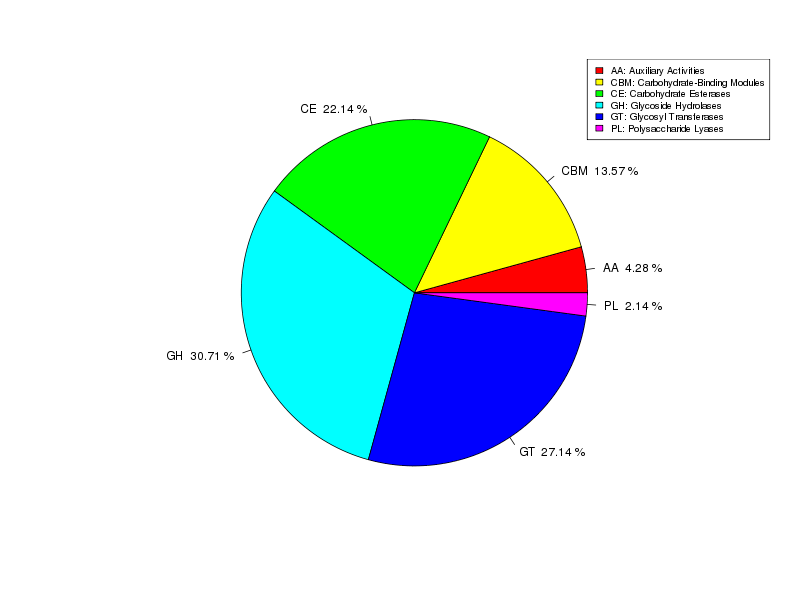
 Annotation génétique – Glucides CAZY
Annotation génétique – Glucides CAZY
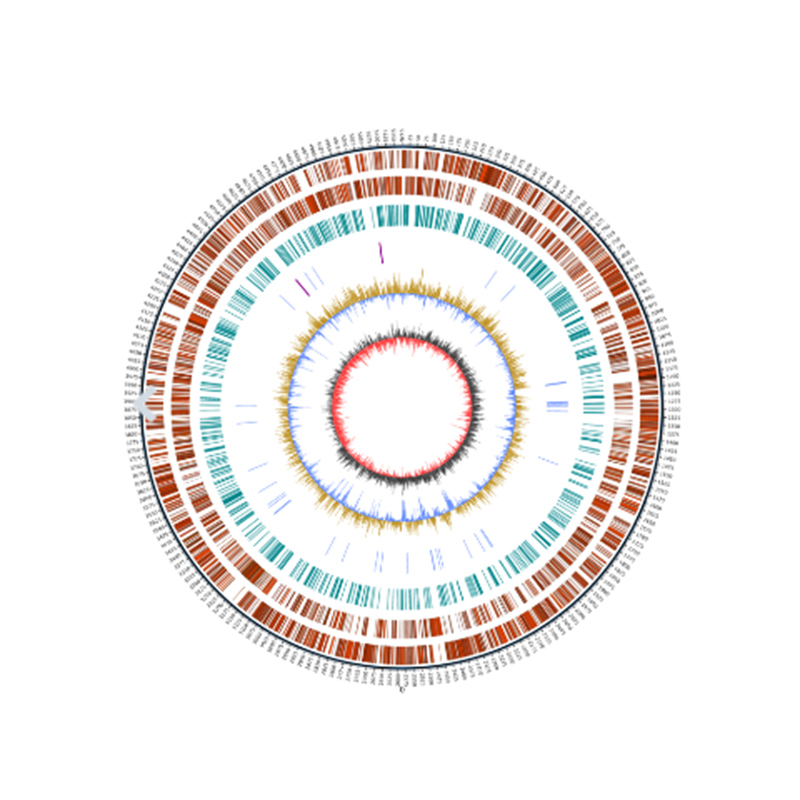
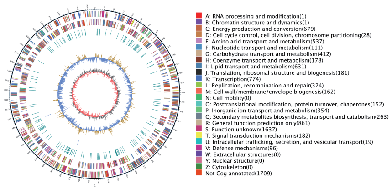
Visualisation du génome – Graphique Circos
Explorez les progrès facilités par les services d'assemblage du génome bactérien de BMKGene à travers une collection organisée de publications.
Dai, W. et coll. (2023) « Découverte de Bacteroides uniformis F18-22 en tant que bactérie probiotique sûre et nouvelle pour le traitement de la colite ulcéreuse du côlon humain sain »,Revue internationale des sciences moléculaires, 24(19), p. 14669. est ce que je : 10.3390/IJMS241914669/S1.
Kang, Q. et coll. (2021) « Isolats multirésistants de Proteus mirabilis transportant blaOXA-1 et blaNDM-1 provenant d'animaux sauvages en Chine : risque croissant pour la santé publique »,Zoologie intégrative, 16(6), pages 798 à 809. est ce que je: 10.1111/1749-4877.12510.
Wang, TT et al. (2017) « La séquence complète du génome de l'endophyte Bacillus flexus KLBMP 4941 révèle son mécanisme de promotion de la croissance des plantes et la base génétique de la tolérance au sel »,Journal de biotechnologie, 260, p. 38-41. est ce que je: 10.1016/J.JBIOTEC.2017.09.001.
Wang, X. et coll. (2021) « Les plasmides de résistance à réplicons multiples de Klebsiella assurent une diffusion étendue de gènes antimicrobiens »,Frontières de la microbiologie, 12, p. 754931. est ce que je : 10.3389/FMICB.2021.754931/BIBTEX.