Séquençage d'amplicons 16S/18S/ITS-PacBio
Caractéristiques des services
● Plateforme de séquençage : PacBio Revio
● Mode séquençage : CCS (lectures HiFi)
● Amplification de la région cible suivie d'une liaison en tandem des amplicons avant la préparation de la bibliothèque de cloches HiFi SMRT
Avantages des services
●Résolution taxonomique plus élevée : Tséquençage d'amplicons courts de Han,permettant des taux de classification OTU plus élevés au niveau des espèces.
●Appel de base très précis: Séquençage en mode PacBio CCS (lectures HiFi).
●Sans isolement: Identification rapide de la composition microbienne dans les échantillons environnementaux.
●Largement applicable: Diverses études sur la communauté microbienne.
●Analyse bioinformatique complète: Le dernier package QIIME2 (aperçu quantitatif de l'écologie microbienne) avec diverses analyses en termes de base de données, annotation, OTU/ASV.
●Une expertise étendue: Avec des milliers de projets de séquençage d'amplicons menés chaque année, BMKGENE apporte plus d'une décennie d'expérience, une équipe d'analyse hautement qualifiée, un contenu complet et un excellent support après-vente.
Spécifications des services
| Bibliothèque | Stratégie de séquençage | Données recommandées |
| Amplicon | PacBio Revio | 10/30/50 K balises (CCS) |
Exemples d'exigences
| Concentration (ng/μL) | Quantité totale (µg) | Volume (µL) |
| ≥5 | ≥0,3 | ≥20 |
Livraison d’échantillon recommandée
Congelez les échantillons dans de l'azote liquide pendant 3 à 4 heures et conservez-les dans de l'azote liquide ou à -80 degrés pour une réservation à long terme. L’envoi d’un échantillon avec de la glace carbonique est requis.
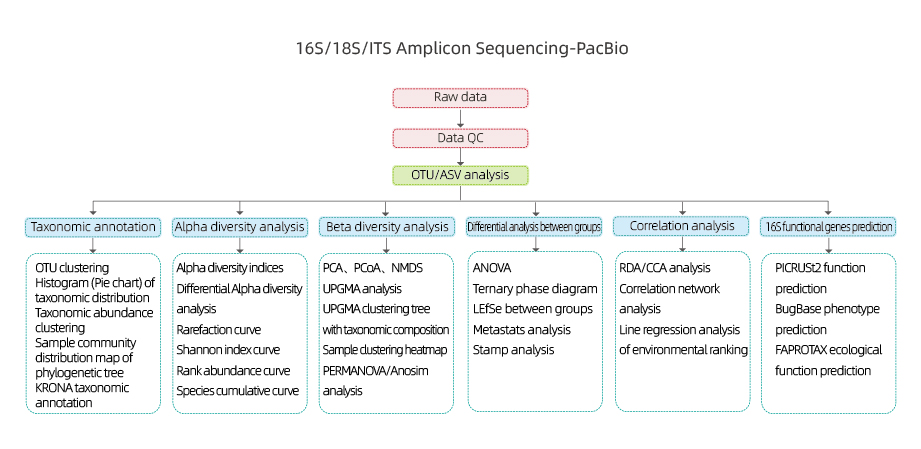
Flux de travail des services

Livraison d'échantillon

Construction d'une bibliothèque

Séquençage

Analyse des données

Services après-vente
Comprend l’analyse suivante :
●Contrôle de la qualité des données brutes
● Clustering OTU/Débruitage (ASV)
●Annotation OTU
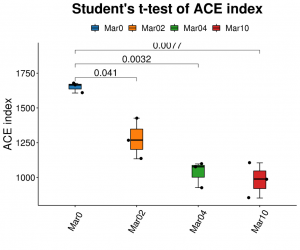
●Analyse de la diversité alpha : plusieurs index, dont Shannon, Simpson et ACE.
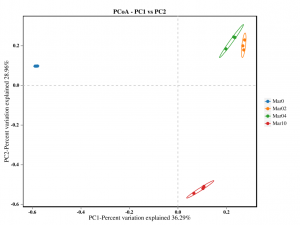
●Analyse de la diversité bêta
●Analyse inter-groupes
●Analyse de corrélation : entre les facteurs environnementaux et la composition et la diversité des OUT
● Prédiction du gène fonctionnel 16S
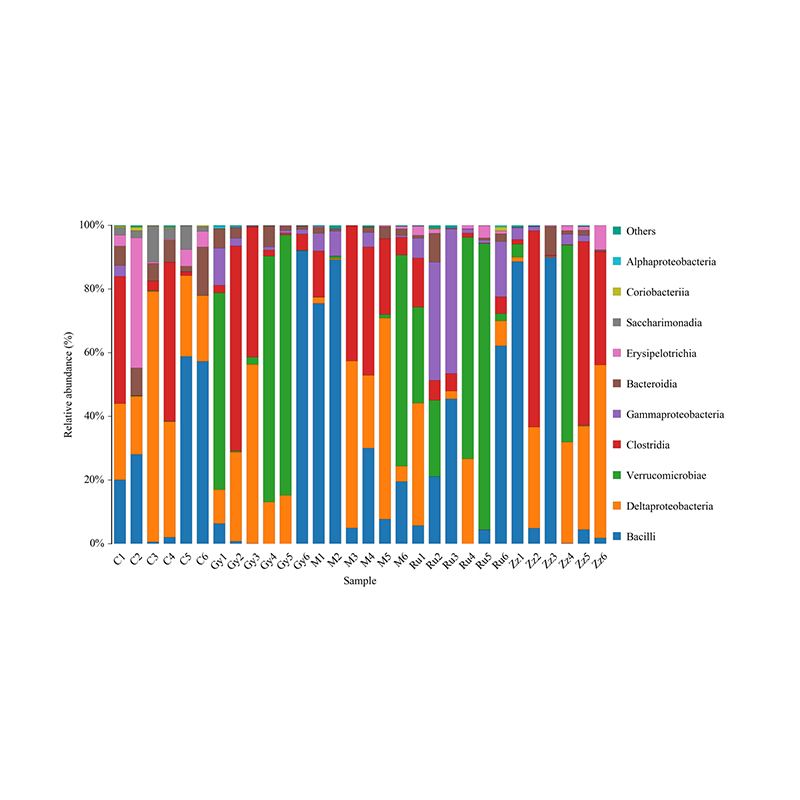
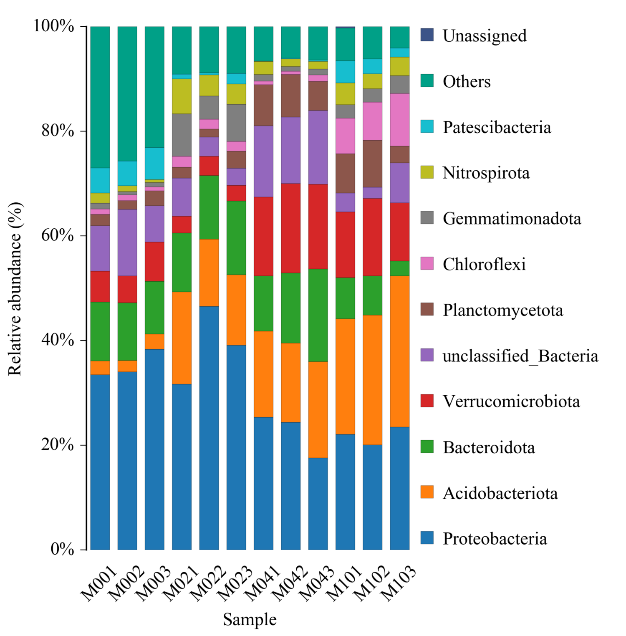
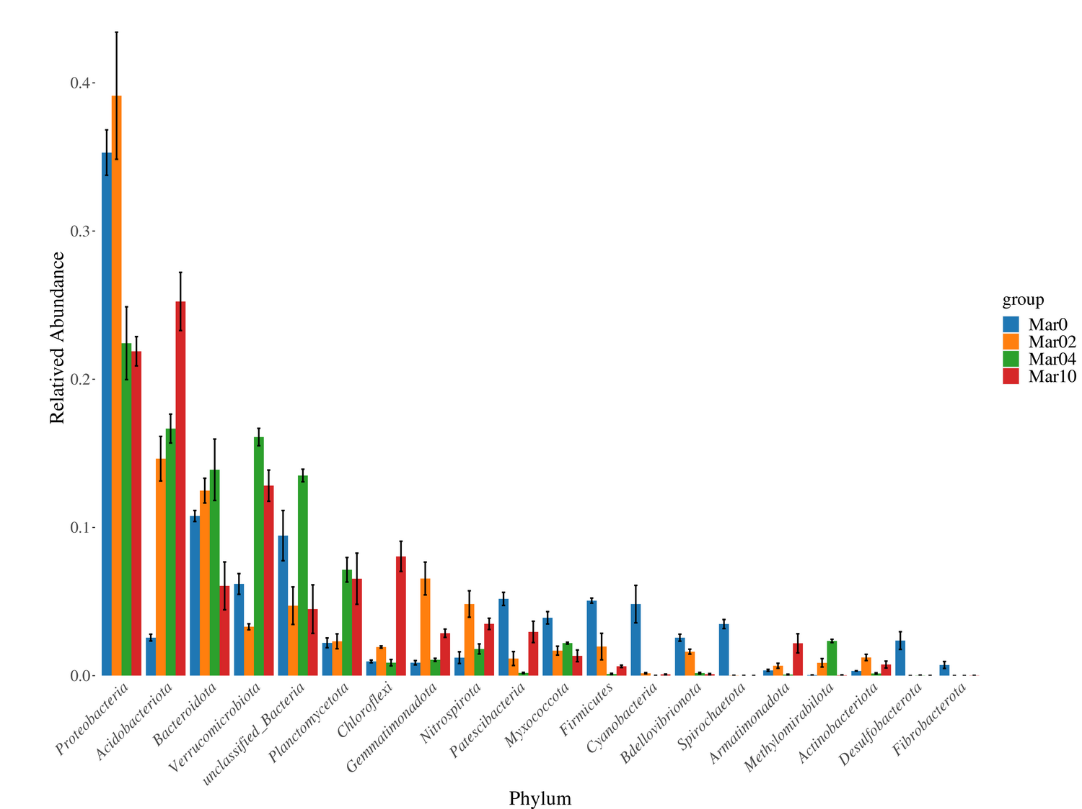
Histogramme de distribution taxonomique
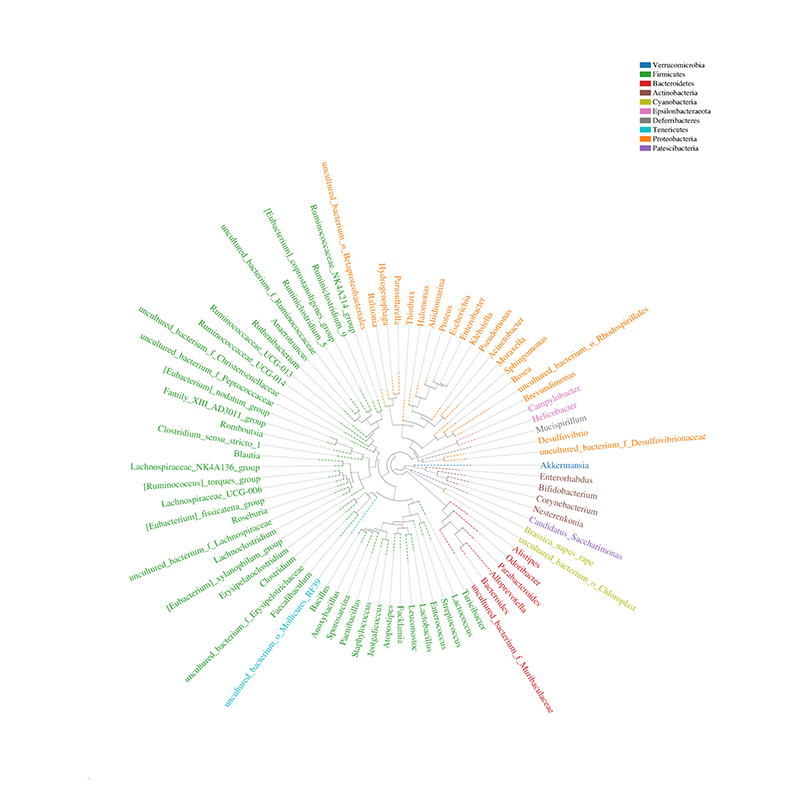
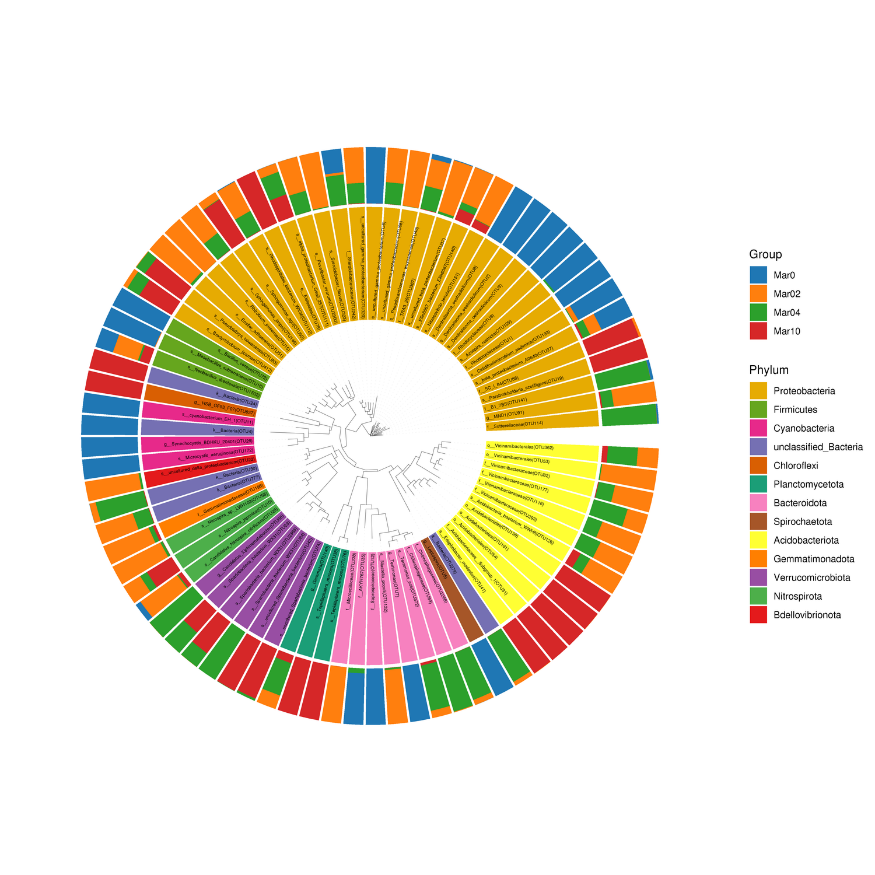
Arbre phylogénétique de répartition communautaire
Analyse de la diversité alpha : ACE
Analyse de la diversité bêta : PCoA
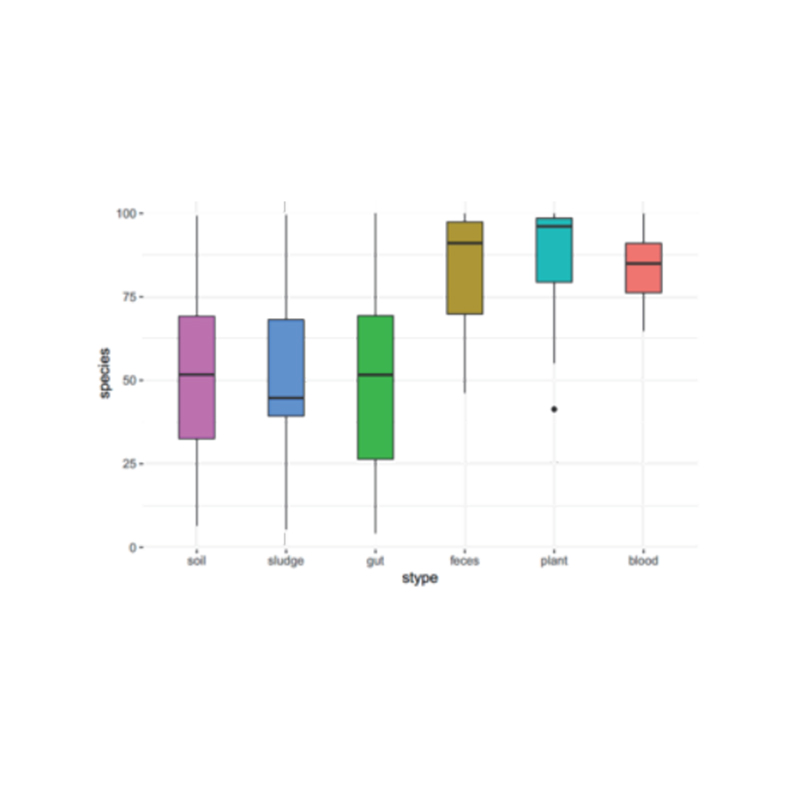
Analyse intergroupe : ANOVA
Explorez les progrès facilités par les services de séquençage d'amplicons de BMKGene avec PacBio à travers une collection organisée de publications.
Gao, X. et Wang, H. (2023) « Analyse comparative des profils et des fonctions bactériennes du rumen lors de l'adaptation à différentes phénologies (regreen vs. Grassy) chez des moutons mérinos alpins à deux stades de croissance sur une prairie alpine », Fermentation, 9 ( 1), p. 16. est ce que je : 10.3390/FERMENTATION9010016/S1.
Li, S. et coll. (2023) « Capturer la matière noire microbienne dans les sols désertiques à l'aide de la métagénomique basée sur la culture et de l'analyse à haute résolution », npj Biofilms and Microbiomes 2023 9 : 1, 9(1), pp. est ce que je: 10.1038/s41522-023-00439-8.
Mu, L. et al. (2022) « Effets des sels d'acides gras sur les caractéristiques de fermentation, la diversité bactérienne et la stabilité aérobie de l'ensilage mélangé préparé avec de la luzerne, de la paille de riz et du son de blé », Journal of the Science of Food and Agriculture, 102(4), pp. 1487. est ce que je: 10.1002/JSFA.11482.
Yang, J. et coll. (2023) « L'interaction entre les biomarqueurs du stress oxydatif et le microbiote intestinal dans les effets antioxydants des extraits de Sonchus brachyotus DC. dans Stress oxydatif intestinal induit par l'oxazolone chez le poisson zèbre adulte », Antioxydants 2023, Vol. 12, page 192, 12(1), p. 192. est ce que je : 10.3390/ANTIOX12010192.