10x transcriptome spatial de la Visium génomique
Schéma technique

Caractéristiques
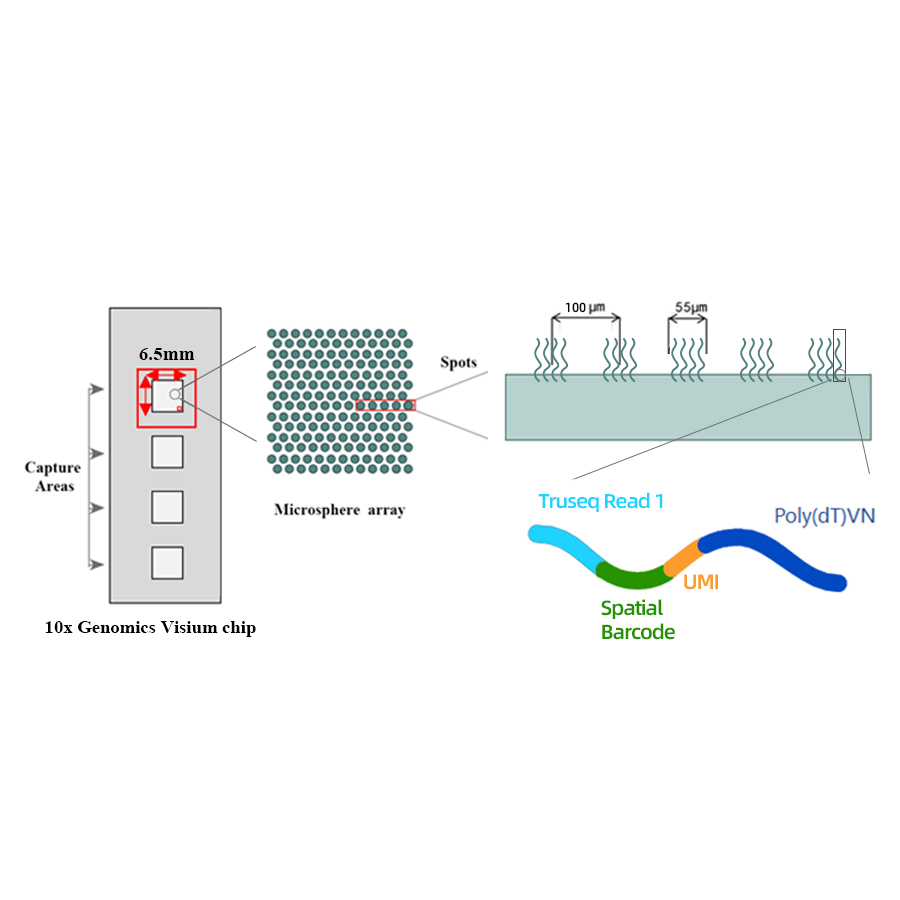
● Résolution: 100 µm
● Diamètre du point: 55 µm
● Nombre de spots: 4992
● Zone de capture: 6,5 x 6,5 mm
● Chaque tache codée à barres est chargée d'amorces composées de 4 sections:
- queue poly (dt) pour l'amorçage de l'ARNm et la synthèse d'ADNc
- Identifiant moléculaire unique (UMI) pour corriger le biais d'amplification
- code-barres spatial
- séquence de liaison de l'amorce de séquençage de lecture partielle
● Talage H&E des sections
Avantages
●Service à guichet unique: intègre toute expérience et étapes basées sur les compétences, notamment la cryo-section, la coloration, l'optimisation des tissus, le codage à barre spatial, la préparation des bibliothèques, le séquençage et la bioinformatique.
● Équipe technique hautement qualifiée: Avec une expérience dans plus de 250 types de tissus et plus de 100 espèces, notamment l'humain, la souris, les mammifères, les poissons et les plantes.
●Mise à jour en temps réel sur l'ensemble du projet: avec un contrôle total des progrès expérimentaux.
●Bioinformatique standard complète:Le package comprend 29 analyses et plus de 100 chiffres de haute qualité.
●Analyse et visualisation des données personnalisées: Disponible pour différentes demandes de recherche.
●Analyse conjointe en option avec séquençage d'ARNm unique
Caractéristiques
| Exigences d'échantillonnage | Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| Échantillons de cryo en octobre (Diamètre optimal: environ 6x6x6 mm³) 2 blocs par échantillon | Bibliothèque d'ADNc 10x Visium | Illumina PE150 | 50k PE Reads par place (60 Go) | Rin> 7 |
Pour plus de détails sur les guidages de préparation des échantillons et le flux de travail de service, n'hésitez pas à parler à unExpert BMKGene
Flux de travail de service
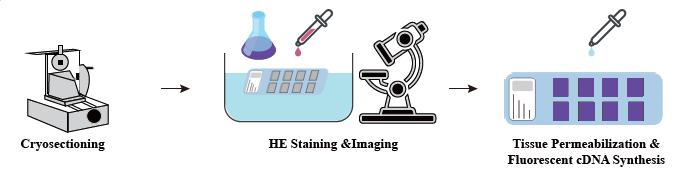
Dans la phase de préparation de l'échantillon, un essai d'extraction d'ARN en vrac initial est effectué pour garantir un ARN de haute qualité peut être obtenu. Au stade d'optimisation des tissus, les sections sont colorées et visualisées et les conditions de perméabilisation pour la libération d'ARNm à partir de tissus sont optimisées. Le protocole optimisé est ensuite appliqué lors de la construction de la bibliothèque, suivi du séquençage et de l'analyse des données.
Le flux de travail de service complet implique des mises à jour en temps réel et des confirmations du client pour maintenir une boucle de rétroaction réactive, assurant une exécution en douceur du projet.

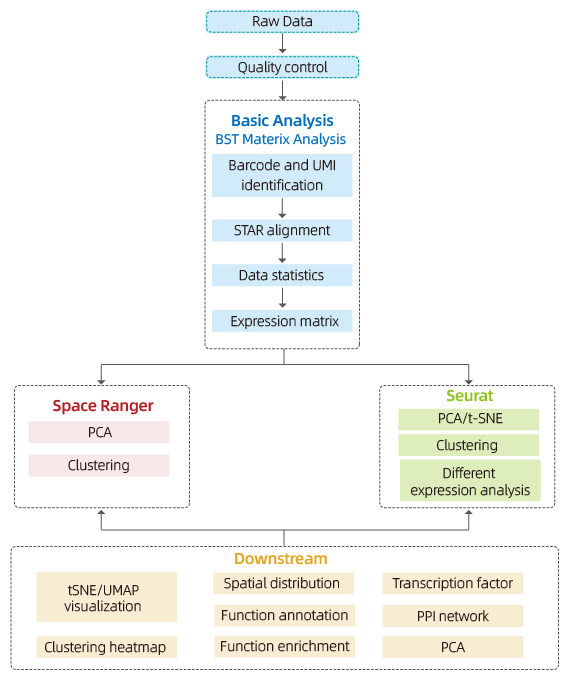
Comprend l'analyse suivante:
Contrôle de la qualité des données:
o Data Sorput and Quality Score Distribution
o Détection de gènes par place
o Couverture tissulaire
Analyse de l'échantillon intérieur:
o Richness Gene
o Clustering ponctuel, y compris une analyse de dimension réduite
o Analyse de l'expression différentielle entre les grappes: identification des gènes marqueurs
o Annotation fonctionnelle et enrichissement des gènes marqueurs
Analyse inter-groupe
o Re-combinaison des taches des deux échantillons (par exemple, malades et contrôle) et re-cluster
o Identification des gènes marqueurs pour chaque cluster
o Annotation fonctionnelle et enrichissement des gènes marqueurs
o Expression différentielle du même cluster entre les groupes
Analyse de l'échantillon intérieur
Clustering ponctuel

Identification des gènes marqueurs et distribution spatiale


Analyse inter-groupe
Combinaison de données des deux groupes et re-cluster
Gènes marqueurs de nouveaux grappes
Explorez les progrès facilités par le service de transcriptomique spatial de BMKGene par 10x Visium dans ces publications en vedette:
Chen, D. et al. (2023) 'MTHL1, un homologue potentiel de drosophile des GPCR d'adhésion des mammifères, est impliqué dans les réactions antitumorales aux cellules oncogéniques injectées dans les mouches',Actes de l'Académie nationale des sciences des États-Unis d'Amérique, 120 (30), p. E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) «L'acier permet une délimitation à haute résolution des données transcriptomiques spatio-temporelles»,Briefings en bioinformatique, 24 (2), pp. 1–10. doi: 10.1093 / bib / bbad068.
Liu, C. et al. (2022) «Un atlas spatio-temporel de l'organogenèse dans le développement des fleurs d'orchidées»,Recherche des acides nucléiques, 50 (17), pp. 9724–9737. doi: 10.1093 / nar / gkac773.
Wang, J. et al. (2023) `` L'intégration de la transcriptomique spatiale et du séquençage d'ARN unique révèle les stratégies thérapeutiques potentielles pour le léiomyome utérin '',Journal international des sciences biologiques, 19 (8), pp. 2515–2530. doi: 10.7150 / ijbs.83510.