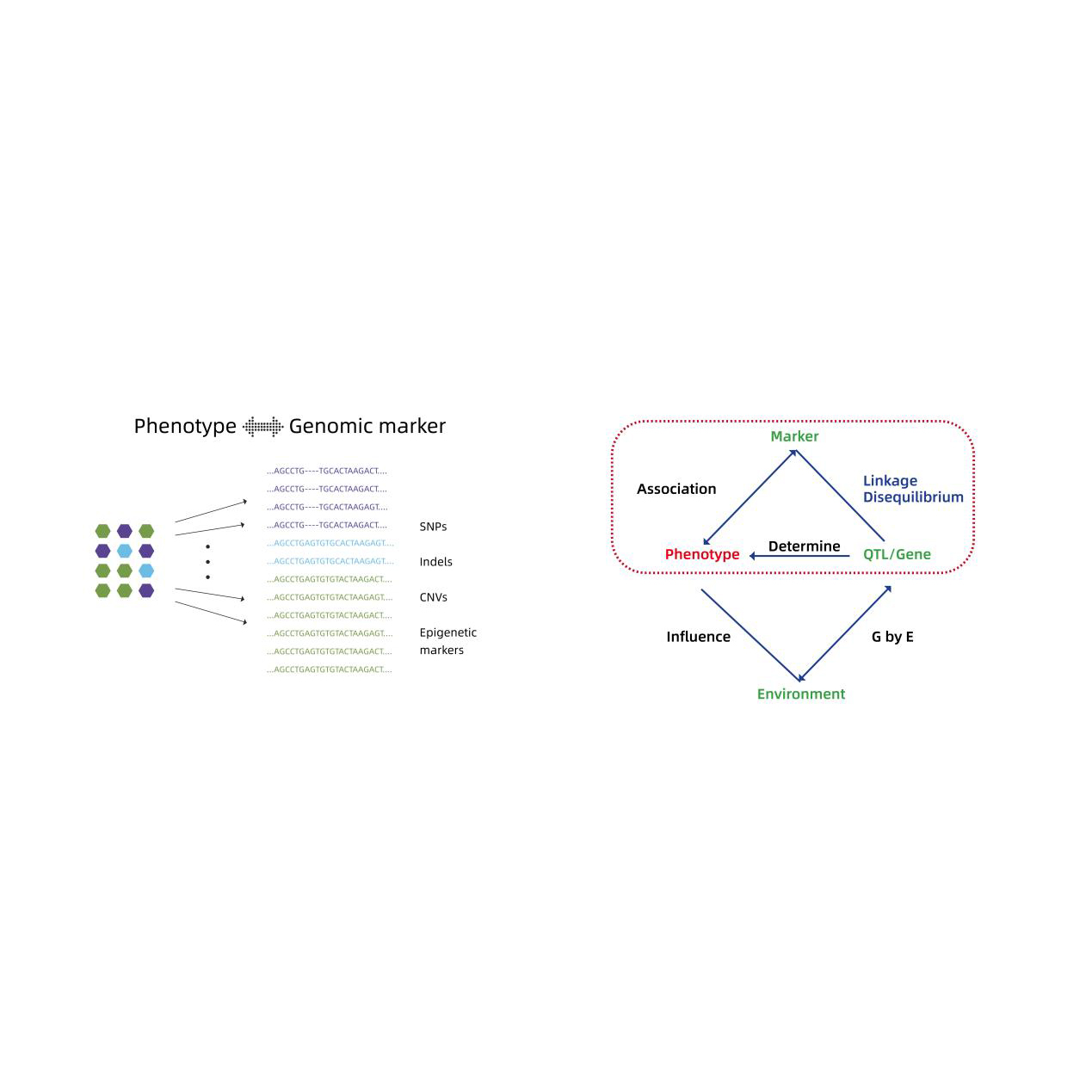
Genomin laajuinen assosiaatioanalyysi
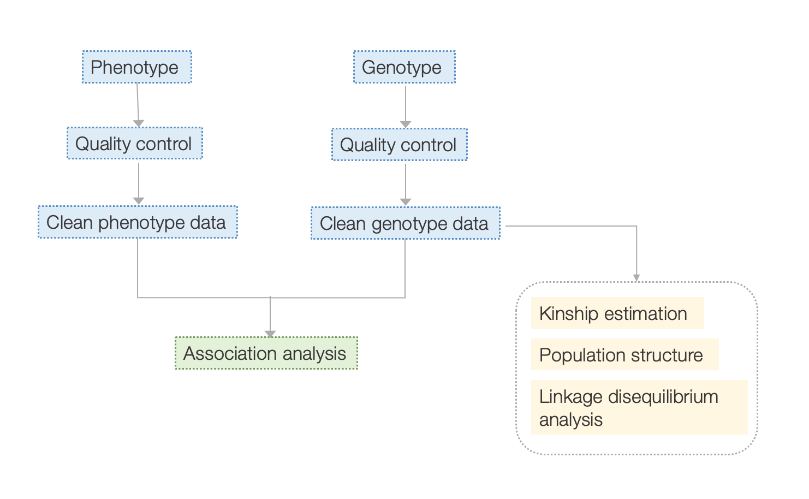
Työnkulku

Palvelun edut
●Laaja asiantuntemus- ja julkaisutiedot: Bmkgene on kertynyt kokemusta GWAS: sta, ja se on suorittanut satoja lajiprojekteja populaation GWAS -tutkimuksessa, avustetut tutkijat julkaisemaan yli 100 artikkelia ja kumulatiivinen vaikutustekijä saavutti 500.
● Kattava bioinformatiikan analyysi: Työnkulku sisältää SNP-piirin assosiaatioanalyysin, ehdokasgeenien ja vastaavan funktionaalisen merkinnän toimittamisen.
●Korkeasti koulutettu bioinformatiikan tiimi ja lyhyt analyysisykli: BMKGENE: n tiimi tarjoaa suuren kokemuksen edistyneestä genomianalyysistä kattavia analyysejä nopealla käännösajalla.
●Mahtamisen jälkeinen tuki:Sitoumuksemme ulottuu projektin valmistumisen jälkeen 3 kuukauden jälkeisellä palvelujaksolla. Tänä aikana tarjoamme projektin seurantaa, vianetsintäapua sekä Q & A-istuntoja tuloksiin liittyvien kyselyjen ratkaisemiseksi.
Palvelun eritelmät ja vaatimukset
| Sekvensointityyppi | Suositeltu väestöasteikko | Sekvensointistrategia | Nukleotidivaatimukset |
| Koko genomin sekvensointi | 200 näytettä | 10x | Pitoisuus: ≥ 1 ng/ µl Kokonaismäärä ≥ 30 ng Rajoitettu tai ei mitään hajoamista tai saastumista |
| Spesifinen-locus monistettu fragmentti (SLAF) | Tunnisteiden syvyys: 10x Tunnisteiden lukumäärä: <400 Mt: WGS suositellaan <1 Gt: 100K -tunnisteet 1 Gt > 2 Gt: 300K -tunnisteet Max 500K -tunnisteet | Pitoisuus ≥ 5 ng/µl Kokonaismäärä ≥ 80 ng Nanodrop OD260/280 = 1,6-2,5 Agaroosigeeli: Ei rajoitettua hajoamista tai saastumista
|
Materiaalivalinta



Eri lajikkeet, alalajit, maalajikkeet/geenikankit/sekoitetut perheet/villit resurssit
Eri lajikkeet, alalajit, maalajikkeet
Puoli-Sib-perhe/täys Sib-perhe/villit resurssit
Huoltotyövirta

Kokeen suunnittelu

Näytteenotto

RNA: n uutto

Kirjaston rakennus

Sekvensointi

Tietojen analysointi

Myynnin jälkeiset palvelut
Sisältää seuraavan analyysin:
- Genomin laajuinen assosiaatioanalyysi: LM, LMM, Emmmax, Fastlmm-malli
- Ehdokasgeenien toiminnallinen merkintä
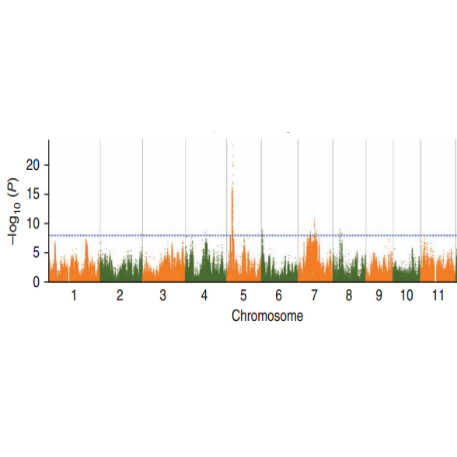
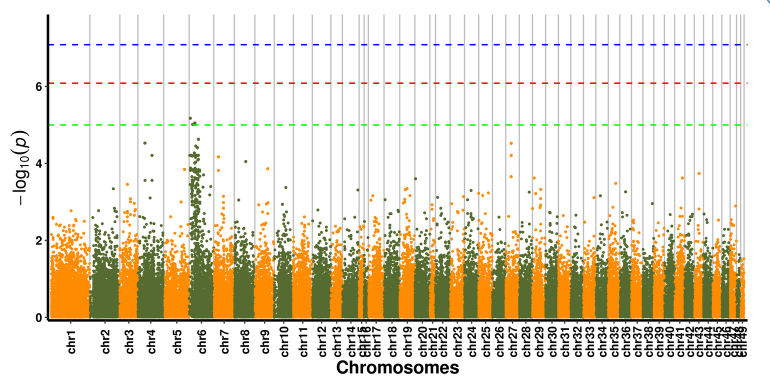
SNP-piirin assosiaatioanalyysi-Manhattan-juoni
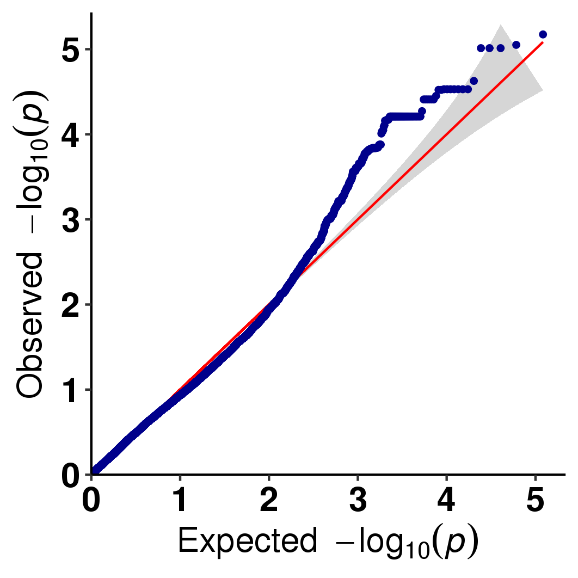
SNP-piirin assosiaatioanalyysi-QQ-kuvaaja
Tutustu Bmkgenen De Gwas -palvelujen helpottamiin edistyksiin kuratoidun julkaisukokoelman kautta:
LV, L. et ai. (2023) 'käsitys ammoniakkitoleranssin geneettisestä perustana partakoneen simpukassa sinonovacula contricta genomin laajuisen assosiaatiotutkimuksen avulla ",Vesiviljely, 569, s. 739351. Doi: 10.1016/j.aquaculture.2023.739351.
Li, X. et ai. (2022) '398 foxtail-hirssi -juttujen monimuotoiset analyysit paljastavat genomiset alueet, jotka liittyvät kodistukseen, metaboliittimuotoihin ja anti-inflammatorisiin vaikutuksiin',Molekyylikasvi, 15 (8), s. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et ai. (2022) 'Hullessin genomin laajuinen assosiaatio kartoitus tuskin fenotyyppejä kuivuusympäristössä',Kasvitieteen rajat, 13, s. 924892. Doi: 10.3389/fpls.2022.924892/bibtex.
Zhao, X. et ai. (2021) 'GMST1, joka koodaa sulfotransferaasia, antaa resistenssin soijapavun mosaiikkiviruskannoille G2 ja G3',Kasvi, solu ja ympäristö, 44 (8), s. 2777–2792. doi: 10.1111/pce.14066.