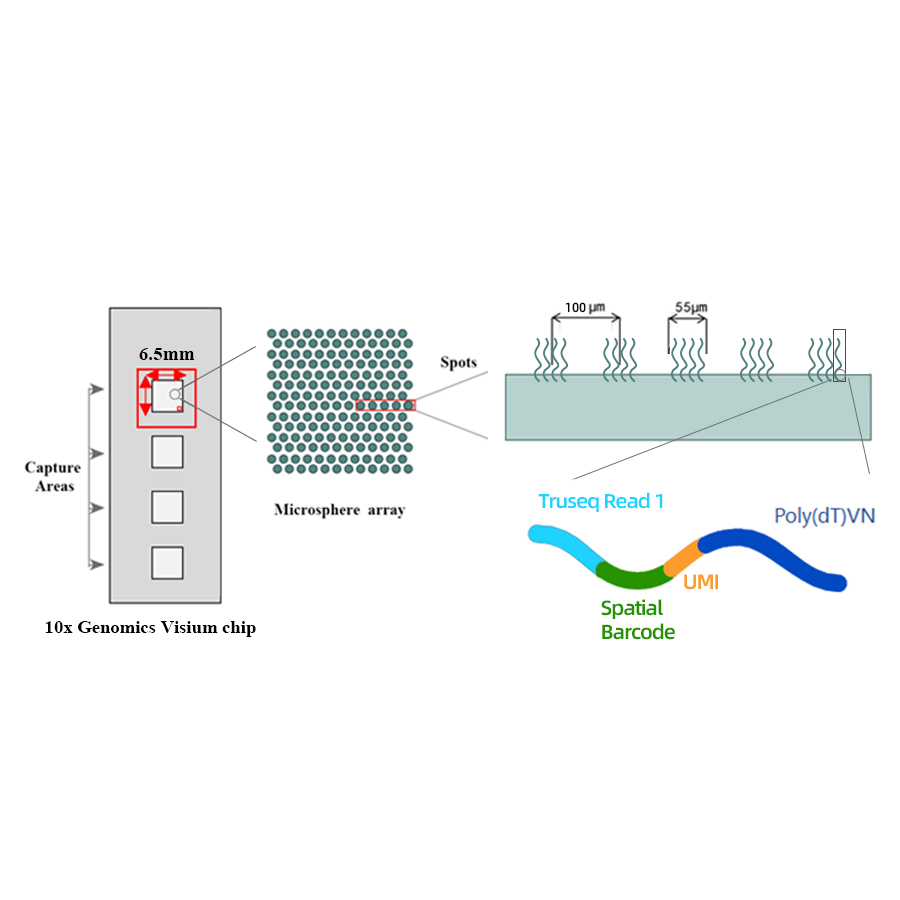
10x Genomics Visium Transcriptom espaziala
Erregimen Teknikoa

Osau
● Ebazpena: 100 μm
● Leku diametroa: 55 μm
● Leku kopurua: 4992
● Capture gunea: 6,5 x 6,5 mm
● Bakartutako leku bakoitza 4 atalez osatutako primariekin kargatzen da:
- MRNA PRIMING ETA CDNA SINTESIAREN AURREKO POLY (DT)
- Identifikatzaile molekular bakarra (UMI) anplifikazio bias zuzentzeko
- Barkode espaziala
- Irakurri partialaren sekuentzia loteslea 1 sekuentziazio primer
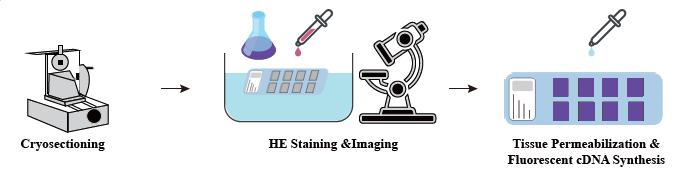
● H & E atalen orbanatzea
Abantailak
●Geldialdi bakarreko zerbitzua: Esperientzia eta trebetasunetan oinarritutako urrats guztiak barneratzen ditu, besteak beste, Cryo-Atting, Ehonting, Ehunen Optimizazioa, Espazial Barcoding, Liburutegiko prestaketa, Sekuentziazioa eta Bioinformatika barne.
● Oso trebea den talde teknikoa: 250 ehun mota baino gehiago eta 100+ espezie baino gehiagoko esperientziak, gizakiak, sagua, ugaztunak, arrainak eta landareak barne.
●Proiektu osoan zehar denbora errealeko eguneratzea: aurrerapen esperimentalaren kontrol osoa.
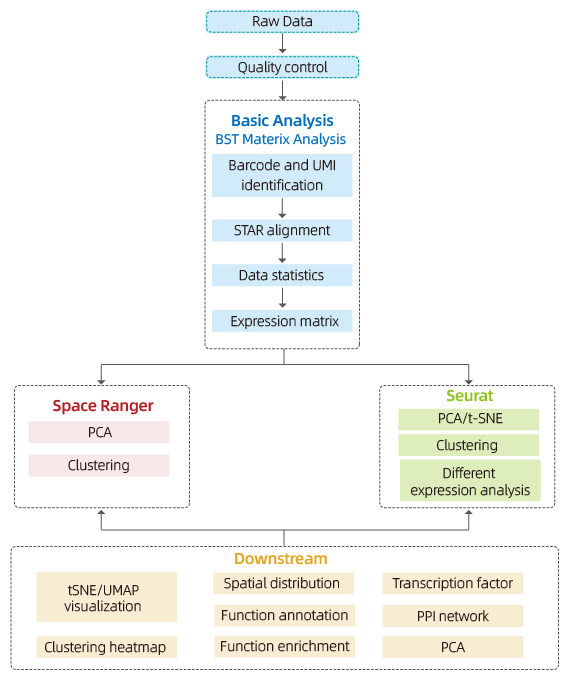
●Bioinformatika estandar integrala:Paketeak 29 analisi eta kalitate handiko 100 figura ditu.
●Datuen azterketa pertsonalizatua eta bistaratzea: Ikerketa eskaera desberdinetarako erabilgarri.
●Aukerako analisi bateratua zelula bakarreko MRNA sekuentziazioarekin
Zehaztapenak
| Laginaren baldintzak | Liburutegi | Sekuentziazio estrategia | Gomendatutako datuak | Kalitatearen kontrola |
| Urrezko kapsulatutako cryo laginak (Diametro optimoa: gutxi gorabehera 6x6x6 mm³) Lagin bakoitzeko 2 bloke | 10x Viium CDNA Liburutegia | Illumina pe150 | 50k pe leku bakoitzeko irakurtzen da (60GB) | Rin> 7 |
Laginak prestatzeko orientazio eta zerbitzu-fluxuei buruzko xehetasun gehiago lortzeko, jar zaitez aske hitz egitekoBMKGENE Aditua
Zerbitzuaren fluxua
Laginak prestatzeko fasean, hasierako RNA erauzteko epaiketa egiten da kalitate handiko RNA lor daitekeela ziurtatzeko. Ehunen optimizazioan etapa atalak zikindu eta bistaratzen dira eta mRNA ehunetik kaleratzeko baldintza baldintzak optimizatzen dira. Protokolo optimizatua liburutegiaren eraikuntzan aplikatzen da eta ondoren sekuentziazio eta datuen analisia eginez.
Zerbitzu osoko lan-fluxuak denbora errealeko eguneratzeak eta bezeroen berrespena dakar, erantzun-erantzunaren begizta erantzuteko, proiektuaren exekuzio leuna bermatuz.

Analisi hau barne hartzen du:
Datuen kalitatearen kontrola:
o Datuen irteera eta kalitatearen puntuazioa banatzea
o Gene detekzioa leku bakoitzeko
o Ehunen estaldura
Barruko laginaren azterketa:
o Gene aberastasuna
o Leku klusterizazioa, dimentsio analisia murriztua barne
o Klusterren arteko adierazpen diferentzialaren azterketa: markatzaile geneak identifikatzea
o Markatzaileen geneen oharpen funtzionala eta aberastea
Talde arteko analisia
o Laginak bi laginetatik (adibidez, gaixotasuna eta kontrola) eta berriro klusterra
o Kluster bakoitzeko markatzaile geneak identifikatzea
o Markatzaileen geneen oharpen funtzionala eta aberastea
o Taldeen arteko kluster beraren adierazpen diferentziala
Barruko laginaren azterketa
Leku klustering

Markatzailearen geneen identifikazioa eta banaketa espaziala


Talde arteko analisia
Bi taldeen eta berriro klusterren datuak
Kluster berrien geneak markatzaile
Arakatu BMKGene-ren espazialen transkriptomikoen zerbitzuak 10x-ek egindako argitalpen hauetan:
Chen, D. et al. (2023) 'MTHL1, mami-atxikimenduaren gpcrs-eko drosophila homologo potentziala da, eulien zelula onkogenikoetan injektatutako erreakzioetan parte hartzen duAmeriketako Estatu Batuetako Estatu Batuetako Akademia Nazionalaren prozedurak, 120 (30), or. E2303462120. DOI: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'Altzairuak bereizmen handiko Datu Transkriptomikoen Datu Espatemologikoen Delinetea gaitzen du',Bioinformatikaren inguruko informazioa, 24 (2), 1-10. DOI: 10.1093 / BIB / BBAD068.
Liu, C. et al. (2022) 'Organogenesiaren atlas spatemporal bat lore orkidea garatzen ari da',Azido nukleikoen ikerketa, 50 (17), 9724-9737 PP. DOI: 10.1093 / NAR / GKAC773.
Wang, J. et al. (2023) 'Transkriptomika espazialak eta nukleo bakarreko RNA sekuentziazioa integratzeak Umetokiko Leiomyoma-ren estrategia terapeutiko potentzialak agerian uzten ditu,Zientzia Biologikoen Nazioarteko Aldizkaria, 19 (8), 2515-2530. or. DOI: 10.7150 / IJBS.83510.