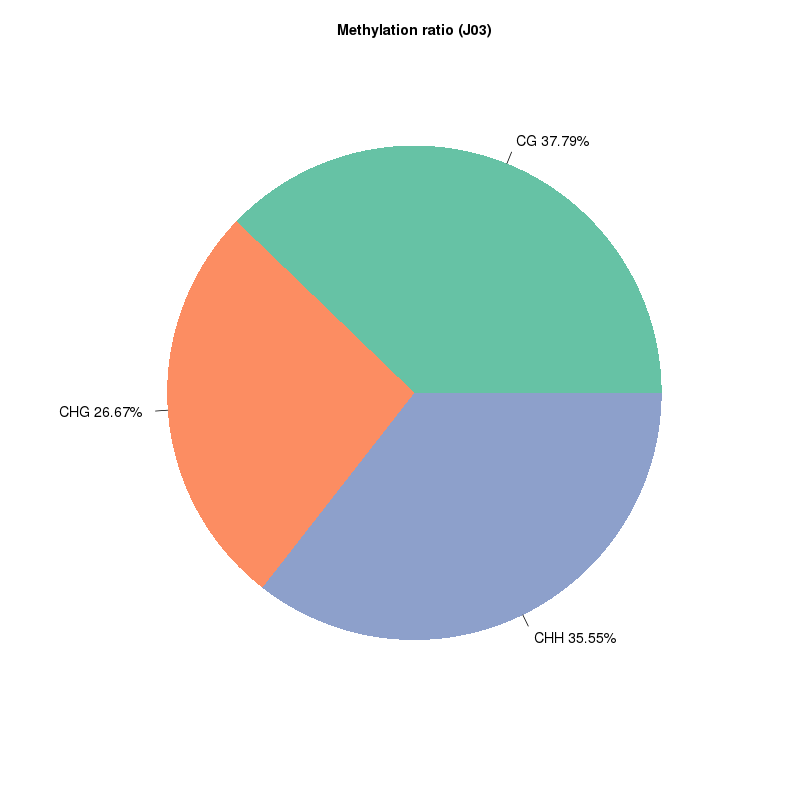Sequenciación completa del genoma del genoma (WGBS)
Características de servicio
● Requiere un genoma de referencia.
● Se agrega ADN lambda para monitorear la eficiencia de conversión de bisulfito.
● Secuenciación en Illumina Novaseq.
Ventajas de servicio
●Estándar de oro para la investigación de metilación del ADN: Esta tecnología de procesamiento de conversión de metilación madura tiene una alta precisión y buena reproducibilidad.
●Cobertura amplia y resolución de una sola base:Detección de sitios de metilación a nivel de todo el genoma.
●Plataforma completa:Proporcione un excelente servicio único del procesamiento de muestras, construcción de la biblioteca, secuenciación al análisis bioinformático.
●Experiencia extensa: Con los proyectos de secuenciación de WGBS completados con éxito en una amplia gama de especies, Bmkgene trae más de una década de experiencia, un equipo de análisis altamente calificado, contenido integral y un excelente apoyo posterior a las ventas.
●Posibilidad de unirse al análisis transcriptómico: Permitir el análisis integrado de WGB con otros datos de OMICS como RNA-seq.
Especificaciones de muestra
| Biblioteca | Estrategia de secuenciación | Salida de datos recomendado | Control de calidad |
| Bisulfito tratado | Illumina PE150 | 30x de profundidad | Q30 ≥ 85% Conversión de bisulfito> 99% |
Requisitos de muestra
| Concentración (ng/µl) | Cantidad total (µg) | Requisitos adicionales | |
| ADN genómico | ≥ 5 | ≥ 400 ng | Degradación o contaminación limitada |
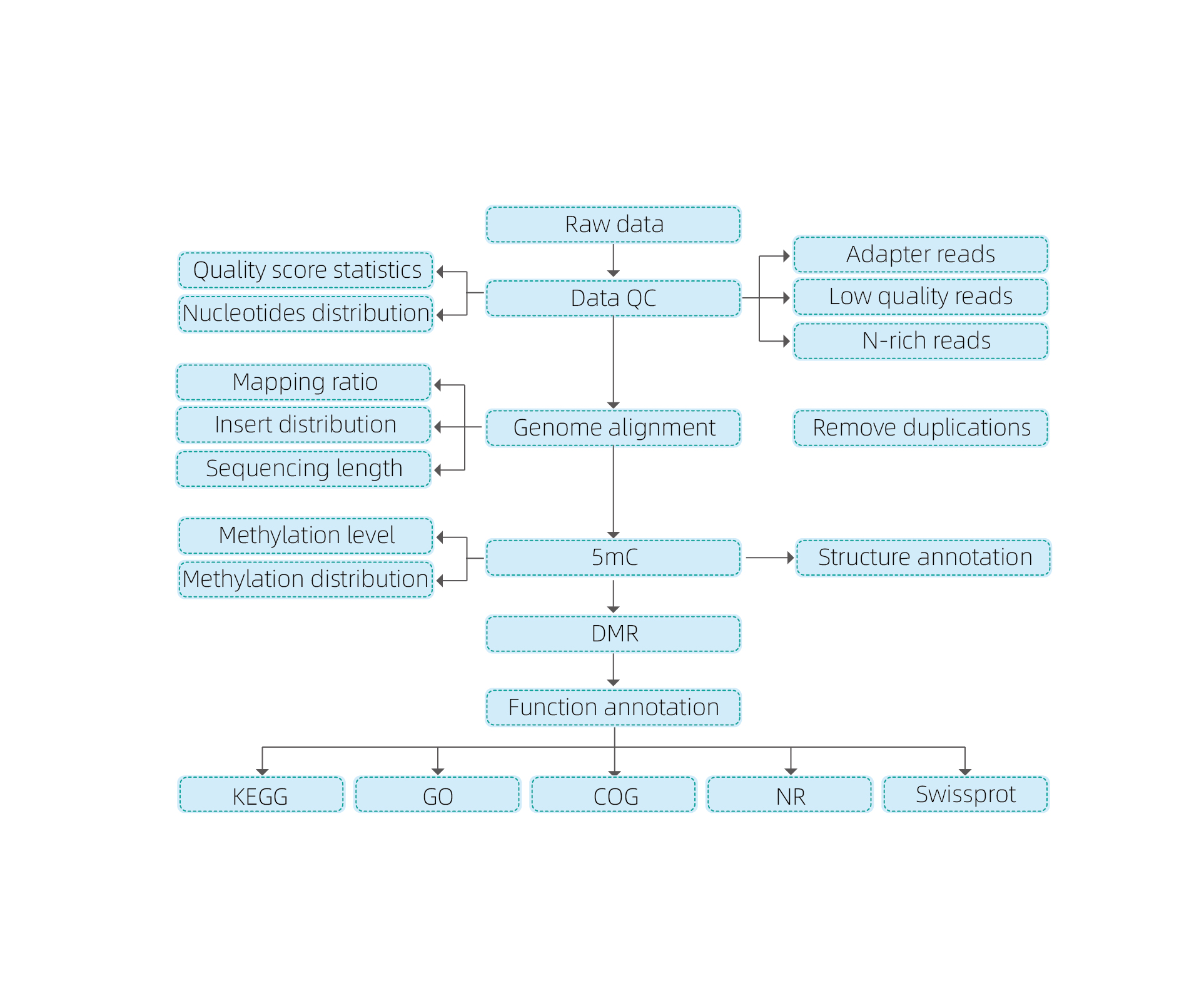
Flujo de trabajo de servicio

Entrega de muestra

Extracción de ADN

Construcción de la biblioteca

Secuenciación

Análisis de datos

Entrega de datos
Incluye el siguiente análisis:
● Control de calidad de secuenciación en bruto;
● Mapeo para hacer referencia al genoma;
● Detección de bases metiladas de 5 mC;
● Análisis de distribución de metilación y anotación;
● Análisis de regiones metiladas diferencialmente (DMR);
● Anotación funcional de genes asociados a DMRS.
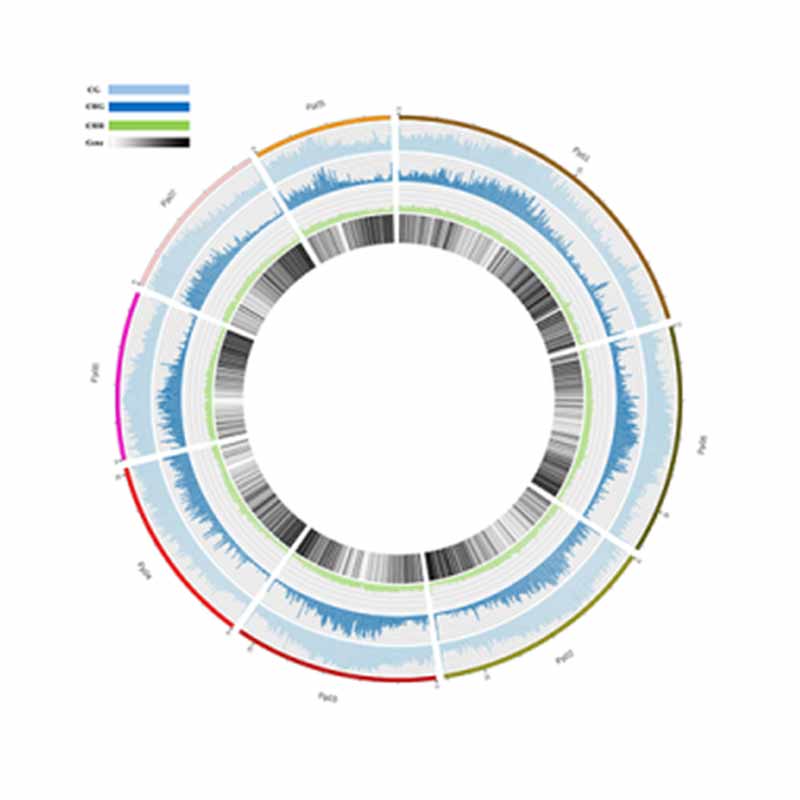
Detección de metilación de 5MC: tipos de sitios metilados
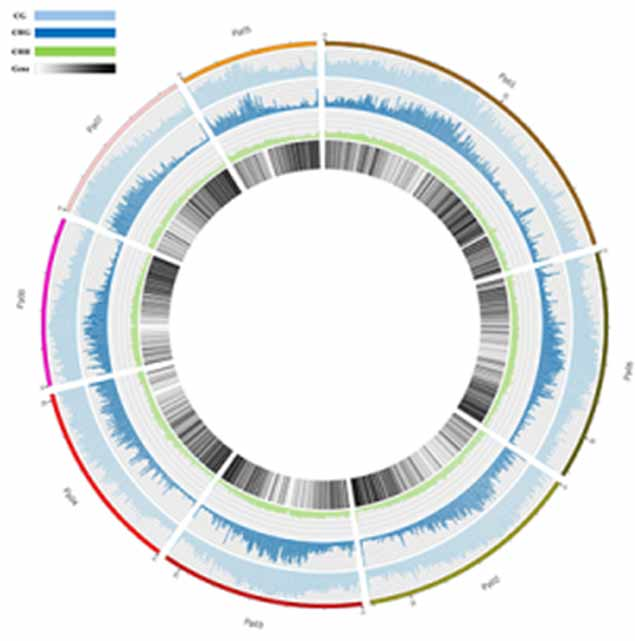
Mapa de metilación. Distribución del genoma de metilación de 5 mc
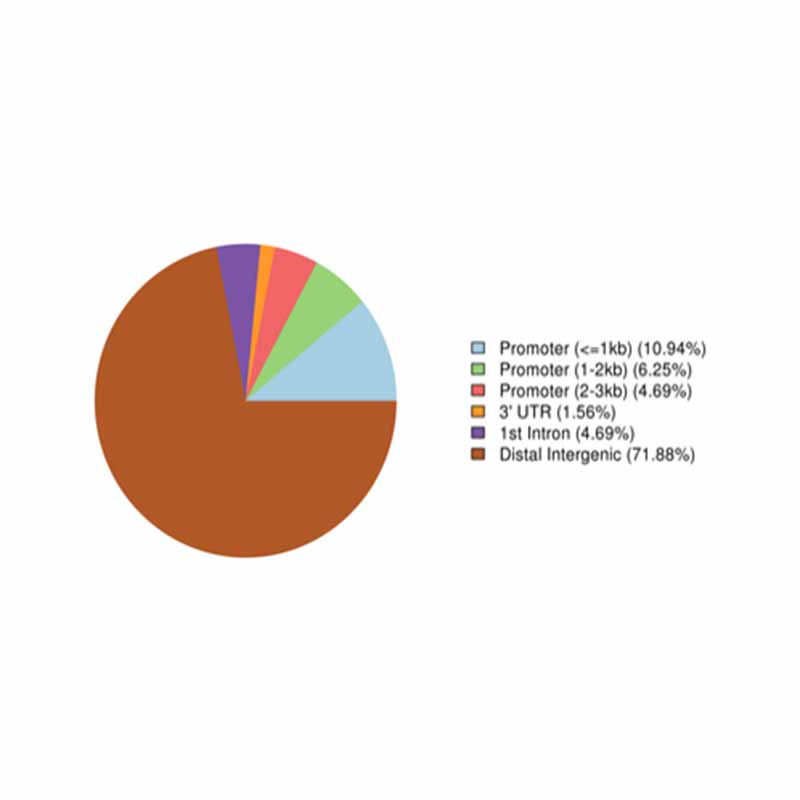
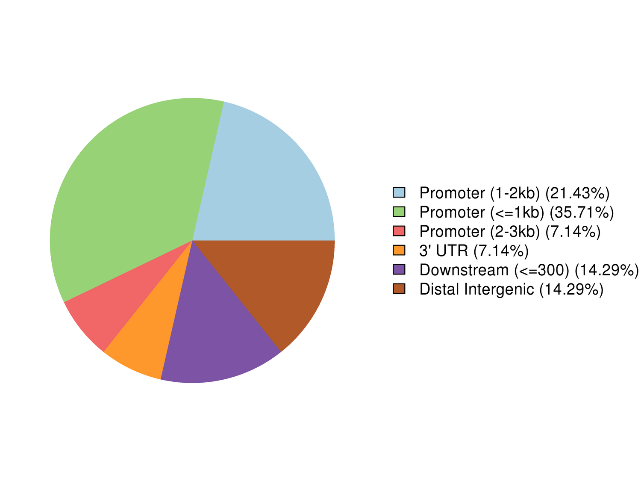
Anotación de regiones altamente metiladas
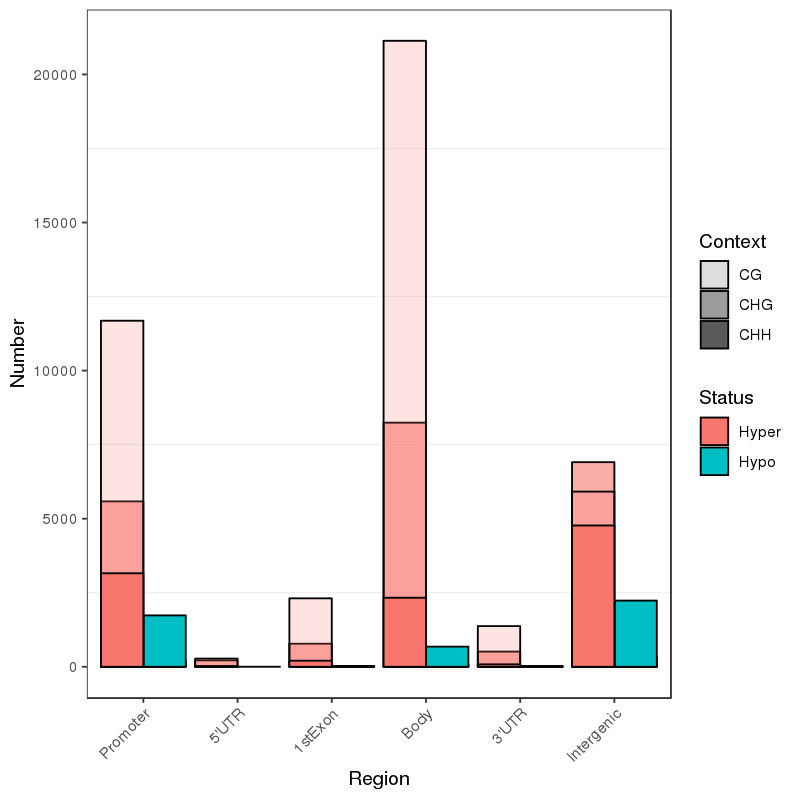
Regiones metiladas diferencialmente: genes asociados
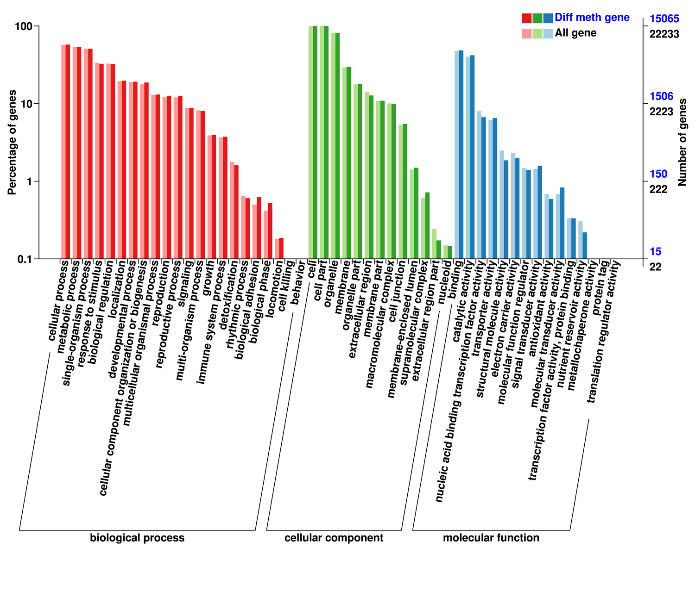
Regiones metiladas diferencialmente: anotación de genes asociados (ontología genética)
Explore los avances de investigación facilitados por los servicios de secuenciación de bisulfito de genoma completo de Bmkgene a través de una colección curada de publicaciones.
Fan, Y. et al. (2020) 'Análisis de los perfiles de metilación del ADN durante el desarrollo del músculo esquelético de ovejas utilizando secuenciación de bisulfito de genoma completo',Genómica de BMC, 21 (1), págs. 1-15. doi: 10.1186/s12864-020-6751-5.
Zhao, X. et al. (2022) 'Nuevo perturbaciones de metilación de ácido desoxirribonucleico en trabajadores expuestos al cloruro de vinilo',Toxicología y salud industrial, 38 (7), pp. 377–388. doi: 10.1177/07482337221098600
Zuo, J. et al. (2020) 'Relaciones entre la metilación del genoma, niveles de ARN no codificantes, ARNm y metabolitos en la maduración de la fruta de tomate',El diario de la planta, 103 (3), pp. 980–994. doi: 10.1111/tpj.14778.