Pequeña secuenciación de ARN-ilumina
Características
● La preparación de la biblioteca incluye un paso de selección de tamaño
● El análisis bioinformático se centró en la predicción de miRNA y sus objetivos
Ventajas de servicio
●Análisis bioinformático integral:Habilitando la identificación de miRNA conocidos y novedosos, identificación de objetivos de miRNAs y anotación funcional correspondiente y enriquecimiento con múltiples bases de datos (KEGG, GO)
●Control de calidad riguroso: Implementamos puntos de control básicos en todas las etapas, desde la preparación de la muestra y la biblioteca hasta la secuenciación y la bioinformática. Este monitoreo meticuloso garantiza la entrega de resultados consistentemente de alta calidad.
●Soporte post-ventas: Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio posterior a la venta de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia de problemas de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
●Experiencia extensa: Con un historial de cierre con éxito sobre múltiples proyectos de SRNA que cubren más de 300 especies en varios dominios de investigación, nuestro equipo aporta una gran experiencia a cada proyecto.
Requisitos de muestra y entrega
| Biblioteca | Plataforma | Datos recomendados | Data QC |
| Tamaño seleccionado | Illumina SE50 | Lecturas de 10m-20m | Q30≥85% |
Requisitos de muestra:
Nucleótidos:
| Conc. (Ng/μL) | Cantidad (μg) | Pureza | Integridad |
| ≥ 80 | ≥ 0.8 | OD260/280 = 1.7-2.5 OD260/230 = 0.5-2.5 Contaminación limitada o sin proteína o ADN que se muestra en el gel. | Rin≥6.0; 5.0≥28s/18S≥1.0; Elevación limitada o no de referencia |
● Plantas:
Raíz, tallo o pétalo: 450 mg
Hoja o semilla: 300 mg
Fruta: 1.2 g
● Animal:
Corazón o intestino: 450 mg
Viscera o cerebro: 240 mg
Músculo: 600 mg
Huesos, cabello o piel: 1.5g
● Artrópodos:
Insectos: 9G
Crustácea: 450 mg
● Sangre entera: 2 tubos
● Células: 106 células
● Sero y plasma:6 ml
Entrega de muestra recomendada
Contenedor: 2 ml de tubo de centrífuga (no se recomienda la láminas de aluminio)
Etiquetado de muestra: grupo+replicar, por ejemplo, A1, A2, A3; B1, B2, B3.
Envío:
1. Picla de secado: las muestras deben empacarse en bolsas y enterrarse en hielo seco.
2. Tubos rnastables: las muestras de ARN se pueden secar en el tubo de estabilización de ARN (por ejemplo, RNASTable®) y enviar a temperatura ambiente.
Flujo de trabajo de servicio

Diseño de experimentos

Entrega de muestra

Extracción de ARN

Construcción de la biblioteca

Secuenciación

Análisis de datos

Servicios posteriores
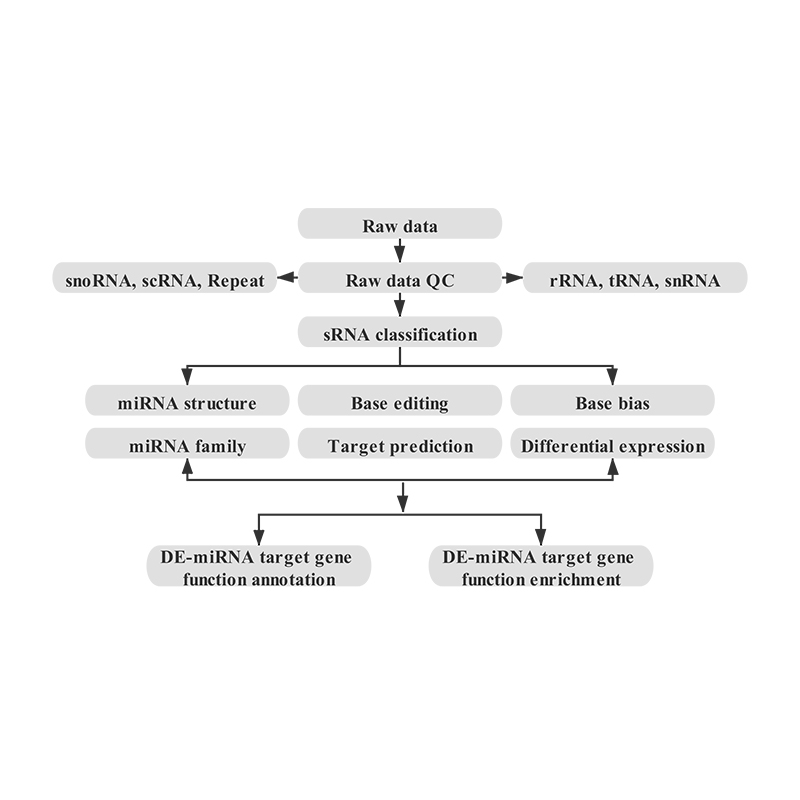
Bioinformática
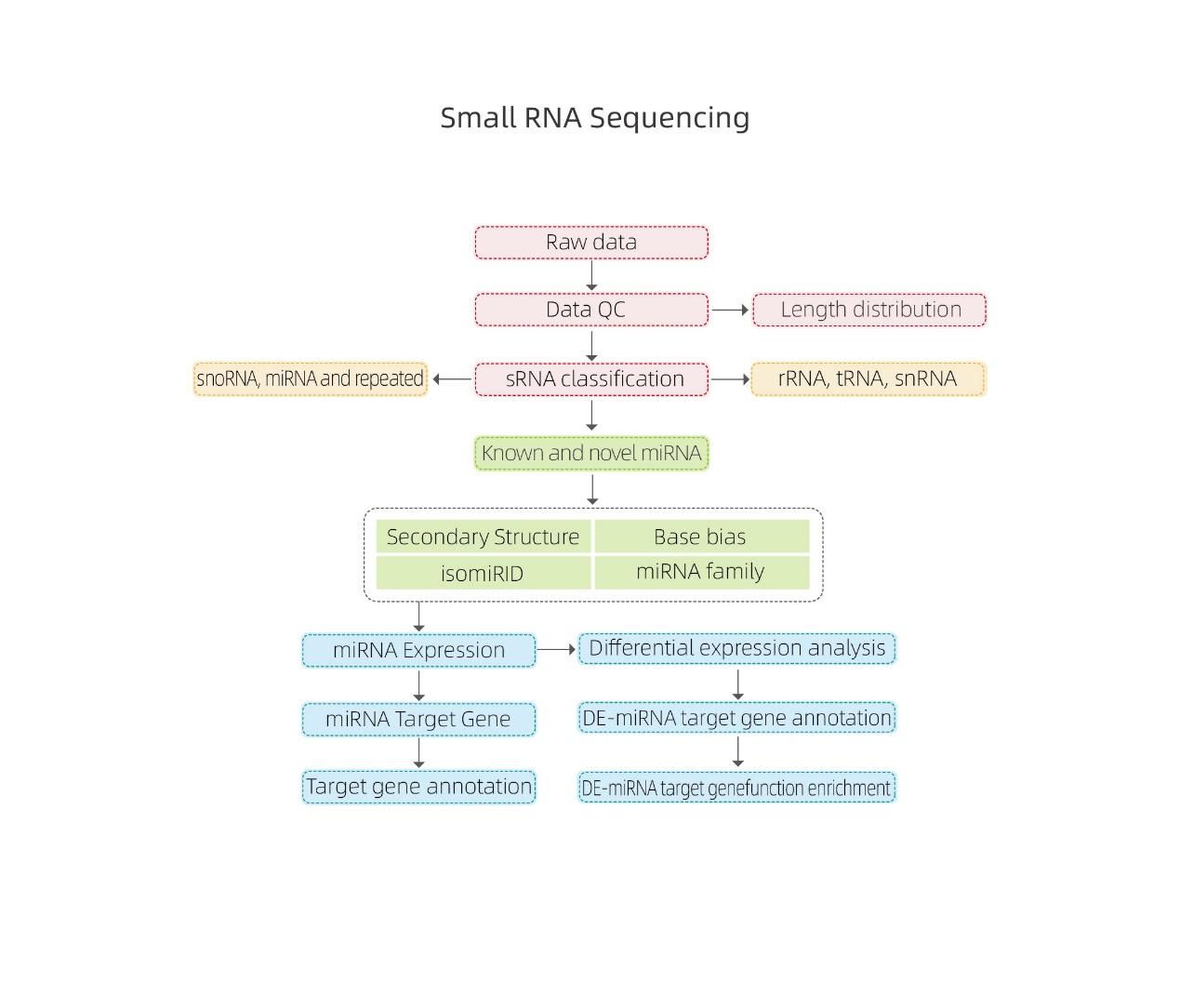 ● Control de calidad de datos sin procesar
● Control de calidad de datos sin procesar
● Clasificación de SRNA
● Alineación con un genoma de referencia
● Identificación de miRNA conocido y nuevo
● Análisis de expresión de miARN diferencial
● Anotación funcional de objetivos de miRNA
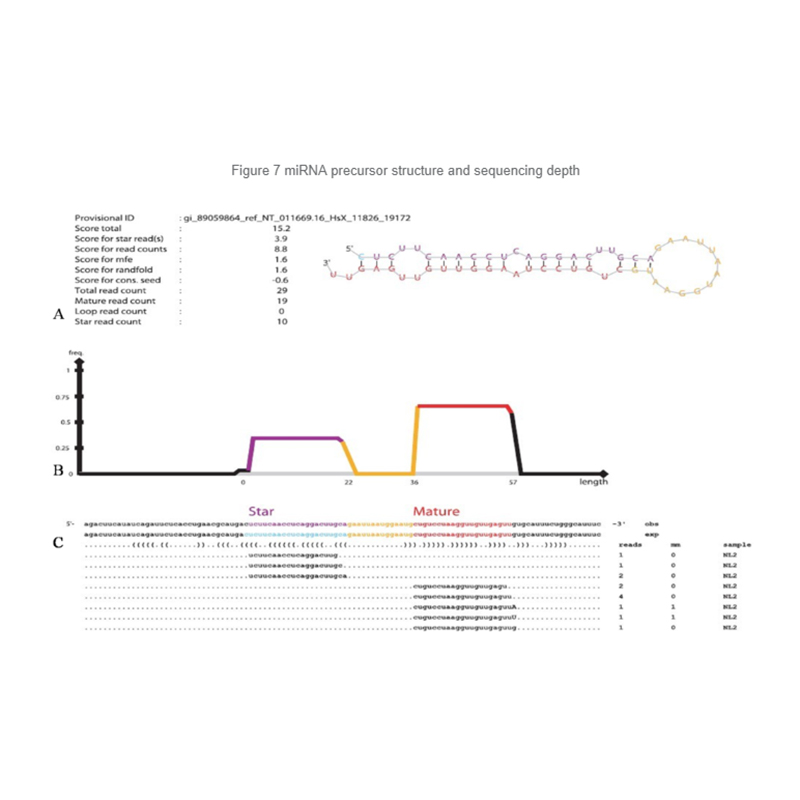
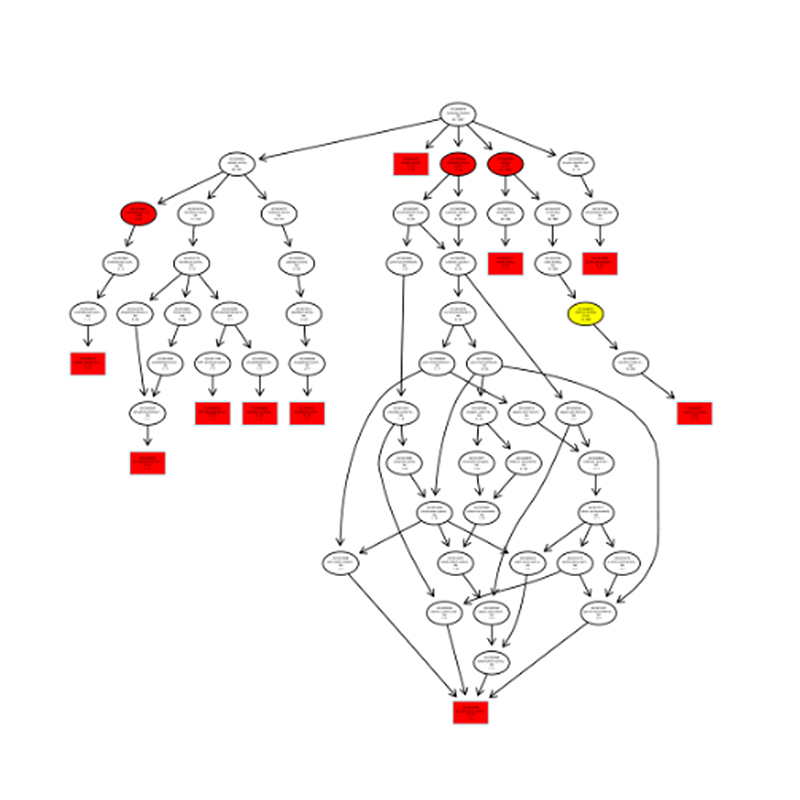
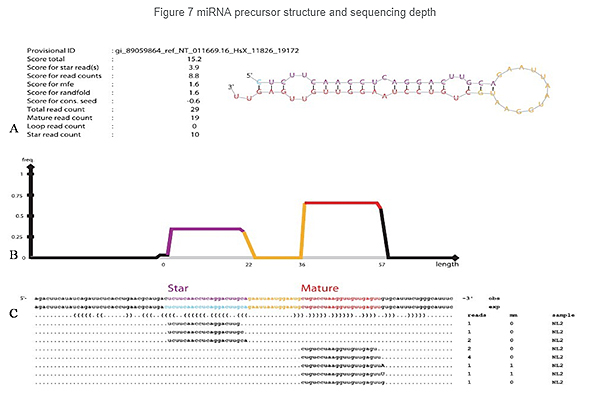
Identificación de miRNA: estructura y profundidad
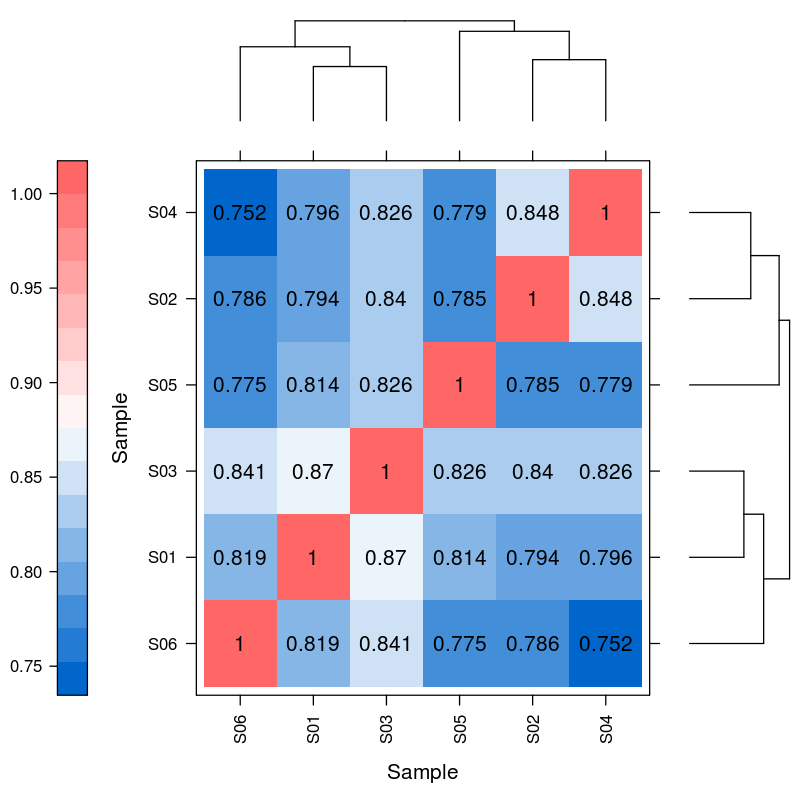
Expresión diferencial de miRNA - Hiacearchical Clustering
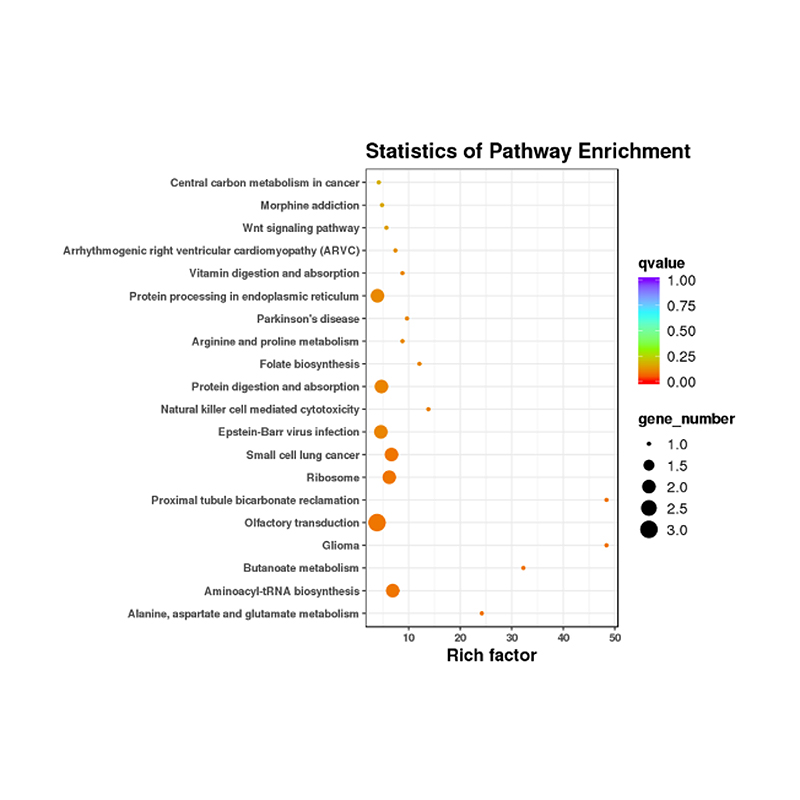
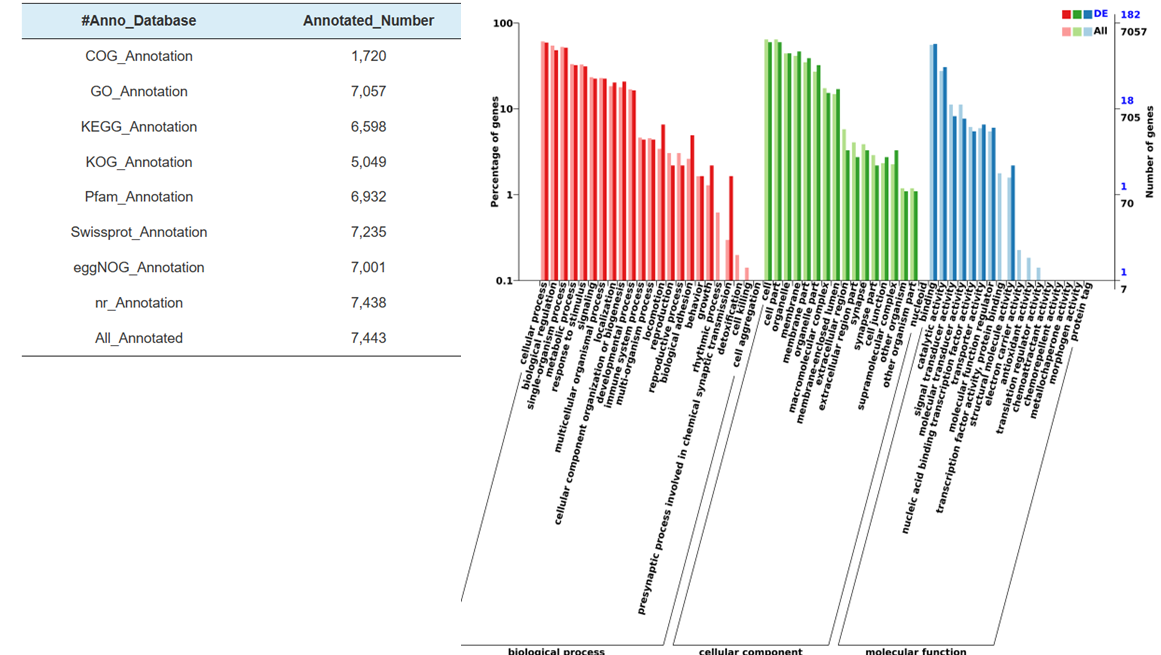
Anotación funcional del objetivo de miRNAs expresados diferencialmente
Explore los avances de investigación facilitados por los servicios de secuenciación SRNA de BMKGENE a través de una colección curada de publicaciones.
Chen, H. et al. (2023) 'Las infecciones virales inhiben la biosíntesis de saponina y la fotosíntesis en Panax Notoginseng', Fisiología y Bioquímica de las plantas, 203, p. 108038. Doi: 10.1016/j.plaphy.2023.108038.
Li, H. et al. (2023) 'La proteína Free1 de la planta que contiene el dominio Free1 se asocia con componentes del microprocesador para reprimir la biogénesis de miRNA', Embo Reports, 24 (1). doi: 10.15252/abrah.202255037/supl_file/abra202255037-sup-0004-sdatafig4.tif.
Yu, J. et al. (2023) 'El microARN ame-bantam-3p controla el desarrollo de la pupal larval al atacar los múltiples dominios del factor de crecimiento epidérmico similar al factor 8 del gen 8 (MEGF8) en el Honeybee, Apis mellifera', International Journal of Molecular Sciences, 24 (6), P) . 5726. Doi: 10.3390/ijms24065726/s1.
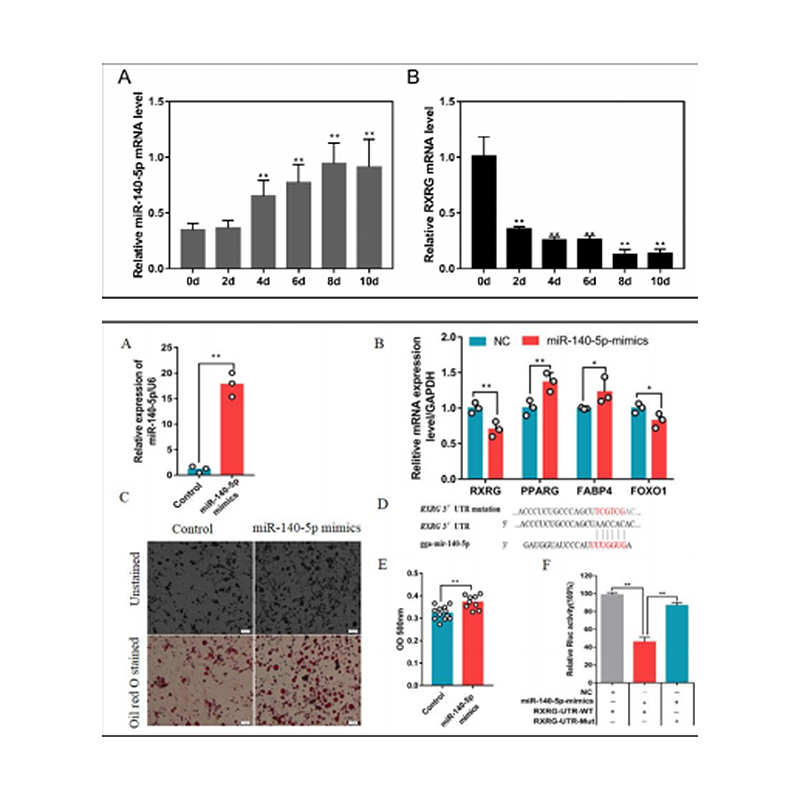
Zhang, M. et al. (2018) 'El análisis integrado de miRNA y los genes asociados con la calidad de la carne revela que GGA-MIR-140-5p afecta el depósito de grasa intramuscular en pollos', fisiología celular y bioquímica, 46 (6), pp. 2421–2433. doi: 10.1159/000489649.