Secuenciación de ARN de un solo núcleo
Esquema técnico
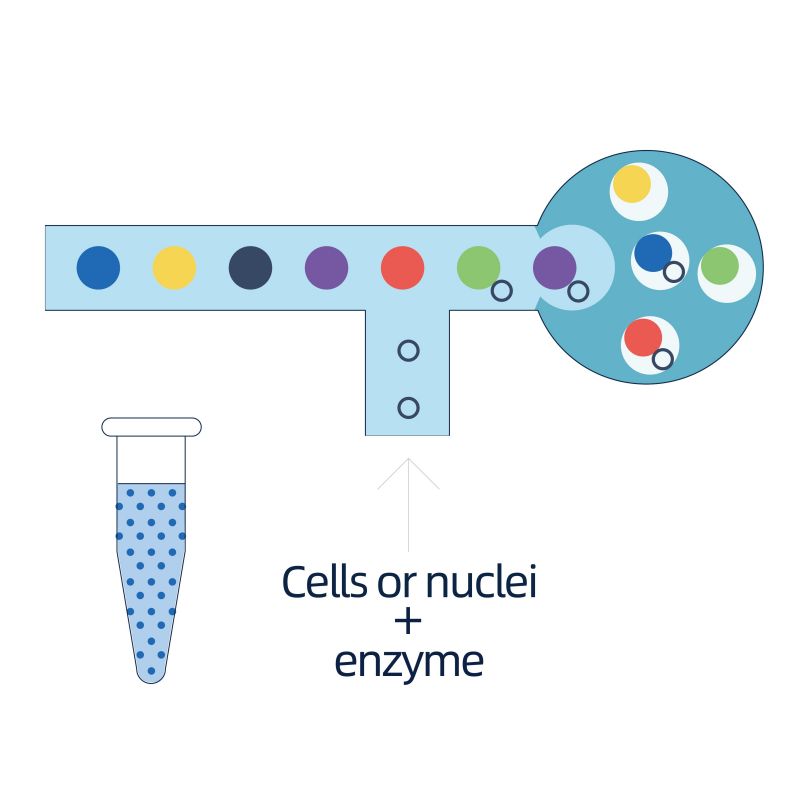
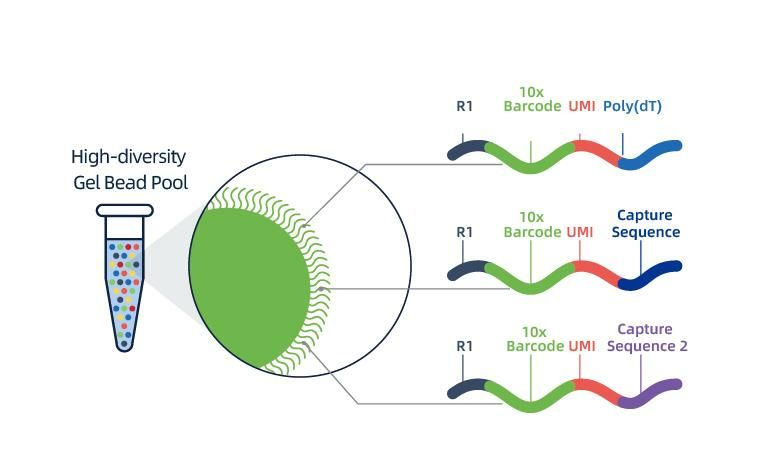
El aislamiento de los núcleos se logra por 10 × Genomics Chromium ™, que consiste en un sistema de microfluídica de ocho canales con cruces dobles. En este sistema, las cuentas de gel con códigos de barras e imprimación, las enzimas y un solo núcleo están encapsulados en caída de aceite del tamaño de un nanolitro, generando gel en la emulsión (GEM). Una vez que se forman GEM, la lisis celular y la liberación de códigos de barras se realizan en cada gema. El ARNm se transcribe inversamente en moléculas de ADNc con códigos de barras 10 × y UMI, que están sujetos aún más a la construcción de la biblioteca de secuenciación estándar.

Características
● Preparación de suspensión de un solo nucleos de tejidos congelados
● Formación de gel en la emulsión (GEM) seguida de la síntesis de ADNc
● Cada cordón en una gema se carga con cebadores compuestos por 4 secciones:
cola de poli (DT) para cebado de ARNm y síntesis de ADNc,
Identificador molecular único (UMI) para corregir el sesgo de amplificación
10x código de barras
Secuencia de unión de la votación de secuenciación de lectura parcial 1
Ventajas
La secuenciación de ARN de un solo núcleo evita las limitaciones de la secuenciación de ARN de una sola célula, habilitando:
● El uso de muestras congeladas y no solo limitadas a muestras frescas
● Baja estrés de las células congeladas en comparación con el tratamiento enzimático de las células frescas, reflejadas en los datos del transcriptoma en forma de genes menos inducidos por el estrés
● No hay necesidad de eliminación previa de glóbulos rojos
● Diámetro de celda ilimitado
● Gran gama de muestras que son elegibles para el análisis, incluidos los tipos de tejido complejos y frágiles que son propensos al agrupamiento o destrucción celular durante la disociación del tejido
Muestras que no se pueden analizar mediante secuenciación de ARN de una sola célula y son elegibles para la secuenciación de ARN de un solo núcleo:
| Célula / tejido | Razón |
| Tejido congelado sin franco | Incapaz de obtener organizaciones frescas o largas |
| Célula muscular, megakaryocito, grasa ... | El diámetro de la celda es demasiado grande para ingresar al instrumento |
| Hígado… | Demasiado frágil para romper, incapaz de distinguir células individuales |
| Célula neuronal, cerebro ... | Más sensible, fácil de estrés, cambiará los resultados de la secuencia |
| Pancreas, tiroides ... | Rico en enzimas endógenas, afectando la producción de suspensión de una sola célula |
Núcleo vs un solo célula
| Soltero | Unicelular |
| Diámetro de celda ilimitado | Diámetro de la célula: 10-40 μm |
| El material puede ser tejido de tejido | El material debe ser tejido fresco |
| Baja estrés de las células congeladas | El tratamiento enzimático puede causar reacción de estrés celular |
| No es necesario eliminar los glóbulos rojos | Los glóbulos rojos deben eliminarse |
| Nuclear expresa bioinformación | Toda la célula expresa bioinformación |
Presupuesto
| Requisitos de muestra | Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| Tejido animal ≥ 200 mg Tejido vegetal ≥ 400 mg | 10x Biblioteca de ADNc de Genomics SN | Illumina PE150 | 100k PE lecturas por celda (100-200 GB) | 700-1200 núcleos/μl y la integridad de los núcleos observados bajo microscopio |
Para obtener más detalles sobre la orientación de preparación de muestras y el flujo de trabajo de servicio, no dude en hablar con unExperto en bmkgene
Flujo de trabajo de servicio

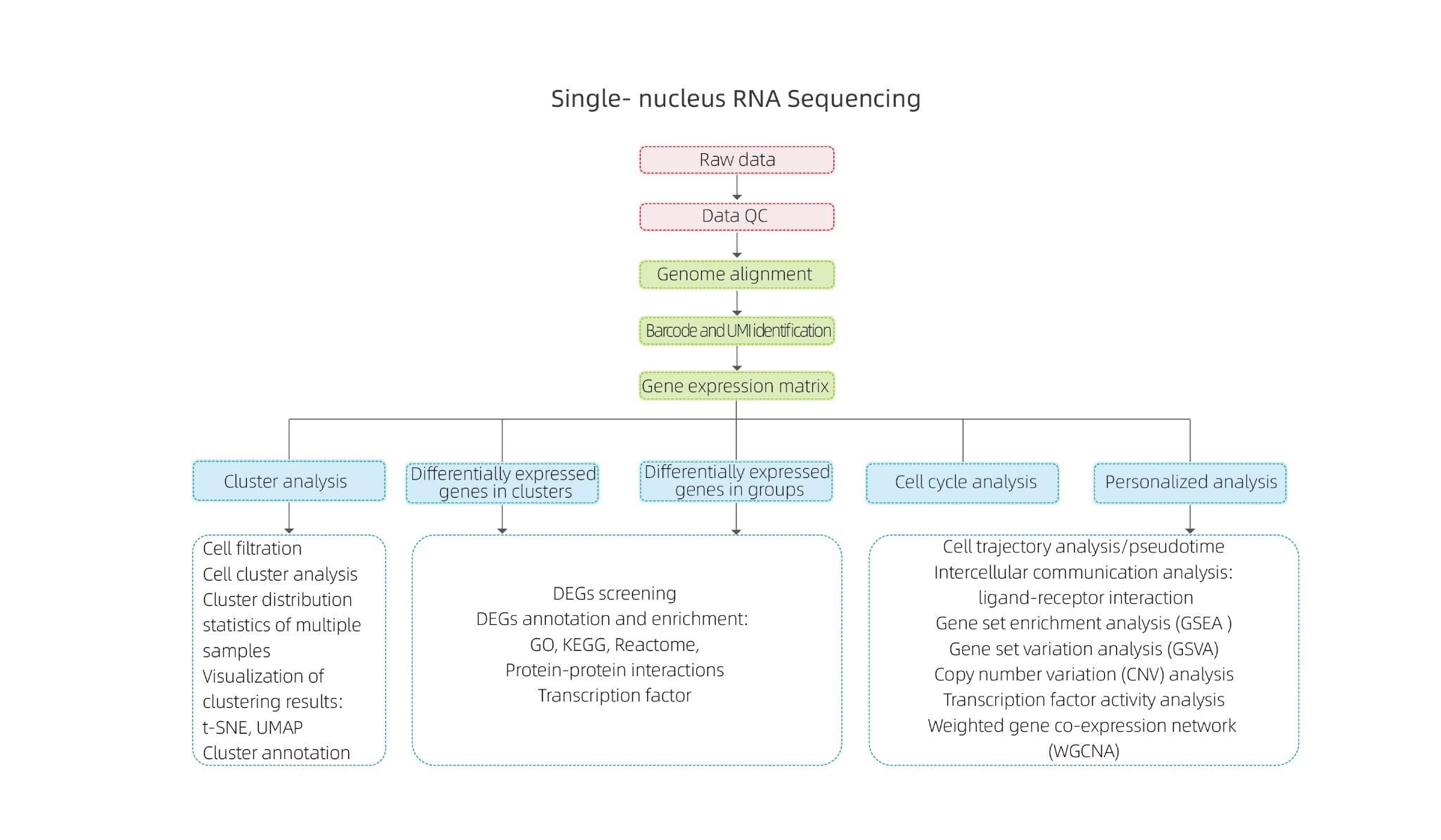
Incluye el siguiente análisis:
● Control de calidad: número de células, detección de genes, identificación precisa de células, moléculas de ARN y cuantificación de expresión
● Análisis de muestra interna:
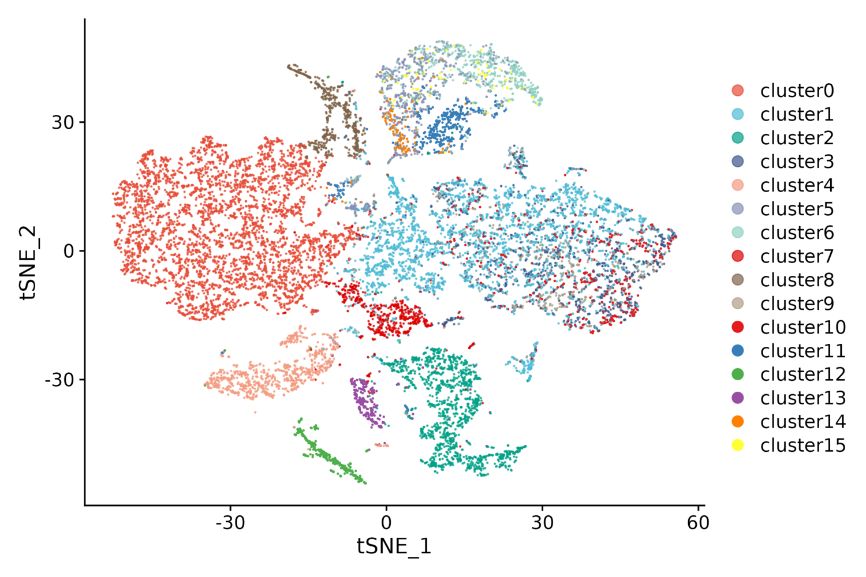
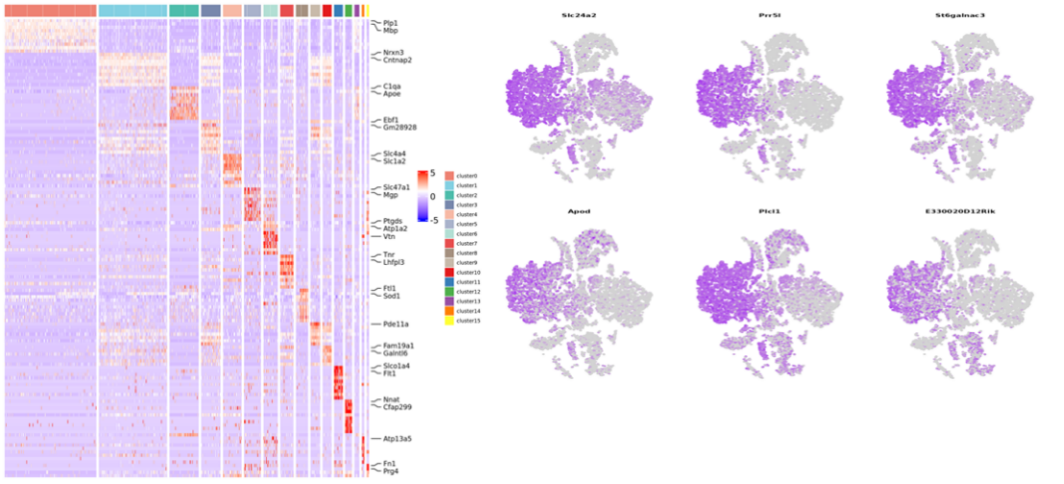
Agrupación celular y anotación de clúster
Análisis de expresión diferencial: identificación de DEG en grupos
Anotación funcional y enriquecimiento de los degs de clúster
● Análisis entre grupos:
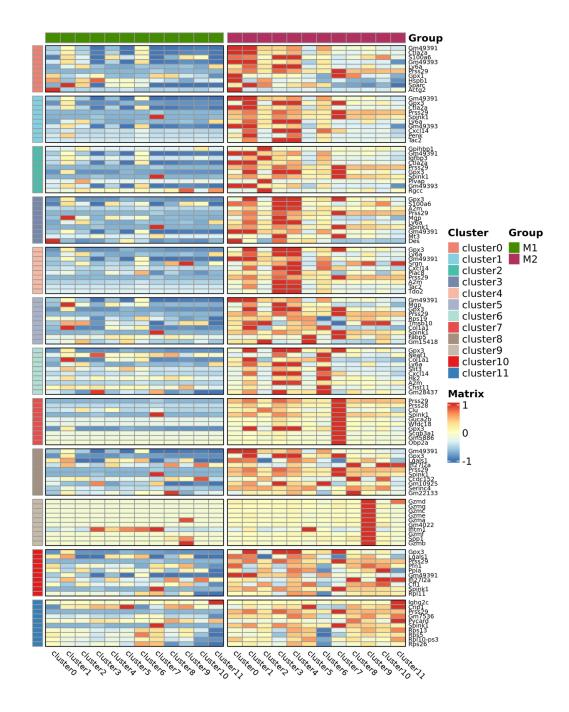
Combinación de datos
Análisis de expresión diferencial: identificación de DEG en grupos
Anotación funcional y enriquecimiento de los degs grupales
● Análisis avanzado:
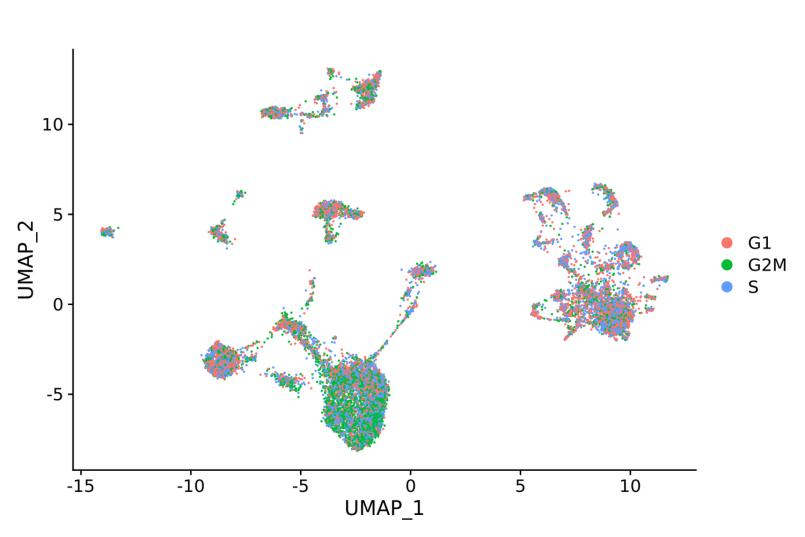
Análisis del ciclo celular
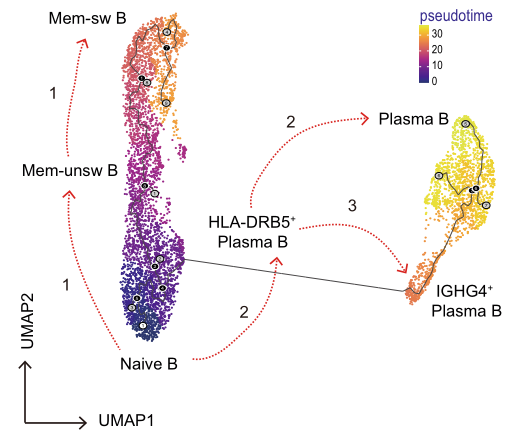
Análisis de pseudotima
Análisis de comunicación celular (CellPhonedB)
Análisis de enriquecimiento del conjunto de genes (GSEA)
Análisis de muestra interna
Agrupación celular:
Análisis de expresión diferencial: DEGS de clúster
Análisis entre grupos
Análisis de expresión diferencial: DEGS grupales
Análisis avanzado:
Análisis de pseudotima:
Análisis del ciclo celular:
Explore los avances facilitados por los servicios de secuenciación de ARN de un solo núcleo de BMKGene por 10x cromo en estas publicaciones destacadas:
Wang, L. et al. (2021) 'El análisis transcriptómico de un solo célula revela el paisaje inmune del pulmón en la exacerbación del asma resistente a los esteroides',Actas de la Academia Nacional de Ciencias de los Estados Unidos de América, 118 (2), p. E2005590118. doi: 10.1073/pnas.2005590118
Zheng, H. et al. (2022) 'Una red reguladora global para la expresión génica desregulada y la señalización metabólica anormal en las células inmunes en el microambiente de la enfermedad de Graves y la tiroiditis de Hashimoto',Fronteras en inmunología, 13, p. 879824. Doi: 10.3389/fimmu.2022.879824/bibtex.
Tian, H. et al. (2023) 'El transcriptoma único de las células descubre la heterogeneidad y las respuestas inmunes de los leucocitos después de la vacunación con Edwardsiella Tarda inactivada en Fleunder (Paralichthys olivaceus)',Acuicultura, 566, p. 739238. Doi: 10.1016/j.aquaculture.2023.739238.
Yu, Y. et al. (2023) 'La terapia fotodinámica mejora el resultado de los inhibidores del punto de control inmune mediante la remodelación de la inmunidad antitumor en pacientes con cáncer gástrico',Cáncer gástrico, 26 (5), pp. 798–813. doi: 10.1007/s10120-023-01409-x/métricas.