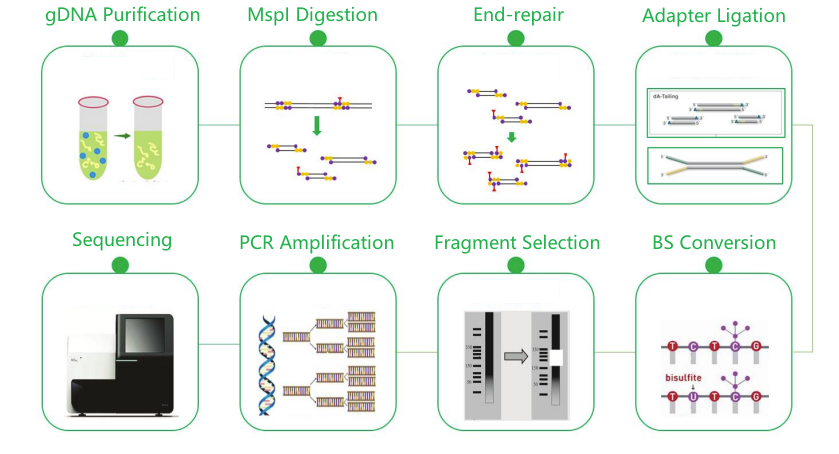
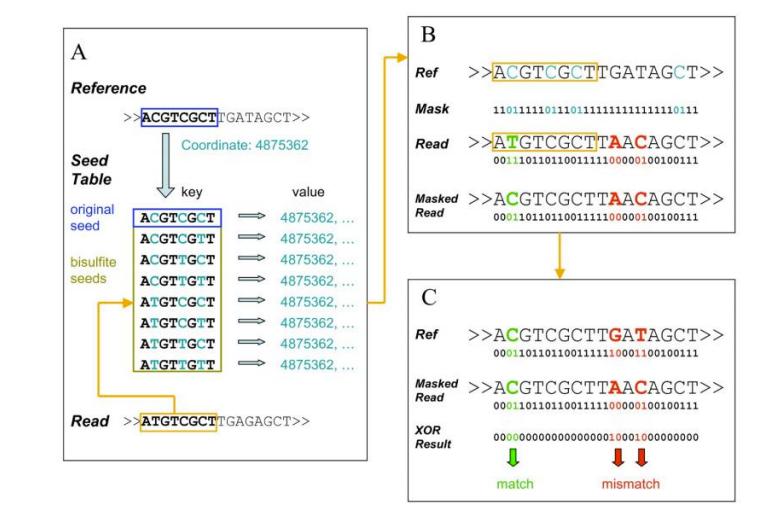
Secuenciación de bisulfito de representación reducida (RRBS)
Características del servicio
● Requiere un genoma de referencia.
● El ADN Lambda se utiliza para controlar la eficiencia de la conversión de bisulfito.
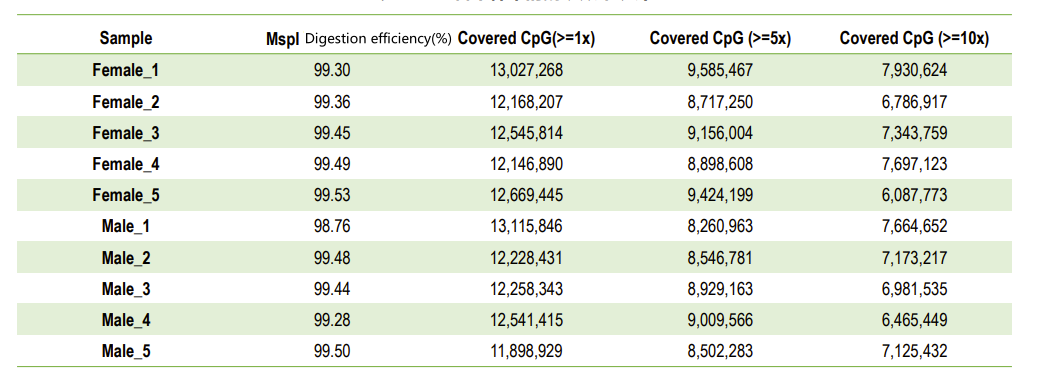
● También se controla la eficiencia de la digestión de MspI.
● Digestión doble enzimática para muestras de plantas.
● Secuenciación en Illumina NovaSeq.
Ventajas del servicio
●Alternativa rentable y eficiente a WGBS: permitiendo que el análisis se realice a un menor coste y con menores requisitos de muestra.
●Plataforma completa:Brindamos un excelente servicio integral desde el procesamiento de muestras, la construcción de bibliotecas y la secuenciación hasta el análisis bioinformático.
●Amplia experiencia: con proyectos de secuenciación RRBS completados con éxito en una amplia gama de especies, BMKGENE aporta más de una década de experiencia, un equipo de análisis altamente capacitado, contenido integral y un excelente soporte posventa.
Especificaciones de servicio
| Biblioteca | Estrategia de secuenciación | Salida de datos recomendada | Control de calidad |
| Biblioteca digerida con MspI y tratada con bisulfito | Iluminación PE150 | 8GB | Q30 ≥ 85% Conversión de bisulfito > 99% Eficiencia de corte MspI > 95% |
Requisitos de muestra
| Concentración (ng/μL) | Cantidad total (μg) |
| |
| ADN genómico | ≥ 30 | ≥ 1 | Degradación o contaminación limitada |
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de datos
Servicios postventa
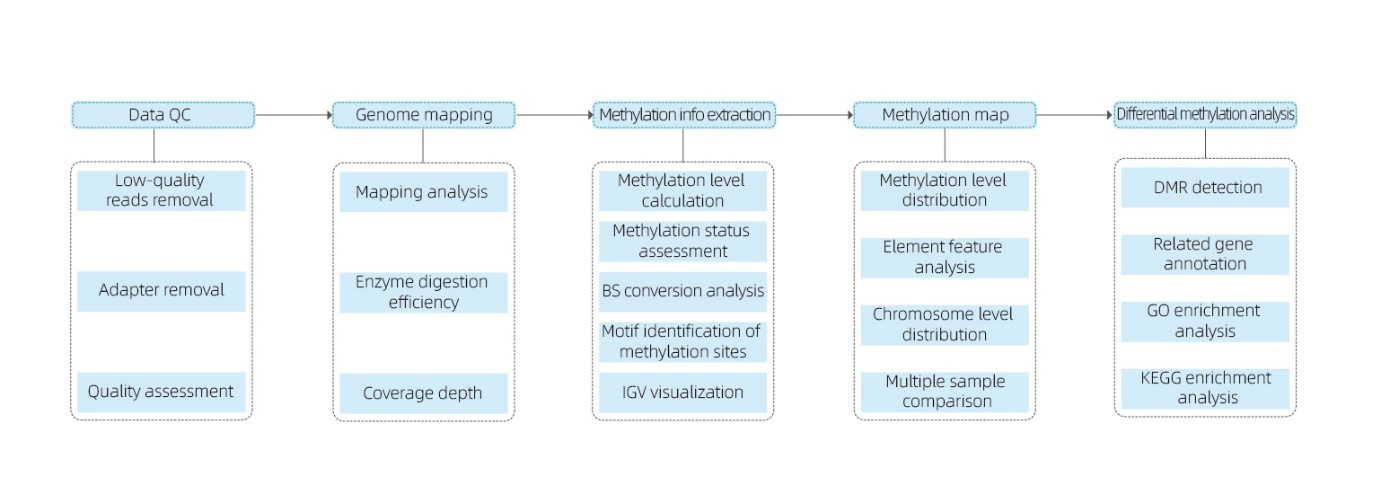
Incluye el siguiente análisis:
● Control de calidad de secuenciación sin procesar;
● Mapeo del genoma de referencia;
● Detección de bases metiladas de 5 mC e identificación de motivos;
● Análisis de la distribución de metilación y comparación de muestras;
● Análisis de regiones metiladas diferencialmente (DMR);
● Anotación funcional de genes asociados a DMR.
Control de calidad: eficiencia de la digestión (en mapeo del genoma)
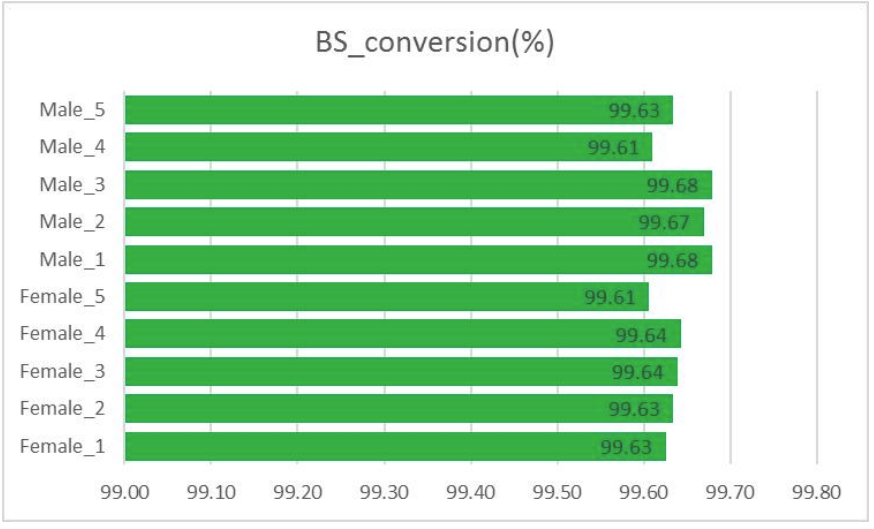
Control de calidad: conversión de bisulfito (en extracción de información de metilación)
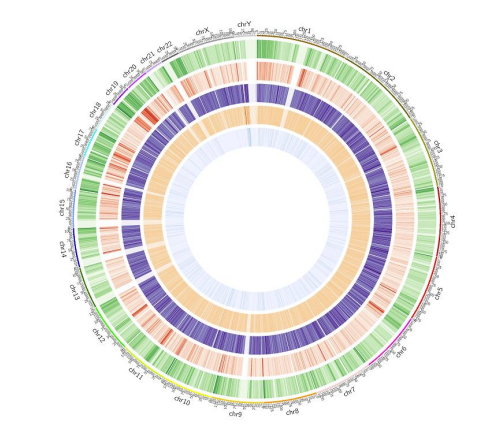
Mapa de metilación: distribución de todo el genoma de metilación de 5 mC
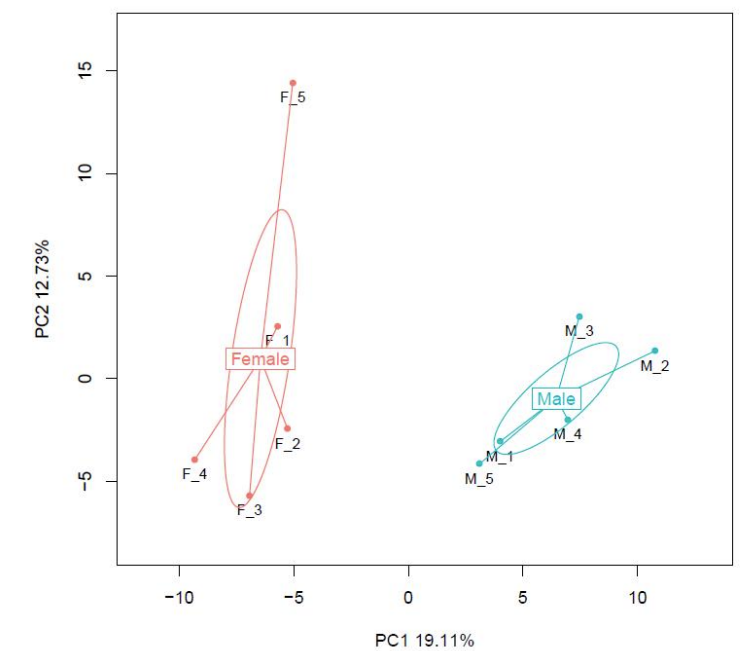
Comparación de muestras: análisis de componentes principales
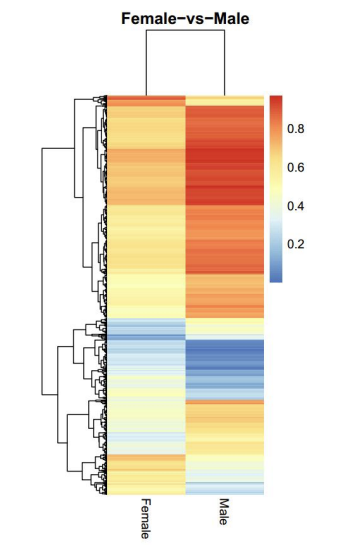
Análisis de regiones metiladas diferencialmente (DMR): mapa de calor
Explore los avances en la investigación facilitados por los servicios de secuenciación de bisulfito del genoma completo de BMKGene a través de una colección seleccionada de publicaciones.
Li, Z. y col. (2022) 'Reprogramación de alta fidelidad en células similares a Leydig mediante activación CRISPR y factores paracrinos',Nexo PNAS, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. y col. (2023) 'Análisis de metilación del ADN de todo el genoma de la composición corporal en gemelos monocigóticos chinos',Revista europea de investigación clínica, 53(11), pág. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) 'Metilación del ADN y relación cintura-cadera: un estudio de asociación de todo el epigenoma en gemelos monocigóticos chinos',Revista de investigación endocrinológica, 45(12), págs. 2365-2376. doi: 10.1007/S40618-022-01878-4.