Secuenciación de ARN procariótico
Características
● El procesamiento de muestras de ARN implicó el agotamiento del ARNr seguido de la preparación de la biblioteca de ARN direccional.
● Análisis bioinformático basado en el alineamiento con un genoma de referencia.
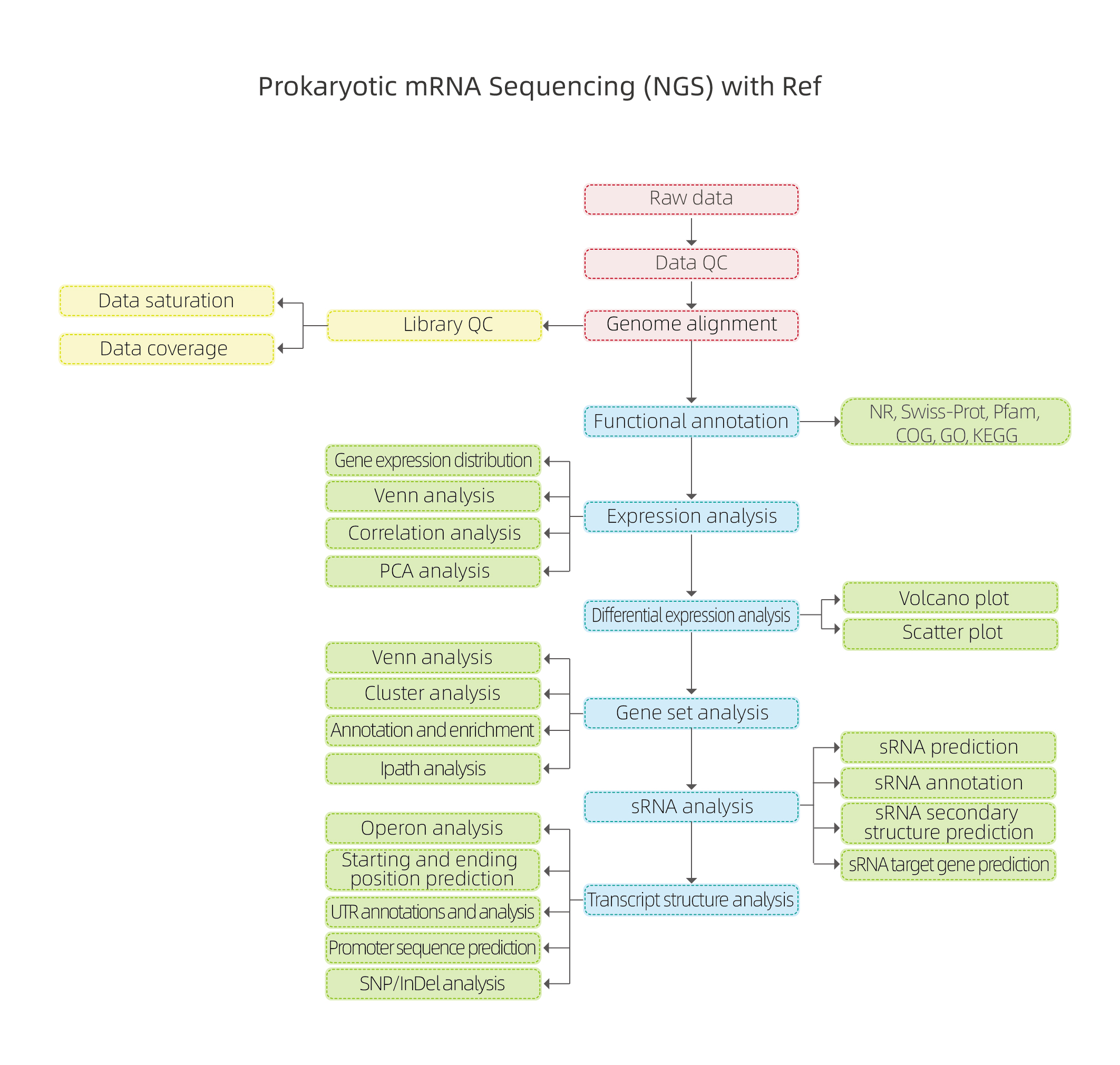
● El análisis incluye expresión genética y DEG, pero también estructura de transcripción y análisis de ARNs.
Ventajas del servicio
●Control de calidad riguroso: implementamos puntos de control centrales en todas las etapas, desde la preparación de muestras y bibliotecas hasta la secuenciación y la bioinformática. Este seguimiento meticuloso garantiza la entrega de resultados consistentes de alta calidad.
●Datos de secuenciación específicos de hebras: debido a que la preparación de la biblioteca de ARN es direccional, lo que permite la identificación de transcripciones antisentido.
●Análisis completo adaptado a transcriptomas procarióticos: el proceso bioinformático incluye no solo el análisis de la expresión génica sino también el análisis de la estructura de la transcripción, incluida la identificación de operones, UTR y promotores. También incluye análisis de sRNA, es decir, anotación y predicción de estructuras secundarias y objetivos.
●Soporte postventa: nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Requisitos de muestra y entrega
| Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| biblioteca direccional agotada de ARNr | Iluminación PE150 | 1-2 GB | Q30≥85% |
Requisitos de muestra:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 50 | ≥ 1 | DO260/280=1,8-2,0 DO260/230=1,0-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | RIN≥6,5 |
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3; B1, B2, B3.
Envío:
1. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
2. Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
Flujo de trabajo de análisis bioinformático
Incluye el siguiente análisis:
● Control de calidad de los datos sin procesar
● Alineación con el genoma de referencia.
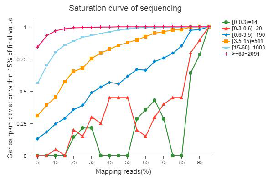
● Evaluación de la calidad de la biblioteca: aleatoriedad de la fragmentación del ARN, tamaño de inserción y saturación de secuenciación
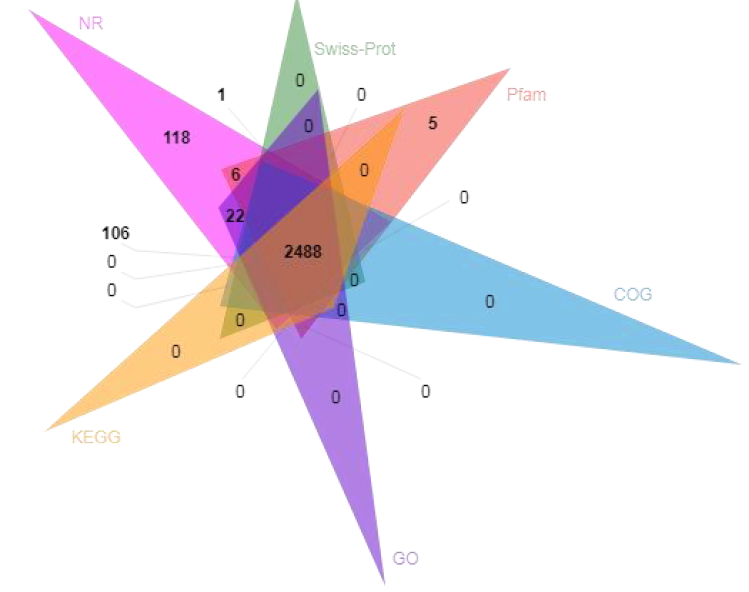
● Anotación funcional de genes codificantes previstos.
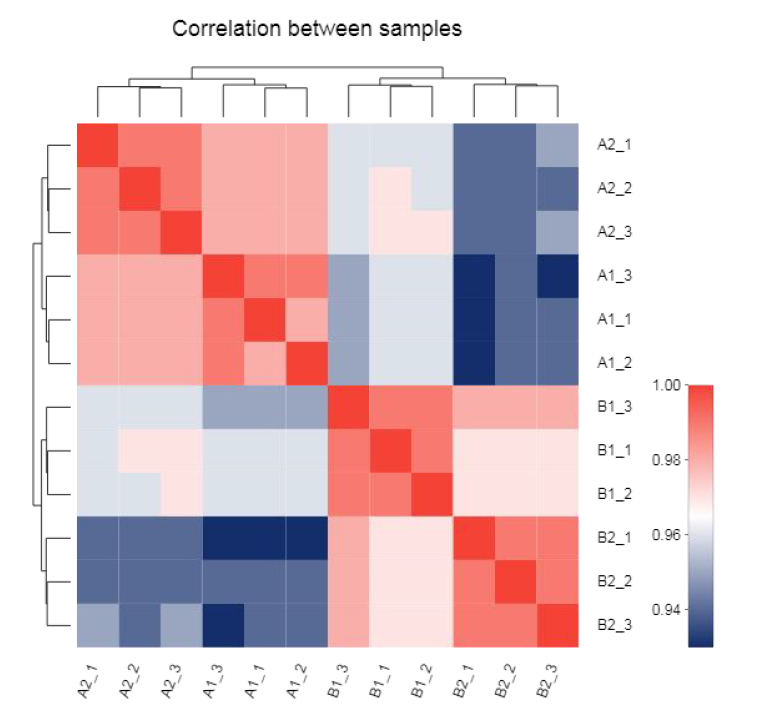
● Análisis de expresión: correlación y análisis de componentes principales (PCA)
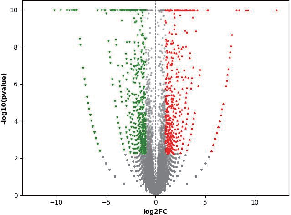
● Expresión genética diferencial (DEG)
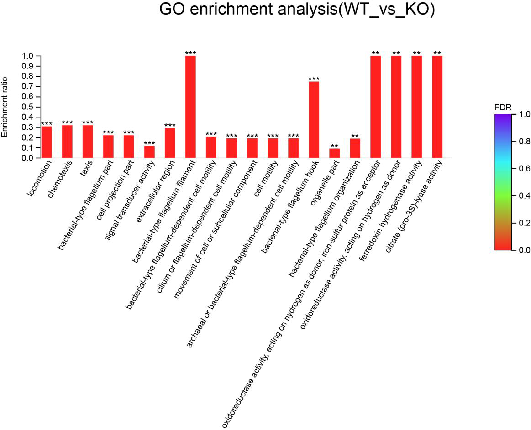
● Anotación funcional y enriquecimiento de DEG
● Análisis de sRNA: predicción, anotación, objetivo y predicción de estructura secundaria.
● Análisis de la estructura de la transcripción: operones, posiciones inicial y final, región no traducida (UTS), promotor y análisis SNP/InDel.
Saturación de secuenciación
Anotación funcional de genes codificantes.
Correlación entre muestras
Análisis de genes expresados diferencialmente (DEG)
Análisis de enriquecimiento funcional.
anotación de ARNs
Explore los avances facilitados por los servicios de secuenciación de ARNm completo Nanopore de BMKGene en esta publicación destacada.
Guan, CP y cols. (2018) 'Cambios globales en el transcriptoma de Staphylococcus epidermidis formador de biopelículas en respuesta a los alcaloides totales de Sophorea alopecuroides',Revista polaca de microbiología, 67(2), pág. 223.doi: 10.21307/PJM-2018-024.