-

Secuenciación Metagenómica -NGS
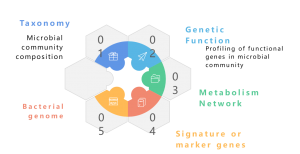
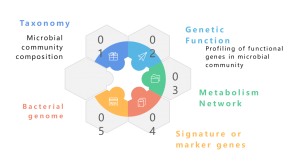
Un metagenoma es una colección del material genético total de una comunidad mixta de organismos, como los metagenomas ambientales y humanos. Contiene genomas de microorganismos tanto cultivables como no cultivables. La secuenciación metagenómica de escopeta con NGS permite el estudio de estos intrincados paisajes genómicos incrustados en muestras ambientales al proporcionar más que perfiles taxonómicos, brindando también información granular sobre la diversidad de especies, la dinámica de abundancia y las estructuras poblacionales complejas. Más allá de los estudios taxonómicos, la metagenómica de escopeta también ofrece una perspectiva de genómica funcional, que permite la exploración de genes codificados y sus supuestas funciones en los procesos ecológicos. Finalmente, el establecimiento de redes de correlación entre elementos genéticos y factores ambientales contribuye a una comprensión holística de la intrincada interacción entre las comunidades microbianas y su entorno ecológico. En conclusión, la secuenciación metagenómica es un instrumento fundamental para desentrañar las complejidades genómicas de diversas comunidades microbianas, iluminando las relaciones multifacéticas entre la genética y la ecología dentro de estos complejos ecosistemas.
Plataformas: Illumina NovaSeq y DNBSEQ-T7
-

Secuenciación metagenómica-TGS
Un metagenoma es una colección del material genético de una comunidad mixta de organismos, como los metagenomas ambientales y humanos. Contiene genomas de microorganismos tanto cultivables como no cultivables. La secuenciación metagenómica permite el estudio de estos intrincados paisajes genómicos incrustados en muestras ecológicas al proporcionar más que perfiles taxonómicos. También ofrece una perspectiva de genómica funcional mediante la exploración de los genes codificados y sus supuestas funciones en los procesos ambientales. Si bien los enfoques tradicionales con secuenciación de Illumina se han utilizado ampliamente en estudios metagenómicos, la llegada de la secuenciación de lectura larga Nanopore y PacBio ha cambiado el campo. Las tecnologías Nanopore y PacBio mejoran los análisis bioinformáticos posteriores, en particular el ensamblaje del metagenoma, asegurando ensamblajes más continuos. Los informes indican que la metagenómica basada en nanoporos y PacBio ha generado con éxito genomas bacterianos completos y cerrados a partir de microbiomas complejos (Moss, EL, et al., Nature Biotech, 2020). La integración de lecturas de Nanopore con lecturas de Illumina proporciona un enfoque estratégico para la corrección de errores, mitigando la baja precisión inherente de Nanopore. Esta combinación sinérgica aprovecha las fortalezas de cada plataforma de secuenciación, ofreciendo una solución sólida para superar limitaciones potenciales y mejorar la precisión y confiabilidad de los análisis metagenómicos.
Plataforma: Nanopore PromethION 48, Illumia y PacBio Revio
-

Secuenciación de bisulfito del genoma completo (WGBS)
La secuenciación con bisulfito del genoma completo (WGBS) es la metodología estándar de oro para la exploración en profundidad de la metilación del ADN, específicamente la quinta posición en la citosina (5-mC), un regulador fundamental de la expresión genética y la actividad celular. El principio subyacente del WGBS implica el tratamiento con bisulfito, que induce la conversión de citosinas no metiladas en uracilo (C a U), dejando las citosinas metiladas sin cambios. Esta técnica ofrece resolución de base única, lo que permite a los investigadores investigar exhaustivamente el metiloma y descubrir patrones de metilación anormales asociados con diversas afecciones, en particular el cáncer. Al emplear WGBS, los científicos pueden obtener conocimientos incomparables sobre los paisajes de metilación de todo el genoma, proporcionando una comprensión matizada de los mecanismos epigenéticos que subyacen a diversos procesos biológicos y enfermedades.
-

Ensayo de cromatina accesible a transposasa con secuenciación de alto rendimiento (ATAC-seq)
ATAC-seq es una técnica de secuenciación de alto rendimiento utilizada para el análisis de accesibilidad de la cromatina en todo el genoma. Su uso proporciona una comprensión más profunda de los complejos mecanismos del control epigenético global sobre la expresión genética. El método utiliza una transposasa Tn5 hiperactiva para fragmentar y etiquetar simultáneamente regiones abiertas de cromatina mediante la inserción de adaptadores de secuenciación. La amplificación por PCR posterior da como resultado la creación de una biblioteca de secuenciación que permite la identificación integral de regiones de cromatina abiertas en condiciones espacio-temporales específicas. ATAC-seq proporciona una visión holística de los paisajes de cromatina accesibles, a diferencia de los métodos que se centran únicamente en sitios de unión de factores de transcripción o regiones específicas modificadas por histonas. Al secuenciar estas regiones de cromatina abiertas, ATAC-seq revela regiones con mayor probabilidad de activar secuencias reguladoras y posibles sitios de unión de factores de transcripción, lo que ofrece información valiosa sobre la modulación dinámica de la expresión génica en todo el genoma.
-

Secuenciación de amplicones 16S/18S/ITS-PacBio
Los genes de ARNr 16S y 18S, junto con la región del espaciador interno transcrito (ITS), sirven como marcadores fundamentales de huellas dactilares moleculares debido a su combinación de regiones altamente conservadas e hipervariables, lo que los convierte en herramientas invaluables para caracterizar organismos procarióticos y eucariotas. La amplificación y secuenciación de estas regiones ofrecen un enfoque sin aislamiento para investigar la composición y diversidad microbiana en varios ecosistemas. Si bien la secuenciación de Illumina generalmente se dirige a regiones hipervariables cortas como V3-V4 de 16S e ITS1, se ha demostrado que se puede lograr una anotación taxonómica superior secuenciando la longitud completa de 16S, 18S e ITS. Este enfoque integral da como resultado porcentajes más altos de secuencias clasificadas con precisión, logrando un nivel de resolución que se extiende a la identificación de especies. La plataforma de secuenciación de una sola molécula en tiempo real (SMRT) de PacBio se destaca por proporcionar lecturas largas (HiFi) altamente precisas que cubren los amplicones de longitud completa, rivalizando con la precisión de la secuenciación de Illumina. Esta capacidad permite a los investigadores obtener una ventaja inigualable: una vista panorámica del panorama genético. La cobertura ampliada eleva significativamente la resolución en la anotación de especies, particularmente dentro de comunidades bacterianas o fúngicas, lo que permite una comprensión más profunda de las complejidades de las poblaciones microbianas.
-

Secuenciación de amplicones 16S/18S/ITS-NGS
La secuenciación de amplicones con tecnología Illumina, dirigida específicamente a los marcadores genéticos 16S, 18S e ITS, es un método poderoso para desentrañar la filogenia, la taxonomía y la abundancia de especies dentro de las comunidades microbianas. Este enfoque implica secuenciar las regiones hipervariables de los marcadores genéticos internos. Introducido originalmente como una huella digital molecular porWoeses et al.En 1977, esta técnica revolucionó la elaboración de perfiles de microbiomas al permitir análisis sin aislamiento. Mediante la secuenciación de 16S (bacterias), 18S (hongos) y el espaciador interno transcrito (ITS, hongos), los investigadores pueden identificar no sólo especies abundantes sino también raras y no identificadas. Ampliamente adoptada como una herramienta fundamental, la secuenciación de amplicones se ha vuelto fundamental para discernir composiciones microbianas diferenciales en diversos entornos, incluida la boca humana, los intestinos, las heces y más allá.
-

Resecuenciación del genoma completo de bacterias y hongos
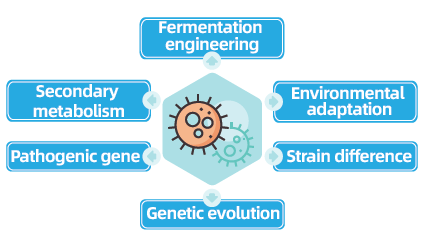
Los proyectos de resecuenciación del genoma completo de bacterias y hongos son fundamentales para avanzar en la genómica microbiana al permitir completar y comparar genomas microbianos. Esto facilita la ingeniería de fermentación, la optimización de procesos industriales y la exploración de vías de metabolismo secundario. Además, la resecuenciación de hongos y bacterias es crucial para comprender la adaptación ambiental, optimizar las cepas y revelar la dinámica de la evolución genética, con amplias implicaciones en la medicina, la agricultura y las ciencias ambientales.
-

PacBio-Secuenciación de amplicones 16S/18S/ITS de longitud completa
La plataforma Amplicon (16S/18S/ITS) se desarrolla con años de experiencia en análisis de proyectos de diversidad microbiana, que contiene análisis básicos estandarizados y análisis personalizados: el análisis básico cubre el contenido de análisis principal de la investigación microbiana actual, el contenido del análisis es rico y completo. y los resultados del análisis se presentan en forma de informes de proyecto; El contenido del análisis personalizado es diverso. Se pueden seleccionar muestras y establecer parámetros de manera flexible de acuerdo con el informe de análisis básico y el propósito de la investigación, para cumplir con los requisitos personalizados. Sistema operativo Windows, sencillo y rápido.
-

Transcriptoma completo de PacBio (sin referencia)
Tomando como entrada los datos de secuenciación de isoformas de Pacific Biosciences (PacBio), esta aplicación puede identificar secuencias de transcripción completas (sin ensamblaje). Al mapear secuencias de longitud completa con respecto al genoma de referencia, las transcripciones se pueden optimizar mediante genes, transcripciones, regiones codificantes, etc. conocidos. En este caso, se puede lograr una identificación más precisa de las estructuras del ARNm, como el empalme alternativo, etc. El análisis conjunto con datos de secuenciación del transcriptoma NGS permite una anotación más completa y una cuantificación más precisa en la expresión a nivel de transcripción, lo que beneficia en gran medida la expresión diferencial y el análisis funcional posteriores.
-

Secuenciación de bisulfito de representación reducida (RRBS)
La secuenciación con bisulfito de representación reducida (RRBS) ha surgido como una alternativa rentable y eficiente a la secuenciación con bisulfito del genoma completo (WGBS) en la investigación de la metilación del ADN. Si bien WGBS proporciona información integral al examinar todo el genoma con una resolución de base única, su alto costo puede ser un factor limitante. RRBS mitiga estratégicamente este desafío al analizar selectivamente una porción representativa del genoma. Esta metodología se basa en el enriquecimiento de regiones ricas en islas CpG mediante escisión de MspI seguida de una selección de tamaño de fragmentos de 200-500/600 pb. En consecuencia, solo se secuencian las regiones próximas a las islas CpG, mientras que aquellas con islas CpG distantes se excluyen del análisis. Este proceso, combinado con la secuenciación con bisulfito, permite la detección de alta resolución de la metilación del ADN, y el enfoque de secuenciación, PE150, se centra específicamente en los extremos de los insertos en lugar de en el medio, lo que aumenta la eficiencia del perfil de metilación. El RRBS es una herramienta invaluable que permite una investigación rentable sobre la metilación del ADN y avanza en el conocimiento de los mecanismos epigenéticos.
-

Secuenciación de ARN procariótico
La secuenciación de ARN permite la elaboración de perfiles completos de todas las transcripciones de ARN dentro de las células en condiciones específicas. Esta tecnología de vanguardia sirve como una herramienta potente, que revela intrincados perfiles de expresión genética, estructuras genéticas y mecanismos moleculares asociados con diversos procesos biológicos. Ampliamente adoptada en la investigación fundamental, el diagnóstico clínico y el desarrollo de fármacos, la secuenciación de ARN ofrece información sobre las complejidades de la dinámica celular y la regulación genética. Nuestro procesamiento de muestras de ARN procariótico está diseñado para transcriptomas procarióticos, lo que implica el agotamiento del ARNr y la preparación de bibliotecas direccionales.
Plataforma: Illumina NovaSeq
-

Secuenciación del metatranscriptoma
Aprovechando la tecnología de secuenciación de Illumina, el servicio de secuenciación de metatranscriptomas de BMKGENE revela la expresión genética dinámica de una amplia gama de microbios, que abarcan desde eucariotas hasta procariotas y virus, en entornos naturales como el suelo, el agua, el mar, las heces y el intestino. Nuestro servicio integral permite a los investigadores profundizar en los perfiles completos de expresión genética de comunidades microbianas complejas. Más allá del análisis taxonómico, nuestro servicio de secuenciación de metatranscriptomas facilita la exploración del enriquecimiento funcional, arrojando luz sobre los genes expresados diferencialmente y sus funciones. Descubra una gran cantidad de conocimientos biológicos mientras navega por los complejos paisajes de expresión genética, diversidad taxonómica y dinámica funcional dentro de estos diversos nichos ambientales.
-

Ensamblaje del genoma fúngico de novo
BMKGENE ofrece soluciones versátiles para genomas de hongos, que satisfacen diversas necesidades de investigación y la integridad genómica deseada. La utilización únicamente de la secuenciación de Illumina de lectura corta permite la generación de un borrador del genoma. Las lecturas cortas y la secuenciación de lectura larga utilizando Nanopore o Pacbio se combinan para obtener un genoma fúngico más refinado con cóntigos más largos. Además, la integración de la secuenciación Hi-C mejora aún más las capacidades, permitiendo lograr un genoma completo a nivel de cromosoma.