-

Secuenciación Metagenómica -NGS
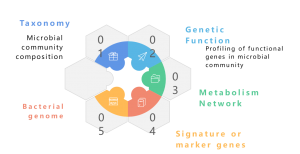
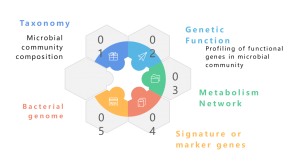
Un metagenoma es una colección del material genético total de una comunidad mixta de organismos, como los metagenomas ambientales y humanos. Contiene genomas de microorganismos tanto cultivables como no cultivables. La secuenciación metagenómica de escopeta con NGS permite el estudio de estos intrincados paisajes genómicos incrustados en muestras ambientales al proporcionar más que perfiles taxonómicos, brindando también información granular sobre la diversidad de especies, la dinámica de abundancia y las estructuras poblacionales complejas. Más allá de los estudios taxonómicos, la metagenómica de escopeta también ofrece una perspectiva de genómica funcional, que permite la exploración de genes codificados y sus supuestas funciones en los procesos ecológicos. Finalmente, el establecimiento de redes de correlación entre elementos genéticos y factores ambientales contribuye a una comprensión holística de la intrincada interacción entre las comunidades microbianas y su entorno ecológico. En conclusión, la secuenciación metagenómica es un instrumento fundamental para desentrañar las complejidades genómicas de diversas comunidades microbianas, iluminando las relaciones multifacéticas entre la genética y la ecología dentro de estos complejos ecosistemas.
Plataformas: Illumina NovaSeq y DNBSEQ-T7
-

Secuenciación metagenómica-TGS
Un metagenoma es una colección del material genético de una comunidad mixta de organismos, como los metagenomas ambientales y humanos. Contiene genomas de microorganismos tanto cultivables como no cultivables. La secuenciación metagenómica permite el estudio de estos intrincados paisajes genómicos incrustados en muestras ecológicas al proporcionar más que perfiles taxonómicos. También ofrece una perspectiva de genómica funcional mediante la exploración de los genes codificados y sus supuestas funciones en los procesos ambientales. Si bien los enfoques tradicionales con secuenciación de Illumina se han utilizado ampliamente en estudios metagenómicos, la llegada de la secuenciación de lectura larga Nanopore y PacBio ha cambiado el campo. Las tecnologías Nanopore y PacBio mejoran los análisis bioinformáticos posteriores, en particular el ensamblaje del metagenoma, asegurando ensamblajes más continuos. Los informes indican que la metagenómica basada en nanoporos y PacBio ha generado con éxito genomas bacterianos completos y cerrados a partir de microbiomas complejos (Moss, EL, et al., Nature Biotech, 2020). La integración de lecturas de Nanopore con lecturas de Illumina proporciona un enfoque estratégico para la corrección de errores, mitigando la baja precisión inherente de Nanopore. Esta combinación sinérgica aprovecha las fortalezas de cada plataforma de secuenciación, ofreciendo una solución sólida para superar limitaciones potenciales y mejorar la precisión y confiabilidad de los análisis metagenómicos.
Plataforma: Nanopore PromethION 48, Illumia y PacBio Revio
-

Secuenciación de amplicones 16S/18S/ITS-PacBio
Los genes de ARNr 16S y 18S, junto con la región del espaciador interno transcrito (ITS), sirven como marcadores fundamentales de huellas dactilares moleculares debido a su combinación de regiones altamente conservadas e hipervariables, lo que los convierte en herramientas invaluables para caracterizar organismos procarióticos y eucariotas. La amplificación y secuenciación de estas regiones ofrecen un enfoque sin aislamiento para investigar la composición y diversidad microbiana en varios ecosistemas. Si bien la secuenciación de Illumina generalmente se dirige a regiones hipervariables cortas como V3-V4 de 16S e ITS1, se ha demostrado que se puede lograr una anotación taxonómica superior secuenciando la longitud completa de 16S, 18S e ITS. Este enfoque integral da como resultado porcentajes más altos de secuencias clasificadas con precisión, logrando un nivel de resolución que se extiende a la identificación de especies. La plataforma de secuenciación de una sola molécula en tiempo real (SMRT) de PacBio se destaca por proporcionar lecturas largas (HiFi) altamente precisas que cubren los amplicones de longitud completa, rivalizando con la precisión de la secuenciación de Illumina. Esta capacidad permite a los investigadores obtener una ventaja inigualable: una vista panorámica del panorama genético. La cobertura ampliada eleva significativamente la resolución en la anotación de especies, particularmente dentro de comunidades bacterianas o fúngicas, lo que permite una comprensión más profunda de las complejidades de las poblaciones microbianas.
-

Secuenciación de amplicones 16S/18S/ITS-NGS
La secuenciación de amplicones con tecnología Illumina, dirigida específicamente a los marcadores genéticos 16S, 18S e ITS, es un método poderoso para desentrañar la filogenia, la taxonomía y la abundancia de especies dentro de las comunidades microbianas. Este enfoque implica secuenciar las regiones hipervariables de los marcadores genéticos internos. Introducido originalmente como una huella digital molecular porWoeses et al.En 1977, esta técnica revolucionó la elaboración de perfiles de microbiomas al permitir análisis sin aislamiento. Mediante la secuenciación de 16S (bacterias), 18S (hongos) y el espaciador interno transcrito (ITS, hongos), los investigadores pueden identificar no sólo especies abundantes sino también raras y no identificadas. Ampliamente adoptada como una herramienta fundamental, la secuenciación de amplicones se ha vuelto fundamental para discernir composiciones microbianas diferenciales en diversos entornos, incluida la boca humana, los intestinos, las heces y más allá.
-

Resecuenciación del genoma completo de bacterias y hongos
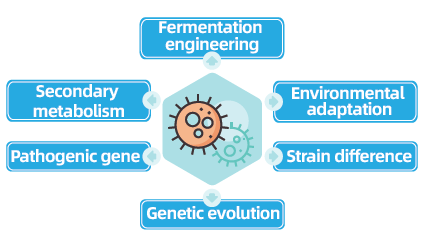
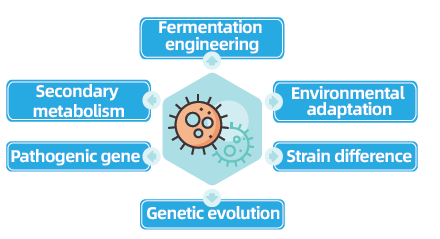
Los proyectos de resecuenciación del genoma completo de bacterias y hongos son fundamentales para avanzar en la genómica microbiana al permitir completar y comparar genomas microbianos. Esto facilita la ingeniería de fermentación, la optimización de procesos industriales y la exploración de vías de metabolismo secundario. Además, la resecuenciación de hongos y bacterias es crucial para comprender la adaptación ambiental, optimizar las cepas y revelar la dinámica de la evolución genética, con amplias implicaciones en la medicina, la agricultura y las ciencias ambientales.
-

Secuenciación de ARN procariótico
La secuenciación de ARN permite la elaboración de perfiles completos de todas las transcripciones de ARN dentro de las células en condiciones específicas. Esta tecnología de vanguardia sirve como una herramienta potente, que revela intrincados perfiles de expresión genética, estructuras genéticas y mecanismos moleculares asociados con diversos procesos biológicos. Ampliamente adoptada en la investigación fundamental, el diagnóstico clínico y el desarrollo de fármacos, la secuenciación de ARN ofrece información sobre las complejidades de la dinámica celular y la regulación genética. Nuestro procesamiento de muestras de ARN procariótico está diseñado para transcriptomas procarióticos, lo que implica el agotamiento del ARNr y la preparación de bibliotecas direccionales.
Plataforma: Illumina NovaSeq
-

Secuenciación del metatranscriptoma
Aprovechando la tecnología de secuenciación de Illumina, el servicio de secuenciación de metatranscriptomas de BMKGENE revela la expresión genética dinámica de una amplia gama de microbios, que abarcan desde eucariotas hasta procariotas y virus, en entornos naturales como el suelo, el agua, el mar, las heces y el intestino. Nuestro servicio integral permite a los investigadores profundizar en los perfiles completos de expresión genética de comunidades microbianas complejas. Más allá del análisis taxonómico, nuestro servicio de secuenciación de metatranscriptomas facilita la exploración del enriquecimiento funcional, arrojando luz sobre los genes expresados diferencialmente y sus funciones. Descubra una gran cantidad de conocimientos biológicos mientras navega por los complejos paisajes de expresión genética, diversidad taxonómica y dinámica funcional dentro de estos diversos nichos ambientales.
-

Ensamblaje del genoma fúngico de novo
BMKGENE ofrece soluciones versátiles para genomas de hongos, que satisfacen diversas necesidades de investigación y la integridad genómica deseada. La utilización únicamente de la secuenciación de Illumina de lectura corta permite la generación de un borrador del genoma. Las lecturas cortas y la secuenciación de lectura larga utilizando Nanopore o Pacbio se combinan para obtener un genoma fúngico más refinado con cóntigos más largos. Además, la integración de la secuenciación Hi-C mejora aún más las capacidades, permitiendo lograr un genoma completo a nivel de cromosoma.
-

Ensamblaje del genoma bacteriano de novo
Brindamos un servicio completo de ensamblaje de genoma bacteriano, garantizando 0 brechas. Esto es posible integrando tecnologías de secuenciación de lectura larga, como Nanopore y PacBio para el ensamblaje y secuenciación de lectura corta con Illumina para la validación del ensamblaje y la corrección de errores de las lecturas de ONT. Nuestro servicio proporciona el flujo de trabajo bioinformático completo desde el ensamblaje, la anotación funcional y el análisis bioinformático avanzado, cumpliendo objetivos de investigación específicos. Este servicio permite el desarrollo de genomas de referencia precisos para diversos estudios genéticos y genómicos. Además, constituye la base para aplicaciones como la optimización de cepas, la ingeniería genética y el desarrollo de tecnología microbiana, lo que garantiza datos genómicos confiables y sin lagunas, cruciales para avanzar en los conocimientos científicos y la innovación biotecnológica.