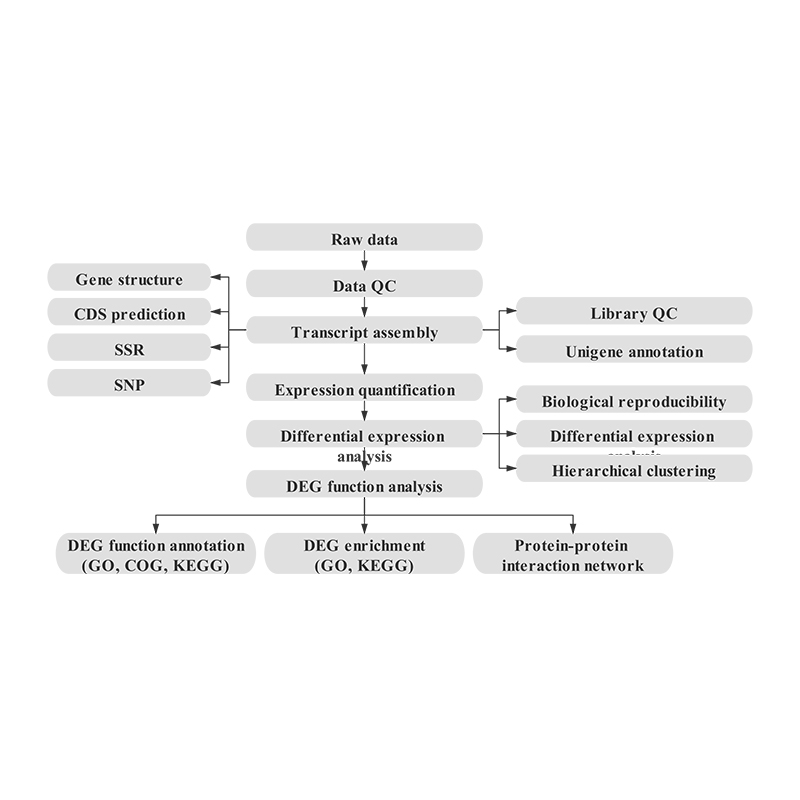
Secuenciación de ARNm-NGS sin base de referencia
Características
● Captura de poli ARNm antes de la preparación de la biblioteca.
● Independiente de cualquier genoma de referencia: basado en el ensamblaje de novo de transcripciones, generando una lista de unigenes que están anotados con múltiples bases de datos (NR, Swiss-Prot, COG, KOG, eggNOG, Pfam, GO, KEGG)
● Análisis bioinformático completo de la expresión genética y la estructura de la transcripción.
Ventajas del servicio
●Amplia experiencia: con un historial de procesamiento de más de 600.000 muestras en BMKGENE, que abarcan diversos tipos de muestras, como cultivos celulares, tejidos y fluidos corporales, nuestro equipo aporta una gran experiencia a cada proyecto. Hemos cerrado con éxito más de 100.000 proyectos de mRNA-Seq en diversos dominios de investigación.
●Control de calidad riguroso: implementamos puntos de control centrales en todas las etapas, desde la preparación de muestras y bibliotecas hasta la secuenciación y la bioinformática. Este seguimiento meticuloso garantiza la entrega de resultados consistentes de alta calidad.
● Anotación completa: utilizamos múltiples bases de datos para anotar funcionalmente los genes expresados diferencialmente (DEG) y realizar el análisis de enriquecimiento correspondiente, proporcionando información sobre los procesos celulares y moleculares subyacentes a la respuesta del transcriptoma.
●Soporte postventa: nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Requisitos de muestra y entrega
| Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| Poli A enriquecido | Iluminación PE150 DNBSEQ-T7 | 6-10 GB | Q30≥85% |
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 10 | ≥ 0,2 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Para plantas: RIN≥4,0; Para animales: RIN≥4,5; 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
● Plantas:
Raíz, Tallo o Pétalo: 450 mg
Hoja o Semilla: 300 mg
Fruta: 1,2 gramos
● Animales:
Corazón o Intestino: 300 mg
Vísceras o Cerebro: 240 mg
Músculo: 450 mg
Huesos, Cabello o Piel: 1g
● Artrópodos:
Insectos: 6g
Crustáceos: 300 mg
● Sangre entera: 1 tubo
● Celdas: 106 células
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3; B1, B2, B3.
Envío:
1. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
2. Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
Bioinformática
Montaje del transcriptoma y selección unigene.
Anotación Unigene
Correlación de muestras y evaluación de réplicas biológicas.
Genes expresados diferencialmente (DEG)
Anotación funcional de DEG
Enriquecimiento funcional de DEG
Explore los avances facilitados por los servicios de secuenciación de ARNm de NGS eucariotas de BMKGene a través de una colección seleccionada de publicaciones.
Shen, F. y col. (2020) 'Ensamblaje de transcriptoma de novo y expresión genética con sesgo sexual en las gónadas del bagre de Amur (Silurus asotus)', Genomics, 112 (3), págs. doi: 10.1016/J.YGENO.2020.01.026.
Zhang, C. y col. (2016) 'Análisis del transcriptoma del metabolismo de la sacarosa durante el hinchamiento y desarrollo del bulbo en cebolla (Allium cepa L.)', Frontiers in Plant Science, 7 (septiembre), p. 212763. doi: 10.3389/FPLS.2016.01425/BIBTEX.
Zhu, C. y col. (2017) 'Ensamblaje, caracterización y anotación de novo para el transcriptoma de Sarcocheilichthys sinensis', PLoS ONE, 12 (2). doi: 10.1371/JOURNAL.PONE.0171966.
Zou, L. y col. (2021) 'El análisis del transcriptoma de novo proporciona información sobre la tolerancia a la sal de Podocarpus macrophyllus bajo estrés salino', BMC Plant Biology, 21 (1), págs. doi: 10.1186/S12870-021-03274-1/FIGURAS/9.