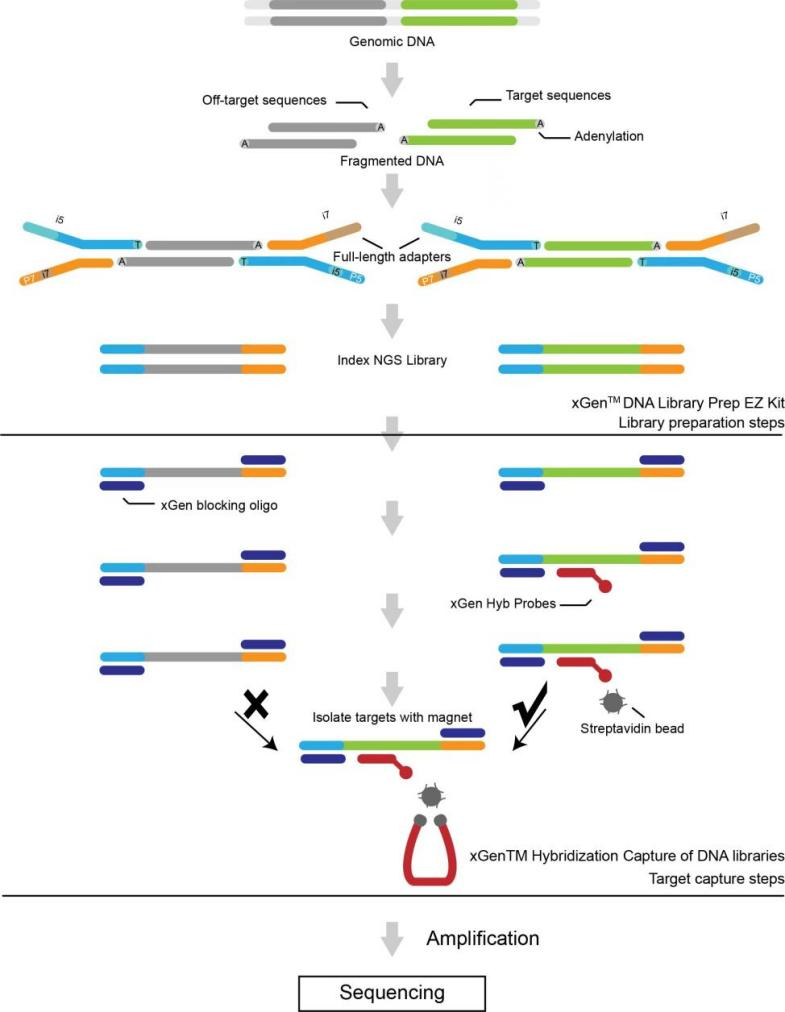
Secuenciación del exoma completo humano
Características del servicio
● Dos paneles de exoma disponibles basados en el enriquecimiento objetivo con sondas: Sure Select Human All Exon v6 (Agilent) y xGen Exome Hybridization Panel v2 (IDT).
● Secuenciación en Illumina NovaSeq.
● Pipeline bioinformático dirigido al análisis de enfermedades o análisis de tumores.
Ventajas del servicio
●Se dirige a la región codificante de proteínas: Al capturar y secuenciar regiones codificantes de proteínas, hWES se utiliza para revelar variantes relacionadas con la estructura de las proteínas.
●Rentable:hWES produce aproximadamente el 85% de las mutaciones asociadas a enfermedades humanas del 1% del genoma humano.
●Alta precisión: Con una alta profundidad de secuenciación, hWES facilita la detección tanto de variantes comunes como de variantes raras con frecuencias inferiores al 1%.
●Control de calidad riguroso: Implementamos cinco puntos de control centrales en todas las etapas, desde la preparación de muestras y bibliotecas hasta la secuenciación y la bioinformática. Este seguimiento meticuloso garantiza la entrega de resultados consistentes de alta calidad.
●Análisis Bioinformático Integral: nuestra cartera va más allá de identificar variaciones en el genoma de referencia, ya que incorpora análisis avanzados diseñados para abordar específicamente preguntas de investigación relacionadas con aspectos genéticos de enfermedades o análisis de tumores.
●Soporte Postventa:Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Especificaciones de muestra
| Estrategia de captura de exones | Estrategia de secuenciación | Salida de datos recomendada |
| Sure Select Human All Exon v6 (Agilent) o Panel de hibridación de exoma xGen v2 (IDT)
| Illumina NovaSeq PE150 | 5-10 GB Para trastornos mendelianos/enfermedades raras: > 50x Para muestras de tumores: ≥ 100x |
Requisitos de muestra
| Tipo de muestra
| Cantidad(Qubit®)
| Concentración | Volumen
| Pureza (NanoDrop™) |
|
ADN genómico
| ≥ 50ng | ≥ 6 ng/μL | ≥ 15 µL | DO260/280=1,8-2,0 sin degradación, sin contaminación
|
Bioinformática

El análisis bioinformático de muestras de enfermedades hWES incluye:
● Control de calidad de datos de secuenciación
● Alineación del genoma de referencia
● Identificación de SNP e InDels
● Anotación funcional de SNP e InDels

El análisis bioinformático de muestras tumorales incluye:
● Control de calidad de datos de secuenciación
● Alineación del genoma de referencia
● Identificación de SNPs, InDels y variaciones somáticas
● Identificación de variantes de la línea germinal
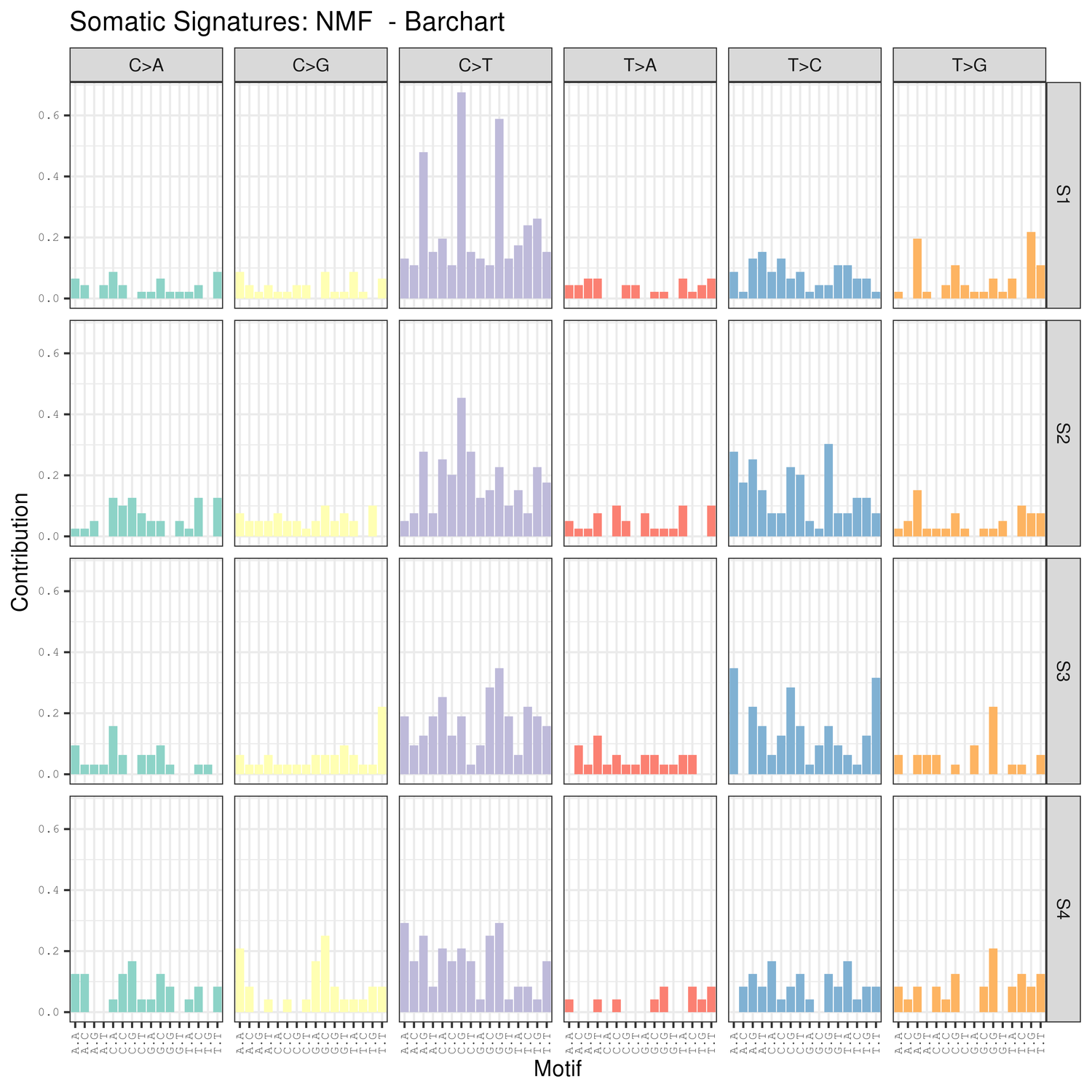
● Análisis de firmas de mutaciones
● Identificación de genes impulsores basados en mutaciones de ganancia de función.
● Anotación de mutación al nivel de susceptibilidad a los medicamentos.
● Análisis de heterogeneidad: cálculo de pureza y ploidía.
Flujo de trabajo del servicio

Entrega de muestra

extracción de ADN

construcción de biblioteca

Secuenciación

Análisis de datos

Entrega de datos

Servicios postventa
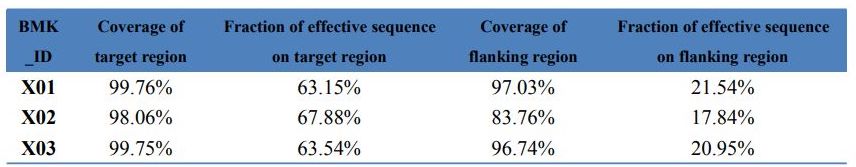
Control de calidad de datos: estadísticas de captura del exoma
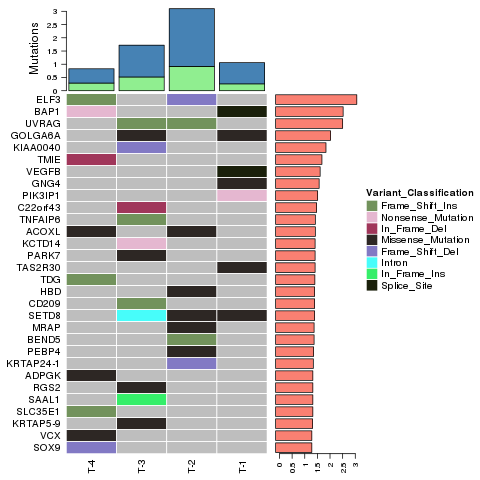
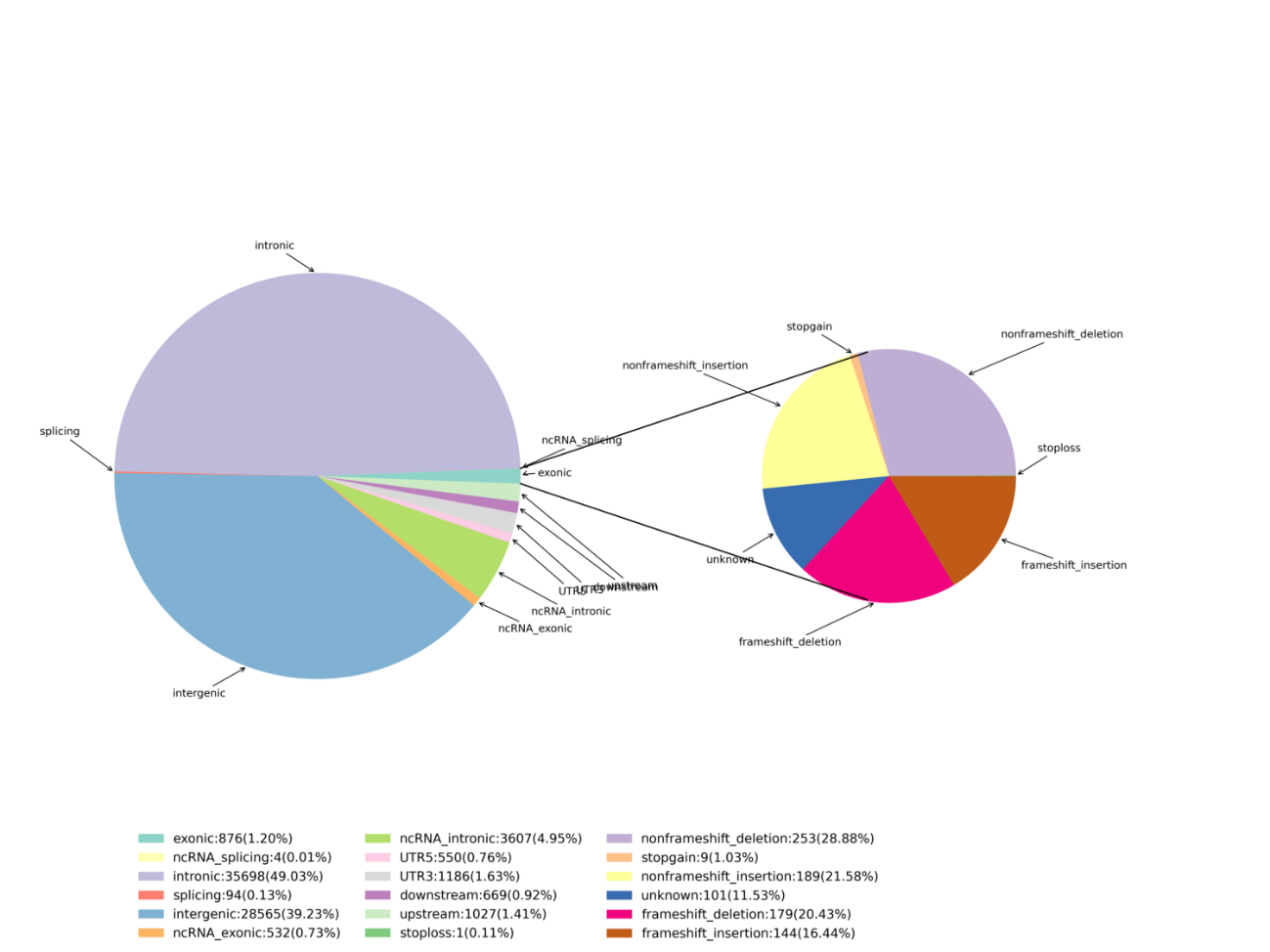
Identificación de variantes – InDels
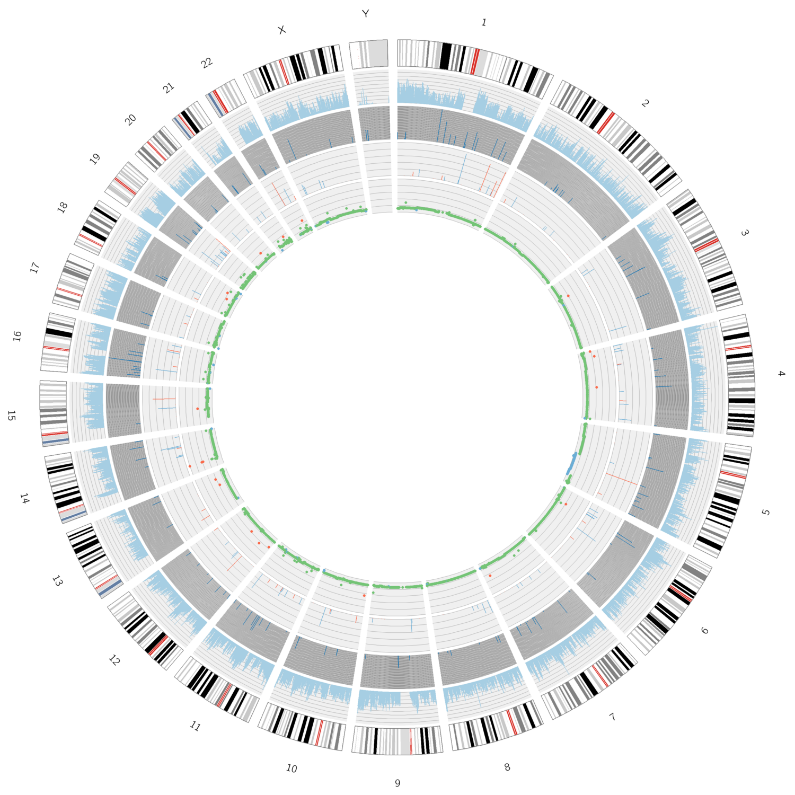
Análisis avanzado: identificación y distribución de SNPs/InDels nocivos – Parcela Circos
Análisis de tumores: identificación y distribución de mutaciones somáticas – Parcela Circos
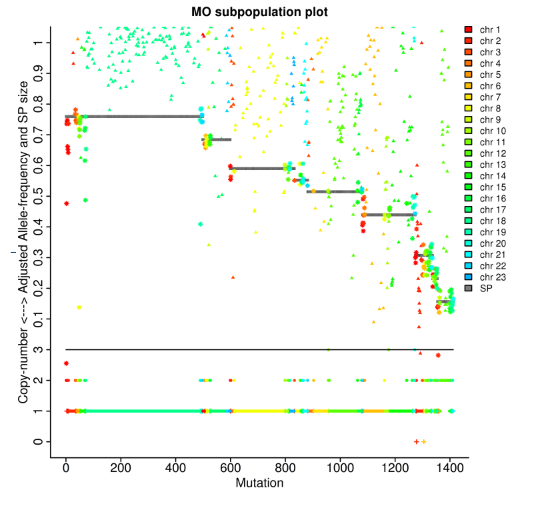
Análisis de tumores: linajes clonales.