Ensamblaje del genoma basado en HI-C
Características de servicio
● Secuenciación en Illumina Novaseq con PE150.
● El servicio requiere muestras de tejido, en lugar de ácidos nucleicos extraídos, para retirarse con formaldehído y conservar las interacciones de ADN-proteína.
● El experimento HI-C implica la restricción y la reparación final de los extremos adhesivos con biotina, seguido de la circularización de los extremos contundentes resultantes mientras preservan las interacciones. El ADN se tira hacia abajo con cuentas de estreptavidina y se purifica para la preparación posterior de la biblioteca.
Ventajas de servicio

Descripción general de Hi-C
(Lieberman-Aiden E et al.,Ciencia, 2009)
●Eliminando la necesidad de datos de población genética:Hi-C sustituye la información esencial requerida para el anclaje contig.
●Densidad de alta marcador:conduciendo a una alta relación contig de anclaje por encima del 90%.
●Experiencia extensa y registros de publicación:BMKGene tiene una amplia experiencia con más de 2000 casos de ensamblaje del genoma HI-C de 1000 especies diferentes y varias patentes. Más de 200 casos publicados tienen un factor de impacto acumulativo de más de 2000.
●Equipo bioinformático altamente calificado:Con patentes internas y copyrights de software para experimentos HI-C y análisis de datos, el software de datos de visualización autodesarrollado permite que el bloqueo manual se mueva, la inversión, la revocación y la rehacer.
●Soporte posterior a las ventas:Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio posterior a la venta de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia de problemas de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
●Anotación integral: Utilizamos múltiples bases de datos para anotar funcionalmente los genes con variaciones identificadas y realizar el análisis de enriquecimiento correspondiente, proporcionando información sobre múltiples proyectos de investigación.
Especificaciones de servicio
| Preparación de la biblioteca | Estrategia de secuenciación | Salida de datos recomendado | Control de calidad |
| Biblioteca Hi-C | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Requisitos de muestra
| Tejido | Monto requerido |
| Víspera de animales | ≥ 2 g |
| Músculo animal | |
| Sangre de mamíferos | ≥ 2 ml |
| Aves de corral/sangre de pez | |
| Planta-hoja fresca | ≥ 3 g |
| Células cultivadas | ≥ 1x107 |
| Insecto | ≥ 2 g |
Flujo de trabajo de servicio

Diseño de experimentos

Entrega de muestra

Construcción de la biblioteca

Secuenciación

Análisis de datos

Servicios posteriores
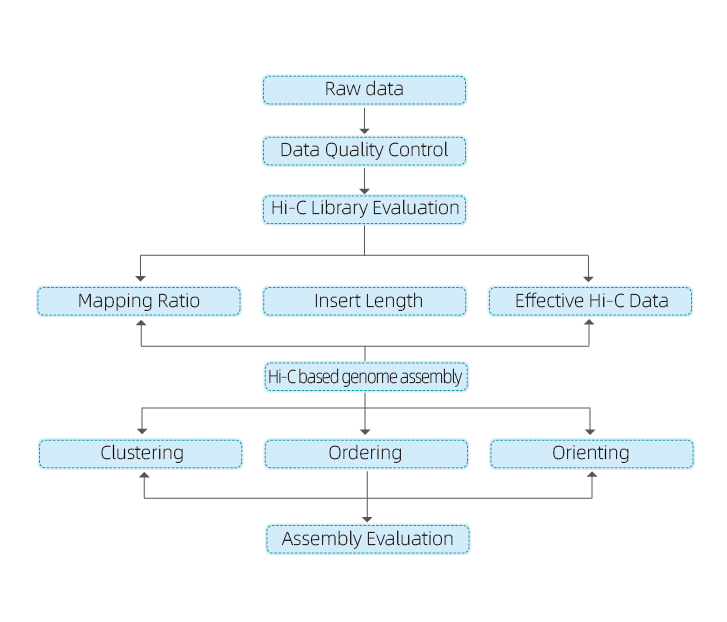
1) QC de datos sin procesar
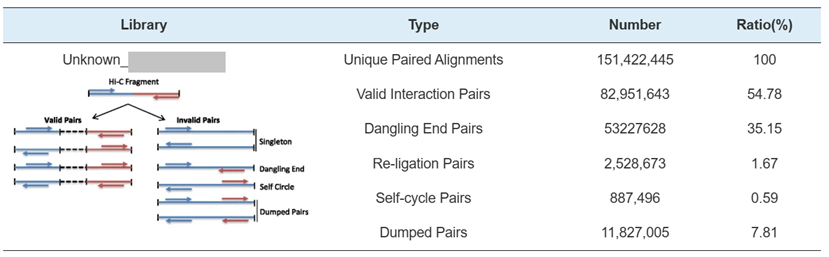
2) Biblioteca Hi-C QC: Estimación de interacciones HI-C válidas
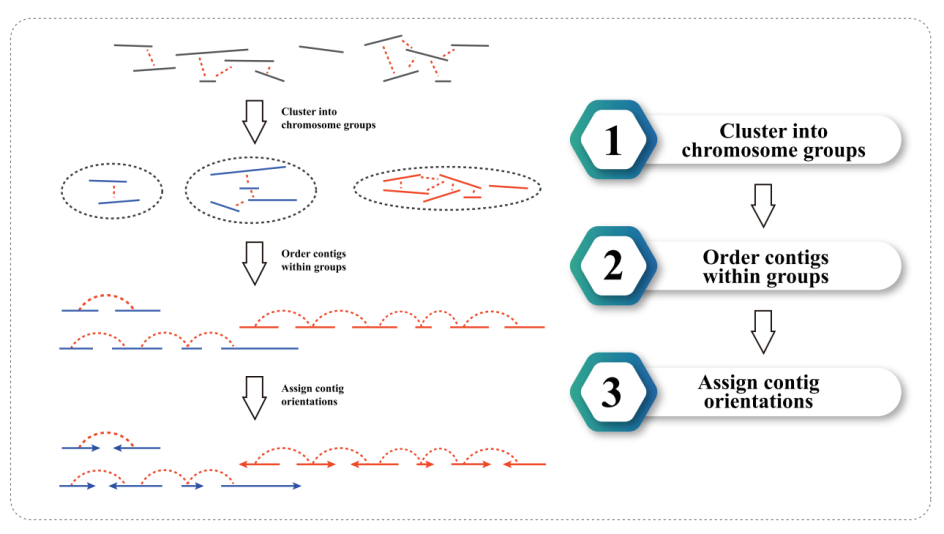
3) Asamblea HI-C: agrupación de contigs en grupos, seguido de orden de contig en cada grupo y asignando orientación contig
4) Evaluación de HI-C
Hi-C Library QC-Estimación de pares de interacción válidos de HI-C
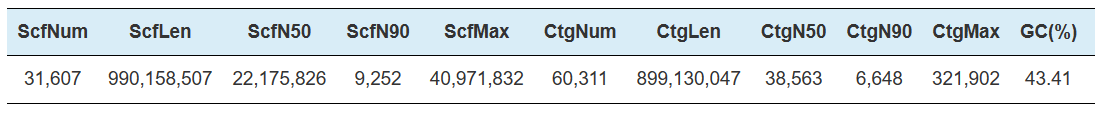
Asamblea HI-C-Estadísticas
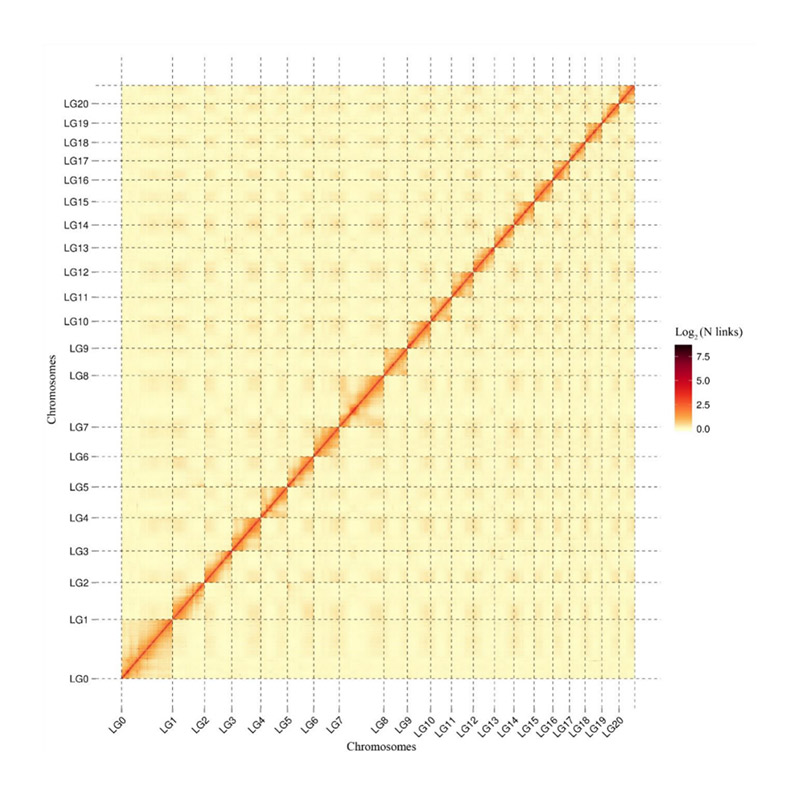
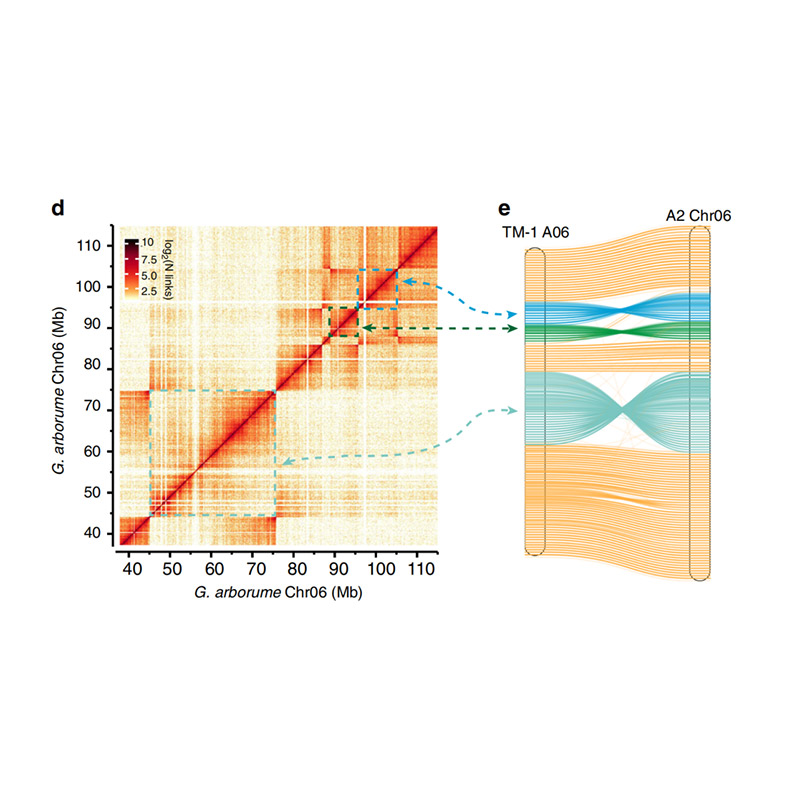
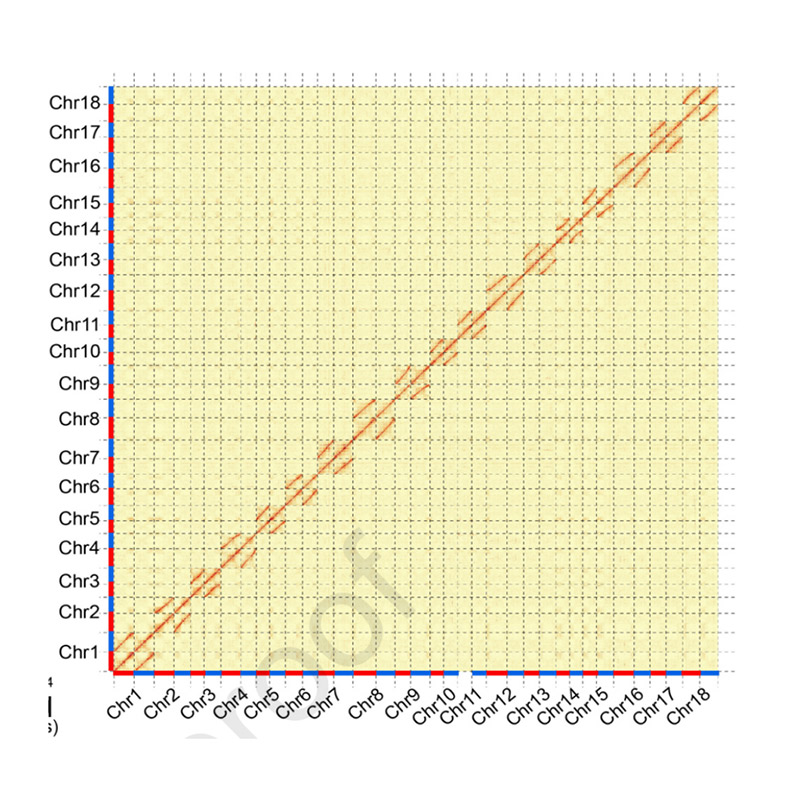
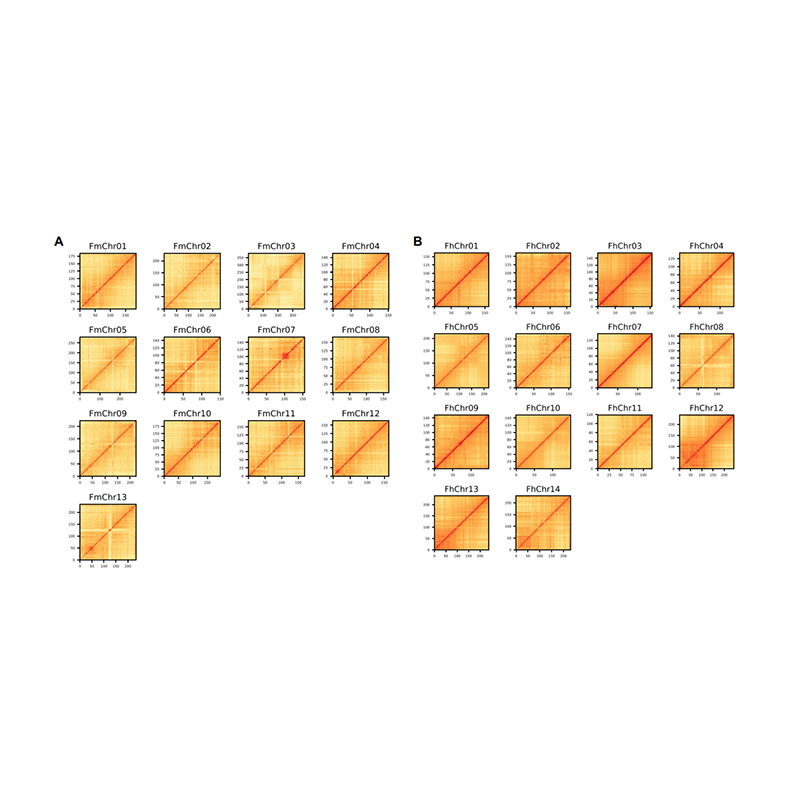
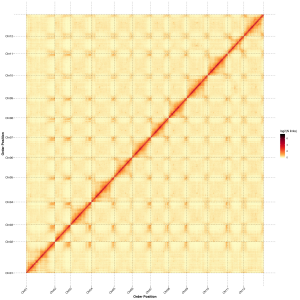
Evaluación posterior al ensamblaje: mapa de calor de la intensidad de la señal entre los contenedores
Explore los avances facilitados por los servicios de ensamblaje Hi-C de BMKGene a través de una colección curada de publicaciones.
Tian, T. et al. (2023) 'Asamblea del genoma y disección genética de un prominente germoplasma de maíz resistente a la sequía', Nature Genetics 2023 55: 3, 55 (3), pp. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'Un ensamblaje a escala cromosómica del genoma asiático de la abeja de abejas de abeja cerana', Frontiers in Genetics, 11, p. 524140. Doi: 10.3389/fgene.2020.00279/bibtex.
Zhang, F. et al. (2023) 'revelando la evolución de la biosíntesis de alcaloides tropanos analizando dos genomas en la familia Solanaceae', Nature Communications 2023 14: 1, 14 (1), pp. 1-18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Los genomas del árbol de banyan y las avisas polinizador proporcionan información sobre la coevolución de pasas de higos', Cell, 183 (4), pp. 875-889.e17. doi: 10.1016/j.cell.2020.09.043