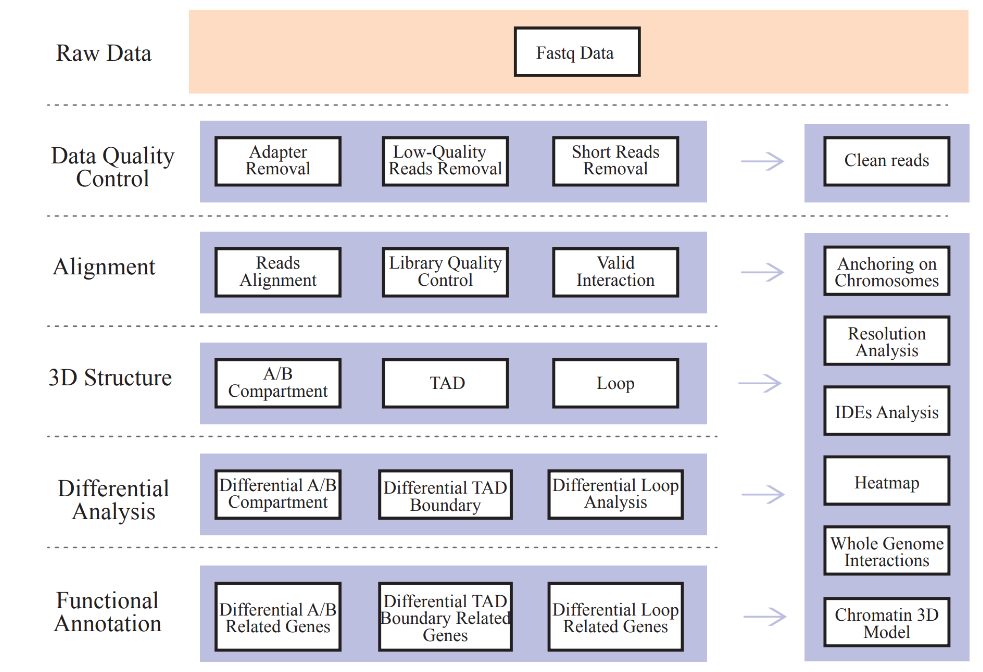
Interacción de cromatina basada en Hi-C
Características del servicio
● Secuenciación en Illumina NovaSeq con PE150.
● El servicio requiere que muestras de tejido, en lugar de ácidos nucleicos extraídos, se entrecrucen con formaldehído y conserven las interacciones ADN-proteína.
● El experimento Hi-C implica la restricción y reparación de los extremos pegajosos con biotina, seguida de la circularización de los extremos romos resultantes preservando al mismo tiempo las interacciones. Luego, el ADN se extrae con perlas de estreptavidina y se purifica para la posterior preparación de la biblioteca.
Ventajas del servicio
●Diseño óptimo de enzimas de restricción: para garantizar una alta eficiencia de Hi-C en diferentes especies con hasta un 93% de pares de interacción válidos.
●Amplia experiencia y registros de publicaciones:BMKGene tiene una amplia experiencia con >2000 proyectos de secuenciación Hi-C de 800 especies diferentes y varias patentes. Más de 100 casos publicados con un factor de impacto acumulado superior a 900.
●Equipo de Bioinformática altamente calificado:con patentes internas y derechos de autor de software para experimentos Hi-C y análisis de datos y un software de visualización de datos de desarrollo propio.
●Soporte Postventa:Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
●Anotación completa: utilizamos múltiples bases de datos para anotar funcionalmente los genes con variaciones identificadas y realizar el análisis de enriquecimiento correspondiente, brindando información sobre múltiples proyectos de investigación.
Especificaciones de servicio
| Biblioteca | Estrategia de secuenciación | Salida de datos recomendada | Resolución de señal Hi-C |
| Biblioteca Hi-C | Iluminación PE150 | Bucle de cromatina: 150x TAD: 50x | Bucle de cromatina: 10Kb TAD: 40Kb |
Requisitos de servicio
| tipo de muestra | Cantidad requerida |
| Tejido animal | ≥2g |
| sangre entera | ≥2ml |
| Hongos | ≥1g |
| Planta-tejido joven | 1g/alícuota, se recomiendan 2-4 alícuotas |
| Células cultivadas | ≥1x107 |
Incluye el siguiente análisis:
● Control de calidad de datos sin procesar;
● Control de calidad de la biblioteca Hi-C y mapeo: pares de interacción válidos y exponentes de decaimiento de interacción (IDE);
● Perfiles de interacción de todo el genoma: análisis cis/trans y mapa de interacción Hi-C;
● Análisis de la distribución de los compartimentos A/B;
● Identificación de TAD y bucles de cromatina;
● Análisis diferencial de elementos de estructura de cromatina 3D entre muestras y anotación funcional correspondiente de genes asociados.
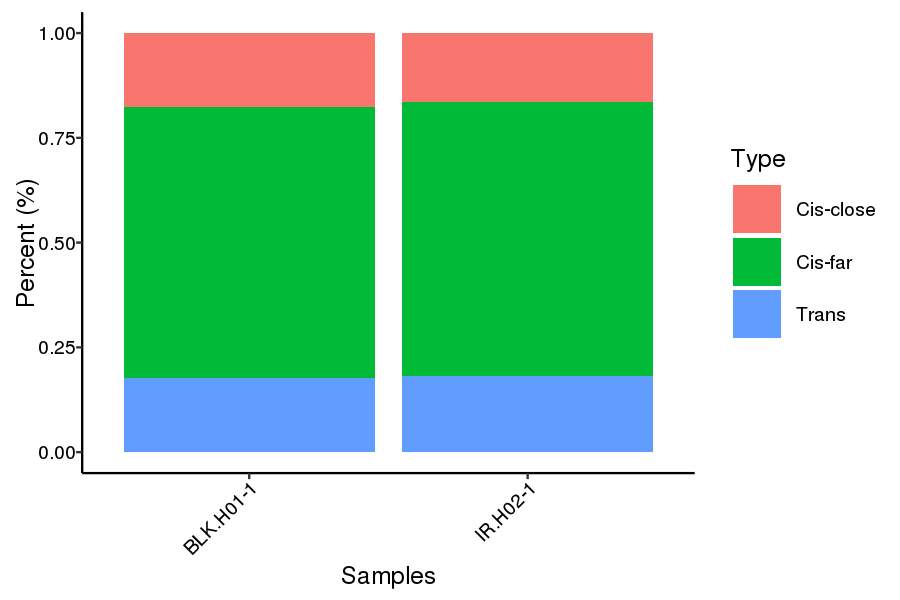
Distribución de proporciones cis y trans
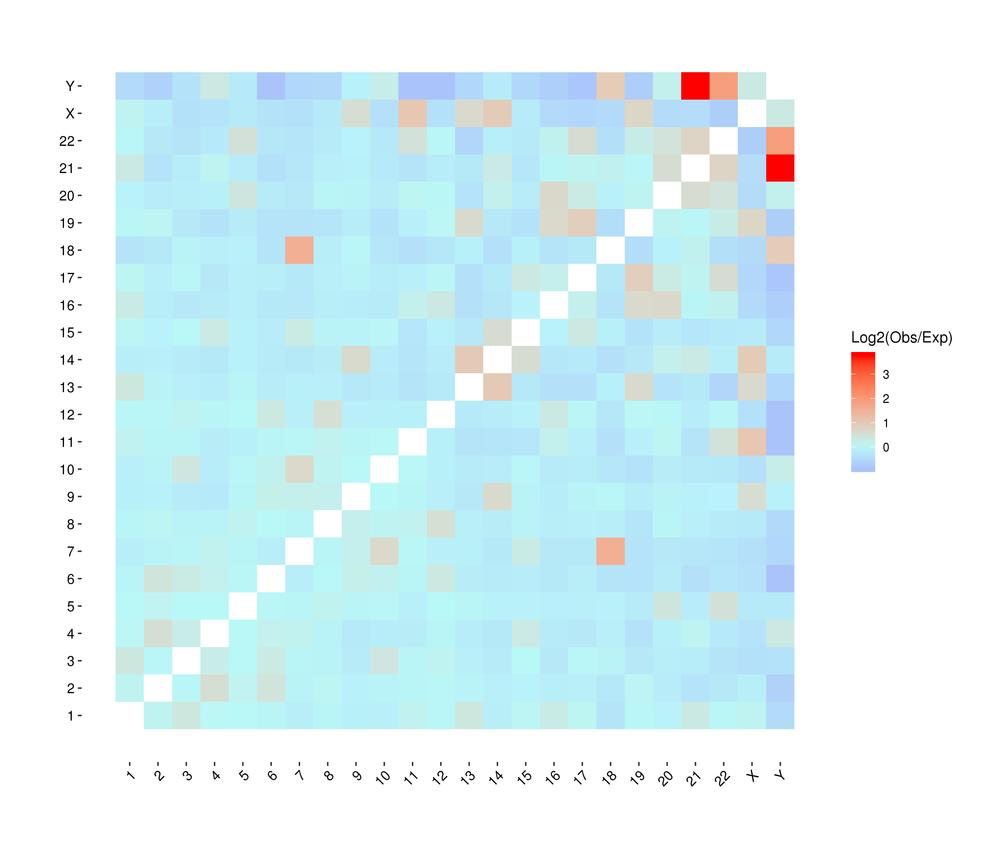
Mapa de calor de interacciones cromosómicas entre muestras.
Distribución de los compartimentos A/B en todo el genoma
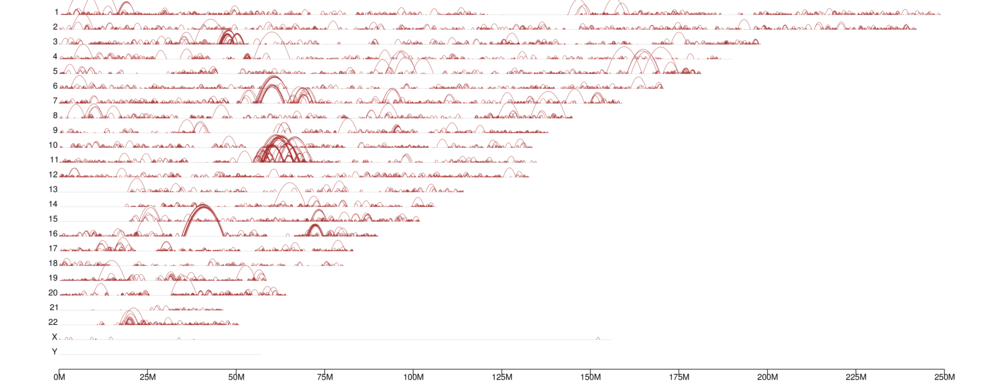
Distribución de bucles de cromatina en todo el genoma.
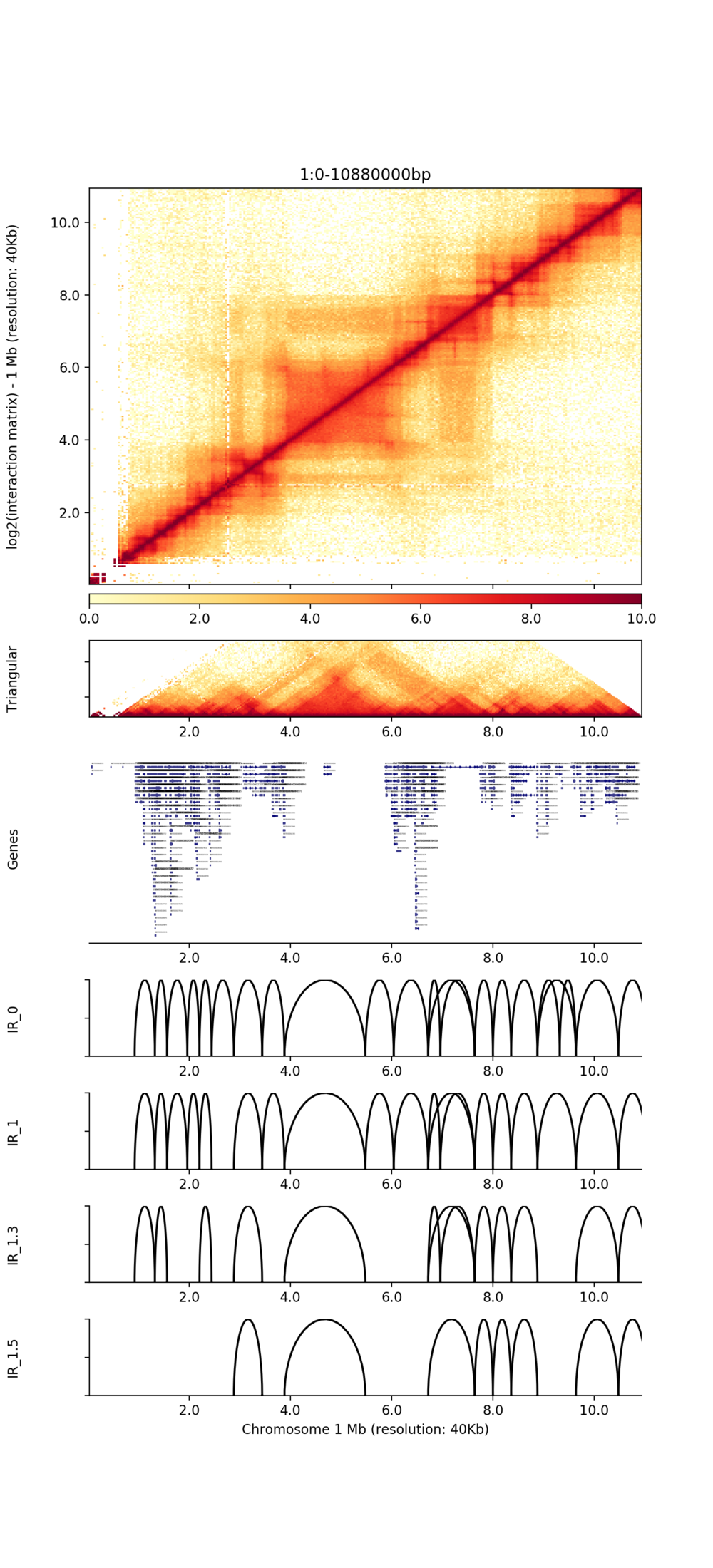
Visualización de TAD
Explore los avances en la investigación facilitados por los servicios de secuenciación Hi-C de BMKGene a través de una colección seleccionada de publicaciones.
Meng, T. y col. (2021) 'Un análisis multiómico integrado comparativo identifica a CA2 como un nuevo objetivo para el cordoma',Neurooncología, 23 (10), págs. 1709-1722. doi: 10.1093/NEUONC/NOAB156.
Xu, L. y col. (2021) 'La desorganización 3D y el reordenamiento del genoma brindan información sobre la patogénesis de NAFLD mediante la secuenciación integrada de Hi-C, Nanopore y ARN',Acta Pharmaceutica Sínica B, 11 (10), págs. 3150–3164. doi: 10.1016/J.APSB.2021.03.022.