Análisis de asociación de todo el genoma
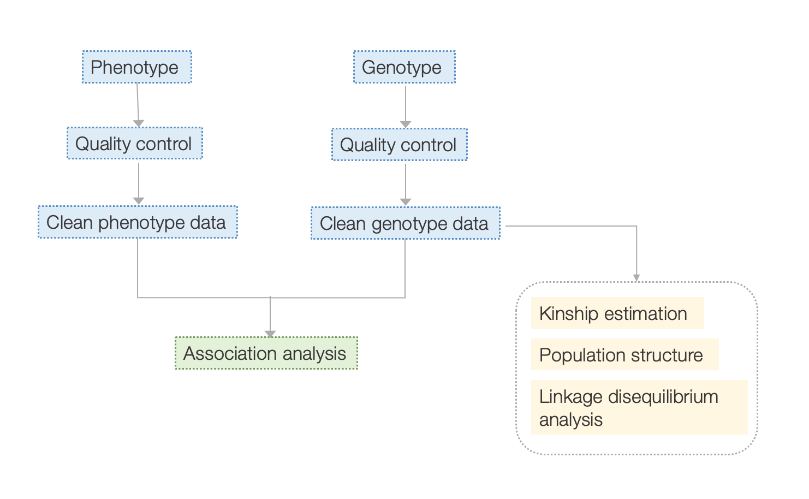
Flujo de trabajo

Ventajas del servicio
●Amplia experiencia y registros de publicaciones.: con experiencia acumulada en GWAS, BMKGene ha completado cientos de proyectos de especies en la investigación de poblaciones GWAS, ayudó a los investigadores a publicar más de 100 artículos y el factor de impacto acumulado alcanzó 500.
● Análisis bioinformático integral: el flujo de trabajo incluye análisis de asociación de rasgos SNP, entregando un conjunto de genes candidatos y su correspondiente anotación funcional.
●Equipo bioinformático altamente cualificado y ciclo de análisis corto.: con una gran experiencia en análisis genómicos avanzados, el equipo de BMKGene ofrece análisis completos con un tiempo de respuesta rápido.
●Soporte Postventa:Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses. Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Especificaciones y requisitos del servicio.
| Tipo de secuenciación | Escala de población recomendada | Estrategia de secuenciación | Requisitos de nucleótidos |
| Secuenciación del genoma completo | 200 muestras | 10x | Concentración: ≥ 1 ng/ µL Cantidad total ≥ 30 ng Degradación o contaminación limitada o nula |
| Fragmento amplificado de locus específico (SLAF) | Profundidad de la etiqueta: 10x Número de etiquetas: < 400 Mb: se recomienda WGS < 1 Gb: 100 000 etiquetas 1GB > 2Gb: 300K etiquetas Máximo 500 000 etiquetas | Concentración ≥ 5 ng/μL Cantidad total ≥ 80 ng Nanogota OD260/280=1,6-2,5 Gel de agarosa: degradación o contaminación nula o limitada
|
Selección de materiales



Diferentes variedades, subespecies, variedades locales/bancos de germoplasma/familias mixtas/recursos silvestres
Diferentes variedades, subespecies, variedades locales.
Familia de medios hermanos/familia de hermanos completos/recursos silvestres
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de datos

Servicios postventa
Incluye el siguiente análisis:
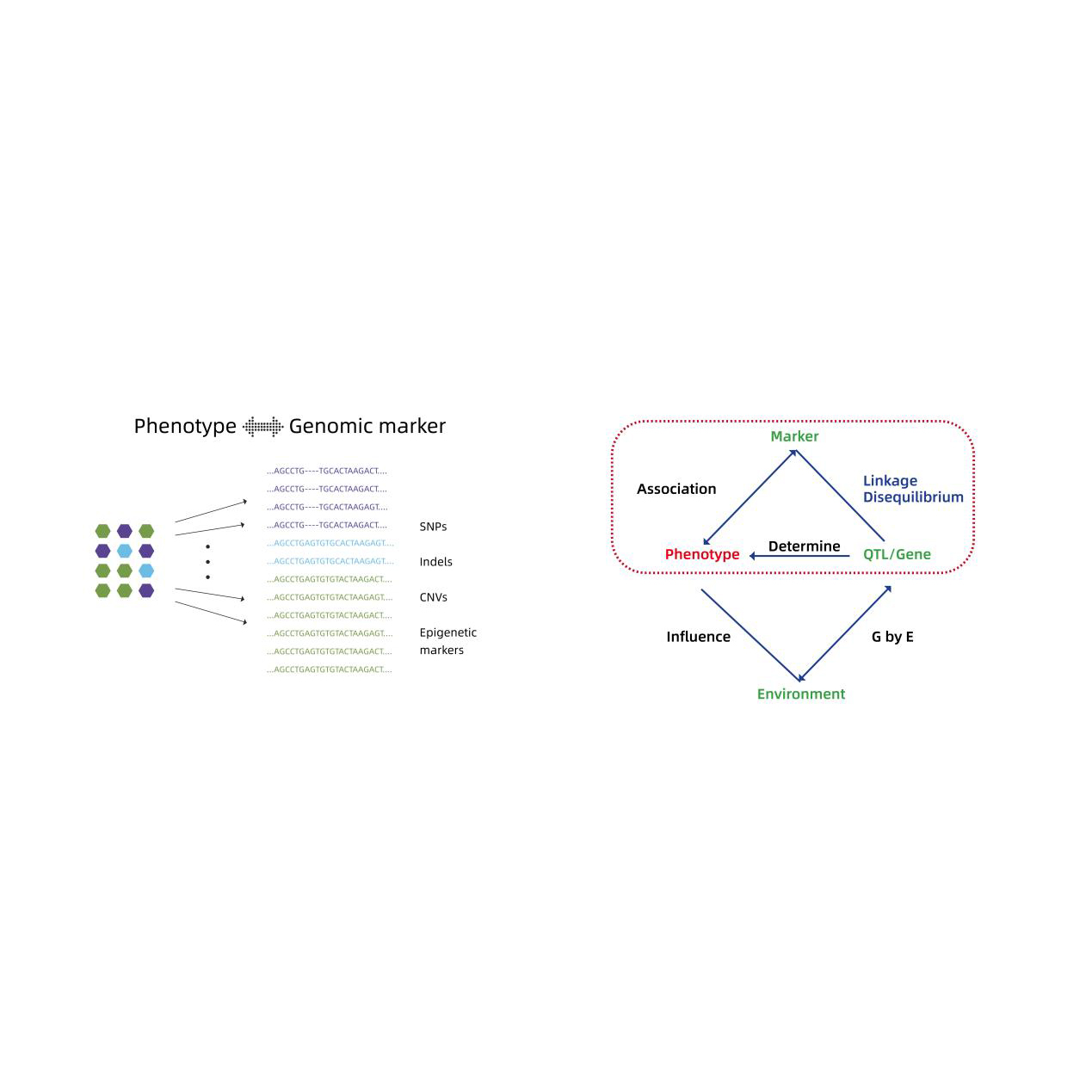
- Análisis de asociación de todo el genoma: modelo LM, LMM, EMMAX, FASTLMM
- Anotación funcional de genes candidatos.
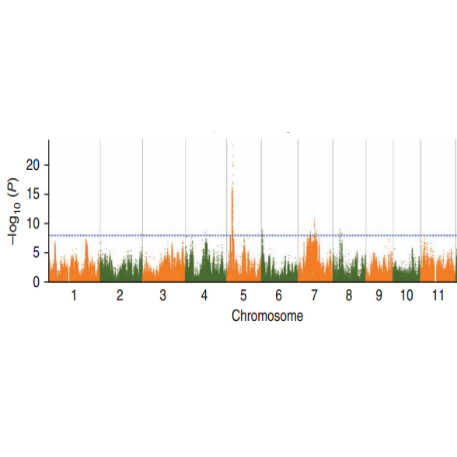
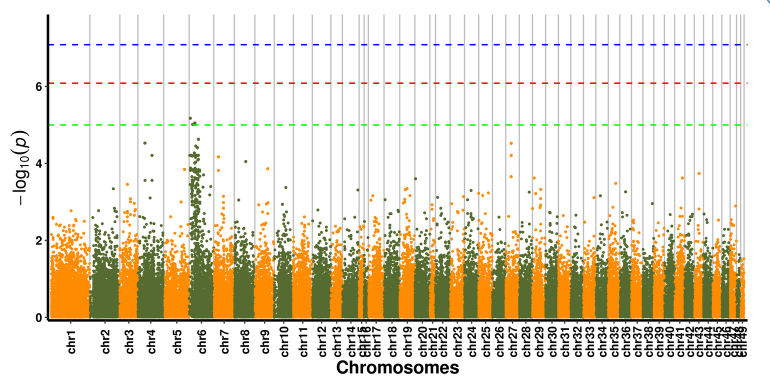
Análisis de asociación de rasgos SNP: diagrama de Manhattan
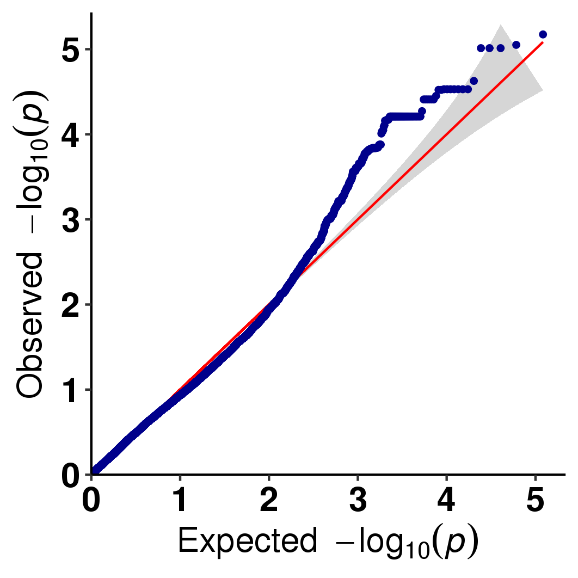
Análisis de asociación de rasgos SNP: gráfico QQ
Explore los avances facilitados por los servicios de GWAS de BMKGene a través de una colección seleccionada de publicaciones:
Lv, L. y col. (2023) 'Conocimiento de la base genética de la tolerancia al amoníaco en la navaja Sinonovacula constricta mediante un estudio de asociación de todo el genoma',Acuicultura, 569, pág. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. y col. (2022) 'Los análisis multiómicos de 398 muestras de mijo cola de zorra revelan regiones genómicas asociadas con la domesticación, rasgos de metabolitos y efectos antiinflamatorios',Planta molecular, 15 (8), págs. 1367-1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. y col. (2022) 'Mapeo de asociación de todo el genoma de fenotipos apenas sin casco en entornos de sequía',Fronteras en la ciencia vegetal, 13, pág. 924892. doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) 'GmST1, que codifica una sulfotransferasa, confiere resistencia a las cepas G2 y G3 del virus del mosaico de la soja',Planta, célula y medio ambiente, 44 (8), págs. 2777–2792. doi: 10.1111/PCE.14066.